摘 要:果胶酶在苎麻酶法脱胶过程中发挥重要作用,但现有果胶酶存在脱胶效率低、脱胶时间长等问题。为了挖掘新型、高效的果胶酶应用于苎麻脱胶工艺,本研究对地衣芽孢杆菌ATCC 14580 的编码序列(coding sequence, CDS)和蛋白序列进行碳水化合物酶注释,发掘了一个尚未验证功能的果胶裂解酶 BlicPL1。构建BlicPL1基因的大肠杆菌BL21表达载体,并进行了诱导表达和分离纯化。本研究首次通过实验证实了BlicPL1的果胶裂解酶活性,其比酶活为3952.54 U/mg,高于多数已报道的多糖裂解酶1(polysaccharide lyase 1, PL1)家族果胶裂解酶。酶学性质研究显示果胶裂解酶BlicPL1最适反应pH为10.0,最适反应温度为60°C;且在反应温度50?80°C和pH10.0?12.0范围内都表现出较高的酶活性,相对酶活力保持在80%以上。果胶裂解酶BlicPL1耐高温、耐碱特性与苎麻脱胶工艺中的高温、高 pH 环境具有很好的适配性。与常规果胶裂解酶依赖Ca2+激活不同,Ca2+对果胶裂解酶BlicPL1的活性不仅无明显促进作用,甚至有一定抑制作用,凸显了其在Ca2+响应机制方面的独特性,表明果胶裂解酶BlicPL1是一种新型果胶裂解酶。进一步将果胶裂解酶BlicPL1用于苎麻脱胶,苎麻失重率为16.17%,苎麻脱胶率达到44.09%;扫描电子显微镜结果显示果胶裂解酶BlicPL1作用后有助于去除苎麻表面残留的中间层状组织并减少游离胶状物质附着,使苎麻表面变得更光滑、纤维变得更加分散;X 射线衍射结果显示果胶裂解酶BlicPL1脱胶后苎麻纤维特征纤维素峰位不变,结晶度从74.59%升至79.69%,表明纤维素占比增加,非纤维素物质被去除。本研究发掘的果胶裂解酶BlicPL1具有活性高,适用于中高温、碱性环境等特点,在苎麻脱胶工艺中具有较大应用潜力,为加速推进果胶酶运用于工业苎麻脱胶工艺奠定了基础。
关键词:果胶裂解酶;基因发掘;异源表达;酶学性质;苎麻脱胶
苎麻具有韧性高、透气性好、吸湿性佳和抗菌能力强等优异性能,是纺织工业中最具价值的材料之一,被公认为“天然纤维女王”[1-2]。苎麻由纤维素及多种胶质构成,包含果胶、纤维素、半纤维素、木质素、脂蜡质等成分,其中胶质占比达 25%?35%[3]。这些胶质成分在纤维中相互交织,限制了纤维的分离和分散,严重影响了苎麻纤维的可纺性和纺织性能,使其难以满足现代纺织工艺对纤维品质的要求[4-5]。因此,脱胶工艺不仅是苎麻加工流程中不可或缺的关键步骤,更是决定苎麻纤维能否实现高附加值应用的核心环节[6-7]。传统的化学脱胶法利用热碱对苎麻纤维进行脱胶处理[8-9],其操作简单且脱胶效果好,但能量投入高,可能会破坏苎麻纤维的结构形态,且产生的废水处理难度大,污染环境[10]。利用生物酶进行苎麻脱胶,苎麻纤维损伤少、能耗低、环境污染小且经济效益高[11],其中果胶酶在苎麻生物脱胶过程中发挥关键作用[12-13]。
果胶酶是一类能降解果胶物质的酶,根据作用方式的不同,果胶酶可分为 3类:原果胶酶、果胶酯酶和果胶解聚酶(包括果胶水解酶和果胶裂解酶)[14]。其中果胶裂解酶随机切割聚半乳糖醛酸的α-1,4-半乳糖醛糖键,在新形成的寡半乳糖醛酸的非还原端生成一个不饱和键;根据氨基酸序列的差异,果胶裂解酶被分为多糖裂解酶(polysaccharide lyase, PL) 1、2、3、9、10等不同家族[15]。果胶酶应用于苎麻脱胶具有低污染的优点,并且可以保留苎麻纤维的固有形态,提高苎麻纤维的质量[16-17]。但是现有果胶酶活性低、对工业环境耐受性差,使得酶法脱胶的成本高、脱胶效率低和脱胶时间长,阻碍了生物酶法在苎麻脱胶工业上的应用[15],因此挖掘新型、耐受性强、高效的果胶酶并应用于苎麻脱胶工艺是十分必要的。
果胶酶广泛存在于细菌、真菌等微生物中,研究表明枯草芽孢杆菌、地衣芽孢杆菌可以产生适用于苎麻脱胶的碱性果胶酶[12-15]。基于此,本研究利用碳水化合物活性酶(Carbohydrate-Active enZYmes, CAZY)数据库对地衣芽孢杆菌模式菌株——地衣芽孢杆菌ATCC 14580进行功能注释,挖掘得到一个尚未验证功能的PL1家族果胶裂解酶BlicPL1,并基于基因克隆技术进行异源表达,最后将表达纯化后的果胶裂解酶应用到苎麻脱胶工艺中,旨在获得能应用苎麻脱胶工艺中的高效新酶源,为解决苎麻加工过程中的苎麻脱胶问题奠定基础。
1材料与方法
1.1菌株、质粒、试剂
大肠杆菌 BL21 (DE3)感受态细胞购自南京诺唯赞生物科技股份有限公司;质粒pET28a和质粒pET23a-sfgfp-sumo为本实验室保存;多聚半乳糖醛酸购自上海源叶生物科技有限公司;本研究其他所用化学试剂均购自国药集团化学试剂有限公司;引物、基因合成及基因测序均由上海生工生物工程股份有限公司完成。
分别配制0.1mol/L醋酸溶液和醋酸钠溶液,通过使用精密pH计测量,逐步滴加混合这2种溶液,使缓冲液的pH值为6.0,用于后续酶学性质检测;分别配制0.1mol/L磷酸氢二钠溶液和磷酸二氢钠溶液,通过使用精密pH计测量,将2种溶液相互滴加混合,使缓冲液的pH值分别达到7.0或8.0,用于后续酶学性质检测;分别配制0.1 mol/甘氨酸溶液和氢氧化钠溶液,通过使用精密pH计测量,将2种溶液相互滴加混合,使缓冲液的pH值分别达到9.0、10.0、11.0、12.0,用于后续酶学性质检测。
1.2果胶酶基因的挖掘、同源建模和分子对接
根据地衣芽孢杆菌ATCC 14580的CDS序列及蛋白质序列,使用CAZY数据库(https://www.cazy.org/)注释其碳水化合物酶,发掘了一个果胶裂解酶 BlicPL1(GenBank 登录号:AAU25568.1)。以果胶裂解酶BlicPL1的序列为模板,在NCBI数据库用 Protein BLAST 检索;将果胶裂解酶BlicPL1与10个PL1家族的果胶裂解酶进行序列比对,以Clustal Omega进行蛋白质序列的多序列比对;通过SWISS-MODEL软件(https://swissmodel.expasy.org/interactive)基于克氏芽孢杆菌碱性果胶裂解酶的晶体结构(PDB 数据库登录号:7xks.1)预测果胶裂解酶 BlicPL1 的三维结构。从PubChem数据库(https://pubchem.ncbi.nlm.nih.gov)下载多聚半乳糖醛酸的三维结构。使用AutoDock Vina 1.2.3对果胶裂解酶BliPL1和底物多聚半乳糖醛酸进行半柔性对接[18],Grid box中心(x, y, z)=(2.234,?30.289,?9.364)Å,尺寸为47.25 Å×39.0 Å×47.25 Å,所有其他对接参数均设置在相应的默认值水平。利用PyMOL 2.6可视化展示果胶裂解酶BliPL1 和底物多聚半乳糖醛酸的相互作用。
1.3果胶裂解酶基因的表达载体构建及蛋白表达纯化
合成果胶裂解酶基因BlicPL1,设计反向扩增引物pET28a-f、pET28a-r,以质粒pET28a为模板通过聚合酶链式反应(polymerase chain reaction, PCR)扩增获得线性化的pET28a,将BlicPL1基因片段与pET28a线性化载体片段通过同源重组的方式连接,构建重组质粒pET28a-BlicPL1,转化到大肠杆菌BL21中,将转化子涂布到含有卡那霉素(100 mg/L)抗性的LB平板上,获得阳性克隆子。为了进一步获得可溶性异源表达蛋白,设计引物sfgfp-sumo-f、sfgfp-sumo-r,以pET23a-sfgfp-sumo质粒为模板通过PCR扩增获得基因sfgfp-sumo。设计反向引物pET28a-BlicPL1-f、pET28a-BlicPL1-r,以质粒pET28a-BlicPL1 为模板通过PCR扩展获得线性化的质粒pET28a-BlicPL1,将线性化的质粒pET28a-BlicPL与sfgfp-sumo基因片段通过同源重组的方式构建重组质粒pET28a-sfgfp、sumo-BlicPL1,转化至大肠杆菌BL21中,获得阳性克隆子。构建重组质粒所用的引物序列如表1所示。将阳性克隆子单菌落接入含有卡那霉素(100 mg/L)抗性 LB液体培养基中,待菌株生长至OD600为0.6时,加入IPTG使其终浓度为0.15 mmol/L,进行诱导表达,16°C、150r/min 诱导表达20h,离心收集菌体,破菌之后上清即为粗酶液。粗酶液流经镍柱材料后,依次使用20mmol/L 和50mmol/L的咪唑缓冲液洗脱柱内的杂蛋白,洗脱过程中持续观察紫外检测器的吸光度数值,直至吸光度不再发生变化,然后使用300mmol/L 咪唑缓冲液进行特异性洗脱,收集目标蛋白。纯化后的目的蛋白通过SDS-PAGE电泳检测蛋白纯度。
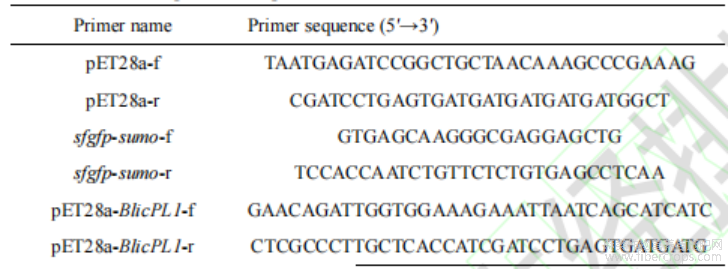
表1 表达载体构建所用 PCR 扩增引物
1.4果胶裂解酶酶活力的测定
取900μL浓度为0.2%的多聚半乳糖醛酸溶液与100μL的酶液混合,混匀后在50°C、pH10.0的反应条件下反应10min,反应结束后12000r/min 离心1 min 取反应上清液,取300µL反应液上清与600µL的3,5二硝基水杨酸(3,5-dinitrosalicylic acid, DNS)充分混合,放入100°C金属浴显色10min后测量 OD540值,根据上清中D-半乳糖醛酸的浓度计算酶活。以牛血清白蛋白为标准蛋白,使用Braford方法测定果胶裂解酶的浓度。果胶裂解酶酶活力的定义为在一定条件下,每min分解多聚半乳糖醛酸产生1µg半乳糖醛酸所需的酶量。
1.5酶学性质的研究
最适反应温度:分别在20、30、40、50、60、70、80°C 的反应温度下将果胶裂解酶与0.2%的多聚半乳糖醛酸溶液(pH10.0)反应10 min,测量不同反应温度下的酶活,将最高酶活力定义为100%,计算其他温度下的相对酶活力。
最适反应 pH:分别用pH6.0、7.0、8.0、9.0、10.0、11.0、12.0 的缓冲溶液配制0.2%的多聚半乳糖醛酸溶液,取果胶裂解酶与不同pH的多聚半乳糖醛酸溶液在60°C 下反应10min,测量不同pH条件下的酶活,将最高酶活力定义为 100%,计算其他pH条件下的相对酶活力。
温度稳定性:分别在25、40、60°C条件下将果胶裂解酶保存在pH10.0的缓冲溶液中4h,每隔1h取样测酶活,将未孵育时(0h)的酶活定义为100%,计算在不同温度孵育不同时间后果胶裂解酶的相对剩余活性。
pH稳定性:将果胶裂解酶分别在pH7.0、8.0、9.0、10.0缓冲溶液中4°C 孵育12 h,每隔3h取一次样,将未孵育时(0h)的酶活定义为100%,计算在不同pH孵育不同时间后果胶裂解酶的相对剩余活性。
金属离子对酶活性的影响:选择 11 种金属离子,分别为 Zn2+、Ca2+、Cu2+、Mg2+、Al3+、Na+、Ba2+、K+、Ni2+、Fe3+、Fe2+;在60°C、pH10.0的反应条件下,分别测定终浓度1mmol/L和10mmol/L的金属离子对酶催化活性的影响,将没有添加任何金属离子时的酶活力定义为100%,计算添加不同金属离子后的果胶裂解酶的相对酶活。酶分子动力学参数的研究:用pH10.0缓冲溶液配制不同浓度的多聚半乳糖醛酸底物,在60°C条件下与果胶裂解酶反应10min,得到不同浓度底物浓度下的果胶裂解酶反应速率,采用Lineweaver-Burk双倒数作图法,得出米氏常数(Km)和最大反应速度(Vmax),并计算催化常数(Kcat)和专一性常数(Kcat/Km)。
1.6苎麻脱胶实验流程
称取1.0g购买自四川省达州市的纯手工剥皮苎麻于100 mL锥形瓶中,加入30mL浓度为0.5 mol/L的NaOH溶液,沸水浴加热处理60min;倒掉旧溶液,添加等浓度新液,再沸水浴120 min。处理后样品经去离子水冲洗至中性,50°C 烘干至恒重,每组实验设置4个重复。通过测定碱处理前后样品干重差值,计算脱胶前苎麻的果胶含量。称取2.0g的纯手工剥皮苎麻,放置于100mL锥形瓶中,加入果胶裂解酶溶液,并加入pH10.0的缓冲液定容至50mL,使果胶裂解酶酶活的终浓度为10U/mL,将不加入果胶裂解酶的处理组作为对照。将锥形瓶瓶口密封放入恒温摇床中 60°C、150r/min反应4 h,反应结束后用去离子水将苎麻表面的胶体物质清洗干净直至水不再浑浊。将清洗后的苎麻放入105°C 干燥箱将苎麻烘干至恒重,用于后续检测。
1.7苎麻脱胶性能测定
使用分析天平记录干燥后的苎麻样品重量,测定苎麻纤维失重率及脱胶率来衡量苎麻脱胶效果。苎麻纤维失重率(%)=[(脱胶前苎麻重量?脱胶后苎麻重量)/脱胶前苎麻重量]×100;苎麻脱胶率(%)=[(脱胶前苎麻果胶含量?脱胶后苎麻果胶含量)/脱胶前苎麻果胶含量]×100。对照组及实验组苎麻处理结束后,随机挑选干燥状态的苎麻纤维,利用导电胶将它们均匀且平整地粘贴到铜板上,确保样品都彼此独立、不相互重叠,对粘贴好的样品进行喷金处理后放置在具备真空环境的扫描电子显微镜设备中检测不同处理的苎麻纤维的微观表面形态。将干燥后的苎麻纤维样品用剪刀剪碎,使用X射线衍射仪测定不同处理组的苎麻纤维晶态结构特征。X射线管工作电压40kV,工作电流30mA,扫描角度范围设定为5°?50°,扫描速度为10(°)/min。纤维素的相对结晶度(%)=[晶体部分的衍射积分强度/(晶体部分的衍射积分强度+非晶部分的衍射积分强度)]×100。
1.8 数据分析
利用SPSS22.0软件对实验数据进行统计分析,每个实验至少重复3次,使用独立样本t检验检测实验组和对照组之间的差异显著性,*** 表示 P<0.001。使用Origin 8.0 软件进行作图。
2结果与分析
2.1果胶酶的挖掘与结构预测
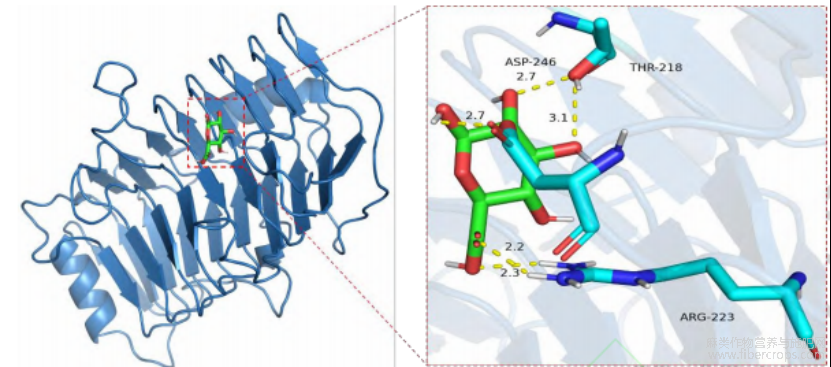
根据CAZY的注释结果,挖掘到一个来源地衣芽孢杆菌的果胶裂解酶BlicPL1,其基因全长1026bp,编码341个氨基酸,蛋白分子量为37.4kDa,其功能尚未验证。如图1所示,通过多序列比对分析发现,果胶裂解酶BlicPL1与已报道的PL1家族果胶裂解酶具有较高的同源性,其保守序列(图1中白色字体、红色背景的氨基酸序列)也基本吻合。进一步运用同源建模法对果胶裂解酶BlicPL1的三级结构进行预测,结果如图2所示,果胶裂解酶BlicPL1具有果胶裂解酶 PL1家族成员的特征性右手β-螺旋折叠,并显示出与果胶裂解酶PL1家族成员的整体结构相似性,表明BlicPL1属于PL1家族的果胶裂解酶。分子对接结果表明,果胶裂解酶BlicPL1第218位的苏氨酸、第223位的精氨酸、第246的天门冬氨酸与多聚半乳糖醛酸形成氢键相互作用力;其第170位的亮氨酸,第195位和199位的丝氨酸,第196位、201位和245位的精氨酸,第217位的谷氨酸与多聚半乳糖醛酸形成范德华相互作用力,表明这些位点可能参与了果胶裂解酶BlicPL1与底物多聚半乳糖醛酸的结合。后续可以通过对这些关键位点进行改造和理性设计来进一步提高果胶裂解酶BlicPL1的酶活性和稳定性,以提升其在苎麻脱胶工业中的应用潜力。

图 1地衣芽孢杆菌来源的果胶裂解酶 BlicPL1 与 PL1 家族果胶裂解酶的多序列比对结果
注:图中编号为果胶裂解酶 NCBI 数据库登录号;白色字体、红色背景的氨基酸位点:保守序列。
图 2果胶裂解酶BlicPL1与底物多聚半乳糖醛酸的分子对接结果
2.2果胶裂解酶基因的克隆表达及纯化
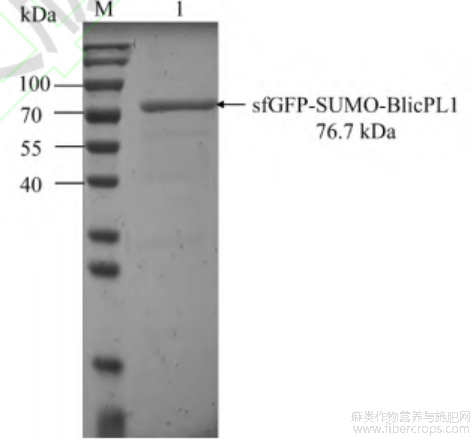
成功构建了果胶裂解酶BlicPL1原核表达菌株大肠杆菌BL21 pET28a-BlicPL1后进行诱导表达,发现目的蛋白大量形成包涵体。在表达载体中加入促溶标签sfgfp-sumo构建原核表达菌株大肠杆菌BL21 pET28a-sfgfp-sumo-BlicPL1,诱导表达后果胶裂解酶BlicPL1成功可溶性表达。纯化结果如图3所示,sfGFP-SUMO-BlicPL1蛋白纯化条带大小为76.7kDa,且纯化后条带单一、纯度较高可用于后续酶学性质研究和工业应用。
图 3果胶裂解酶 BlicPL1 分离纯化后 SDS-PAGE 电泳结果
2.3最适反应温度和温度稳定性分析
如图4A所示,果胶裂解酶BlicPL1的最适反应温度为60°C,且在50?80°C的反应温度范围内相对酶活保持在90%以上。果胶裂解酶BlicPL1温度稳定性如图4B所示,分别在25、40 和60°C 的环境条件下孵育1h后,剩余酶活力均超过60%,其中在25°C条件下的稳定性最好,孵育1h后剩余酶活超过 0%。

图4化肥减量配施有机肥与密度互作下胡麻和土壤指标间的相关性果胶裂解酶 BlicPL1 的最适反应温度、pH 及温度、pH 稳定性
注:A:最适反应温度;B:温度稳定性;C:最适反应 pH;D:pH 稳定性。
2.4 最适反应 pH 及 pH 稳定性分析
如图4C所示,果胶裂解酶BlicPL1的最适反应pH为10.0,且在pH10.0?12.0碱性 条件下果胶裂解酶BlicPL1酶活力缓慢降低,相对酶活保持在80%以上。基于果胶裂解酶BlicPL1的碱性催化特性,进一步研究了该酶在pH 7.0?10.0 范围内的稳定性。结果 如图4D所示,果胶裂解酶BlicPL1在pH 8.0?10.0的碱性环境中均表现出较好的稳定 性,孵育12h后,其剩余酶活力均能超过90%,且在碱性条件(pH8.0?10.0)中的稳定性 优于中性环境(pH7.0)。果胶裂解酶BlicPL1在碱性条件下的耐受能力较强、反应速度最 快,属于碱性果胶裂解酶。
2.5 金属离子对果胶裂解酶 BlicPL1 活性的影响
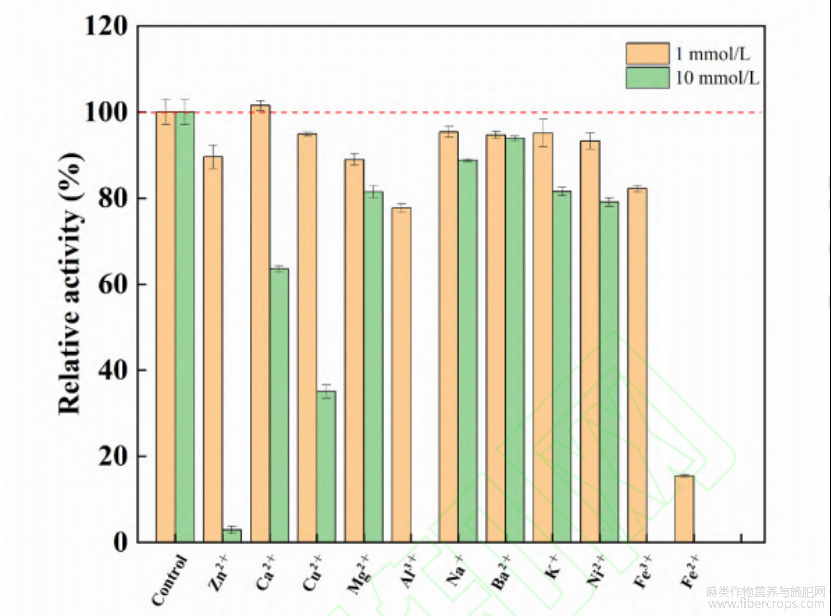
不同金属离子对果胶裂解酶BlicPL1活性的影响结果如图5所示,除1mmol/L 的 Ca2+对果胶裂解酶BlicPL1活性有轻微的促进作用外,其他金属离子对果胶裂解酶BlicPL1活性都有不同程度的抑制作用,其中1mmol/ 的 Fe2+对果胶裂解酶BlicPL1具 有明显的抑制作用,其剩余酶活不足20%。10mmol/L的金属离子对果胶裂解酶BlicPL1活性都有一定的抑制作用,其中 Zn²?、Al³?、Fe³?和Fe²?对果胶裂解酶BlicPL1表现出强烈抑制作用,其剩余酶活不足5%甚至完全失活。常规果胶裂解酶通常依赖 Ca2+激活, 其酶活通常可以被不同浓度的 Ca2+显著激活达到原酶活的数倍[12,15],最高可激活至原酶活的23倍[15]。本研究发现1mmol/L的Ca2+对果胶裂解酶BlicPL1活性仅有1.6%的轻微促进作用,10mmol/L的Ca2+对果胶裂解酶BlicPL1的活性抑制水平达36.4%,说明果胶裂解酶BlicPL1的酶活不依赖Ca2+激活,其作用机制不同于常规果胶裂解酶的Ca2+结合模式,凸显了其在Ca2+响应机制上的独特性,表明果胶裂解酶BlicPL1是一种新型果胶裂解酶,其具体作用机制有待进一步深入研究。
图 5 金属离子对果胶裂解酶 BlicPL1 酶活性的影响
2.6 果胶裂解酶动力学参数
基于果胶裂解酶BlicPL1的最适反应温度(60°C)及最适反应pH(10.0),在最适条件 下测得果胶裂解酶BlicPL1的比酶活为3952.54U/mg。通过 Lineweaver-Burk双倒数作图法,得到果胶裂解酶BlicPL1的米氏常数(Km)为11.27mg/mL,最大反应速率(Vmax)为0.13mg/(mL·min),催化常数(Kcat)为11.79min-1,专一性常数(Kcat/Km)为1.04 mL/(mg·min)。
2.7 果胶裂解酶 BlicPL1 在苎麻脱胶应用中的效果
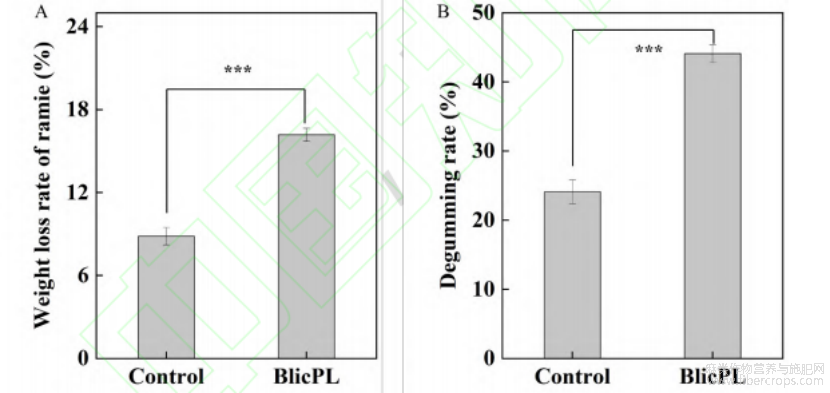
经检测本研究所用的手工剥皮苎麻果胶含量为36.67%,苎麻脱胶效果如图 6所示,碱性环境(pH10.0)也有一定的脱胶效果,因此对照组苎麻也有一定的失重率和脱胶率;但果胶裂解酶BlicPL1处理后的苎麻失重率和脱胶率分别为16.17%和44.09%,显著高于对照组。不同处理组的苎麻外观形态如图7所示,对照组苎麻颜色较深且硬度较高,果胶裂解酶BlicPL1处理后的苎麻纤维颜色浅且具有较好的柔韧性和纤维分散性。进一步利用扫描电子显微镜观察不同处理苎麻样品的纤维形态,如图7C所示,对照组的苎麻表面存在较多的非纤维素成分,呈现出粗糙的形态,由于苎麻纤维中间有果胶等黏性物质相互黏连,因此没有观察到单一的苎麻纤维;如图7D所示,经过果胶裂解酶BlicPL1处理后的苎麻纤维表面光滑。这些结果表明果胶裂解酶BlicPL1具有良好的苎麻脱胶效 果,果胶裂解酶BlicPL1作用苎麻之后有助于去除残留的中间层状组织,使苎麻表面变 得更光滑且可以减少游离胶状物质附着,因此果胶裂解酶BlicPL1处理之后苎麻纤维变得更加分散。
图6果胶裂解酶 BlicPL1 的苎麻脱胶效果
注:A:苎麻失重率;B:苎麻脱胶率。*** 表示 P<0.001。

图7果胶裂解酶 BlicPL1 处理后的苎麻表观形态和扫描电镜结构
注:A:缓冲液处理后苎麻表观形态;B:果胶裂解酶 BlicPL1 处理后的苎麻表观形态;C:
缓冲液处理后苎麻扫描电镜结构;D:果胶裂解酶 BlicPL1 处理后的苎麻扫描电子显微镜结构。标尺为 10 μm。
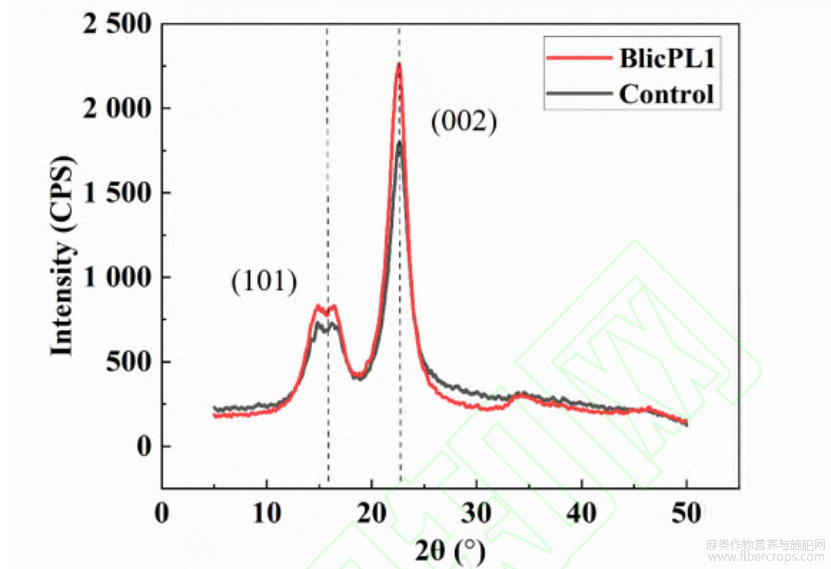
苎麻脱胶后的X射线衍射结果如图8所示,22°?23的2θ范围的主衍射带表征纤维 素Ⅰ(图8中002特征峰)的晶体群;2θ处于14.8°?16.8之间的衍射带代表纤维素Ⅱ(图8中101特征峰)的晶体群。对照组和果胶裂解酶BlicPL1处理后的苎麻纤维的特征纤维素峰位无明显差异,且曲线变化趋势相近,表明经果胶裂解酶BlicPL1脱胶处理后苎麻纤维的结晶形态没有变化。对照组苎麻纤维的结晶度为74.59%,果胶裂解酶BlicPL1处理后 的苎麻纤维结晶度提升至79.69%。结晶度的上升表明纤维素的含量增高,说明苎麻中的非纤维素物质已被去除,表明果胶裂解酶BlicPL1仅仅破坏了含有杂胶体的苎麻纤维的无定形区域。
图8果胶裂解酶 BlicPL1 处理或缓冲液处理后的苎麻纤维 X 射线衍射分析结果
注:CPS 表示每秒计数。
3讨论与结论
苎麻脱胶工艺是决定苎麻纤维高值化应用的重要环节[6-7],利用热碱对苎麻纤维进行脱胶处理的传统化学脱胶法会产生大量工业废水、污染环境[10]。果胶酶应用于苎麻脱胶工艺,不仅可以减少能源消耗和环境污染,还可以保留苎麻纤维的固有形态,提高苎麻纤维的质量[19-20],为苎麻脱胶的绿色生产工艺提供了可行方案。但是,现有果胶酶在实际生产中存在一些缺陷:首先,现有果胶酶的活性低,导致脱胶效率低、成本高[15,21];其次,苎麻脱胶工艺的生产环境主要为高温、碱性环境,果胶酶在高温及强碱条件下的稳定性较差,容易受到外界环境的影响而失去活性[15]。这些因素限制了果胶酶在苎麻脱胶工艺中的应用,因此挖掘新型、高效、耐受性强的果胶酶并应用于苎麻脱胶工艺是十分必要的。本研究利用CAZY数据库从地衣芽孢杆菌中发掘了一个果胶裂解酶基因BlicPL1,并构建了其原核表达系统。果胶裂解酶BlicPL1与PL1家族果胶裂解酶的保守序列与功能域高度相似,具有PL1家族标志性的右手β-螺旋折叠结构,属于PL1家族果胶裂解酶。本研究首次通过实验证实了BlicPL1的果胶裂解酶活性,果胶裂解酶BlicPL1的比酶活为3952.54 U/mg,其活性高于多数已报道的PL1家族果胶裂解酶活性,如来源于克劳氏芽孢杆菌和地衣芽孢杆菌的PL1家族果胶裂解酶BliPelA和BacPelA的比酶活分别为320.0 U/mg 和675.5 U/mg[12,22]。果胶裂解酶BlicPL1表现出优异的耐高温特性,其最适温度为60°C,在50?80°C的高温范围内仍保持90%以上的相对酶活性。相较于常规果胶裂解酶,如韦勒森氏芽孢杆菌来源的果胶裂解酶BvPelB在环境温度超过50°C后酶活性急剧衰减[23],果胶裂解酶BlicPL1的高温耐受特性显著拓展了其在高温工业环境中的应用潜力。果胶裂解酶BlicPL1在碱性环境下具有较高的稳定性和催化活性,其在pH10.0?12.0 的强碱性环境中相对酶活保持在80%以上,在pH8.0?10.0的碱性环境下孵育12h后的剩余酶活维持在90%以上。这种特性使得其在纺织工业常用的pH9.0?11.0的强碱性脱胶环境下,既能维持高效催化活性,又可以抵抗长时间碱性暴露导致的不可逆失活,显著提升了其在连续化生产工艺中的适用性,在苎麻脱胶流程中,无需频繁调节反应体系pH值即可实现胶质成分的持续降解。苎麻脱胶实验结果显示,果胶裂解酶BlicPL1表现出较好的脱胶效率,能显著去除果胶,苎麻失重率达16.17%,其苎麻脱胶效果优于多数已报道果胶裂解酶,如不同来源的果胶裂解酶PEL168、PelN、BspPel、B-pN-pelA 分别处理苎麻后其失重率分别为15.0%、12.0%、9.2%、13.5%[24-27]。果胶裂解酶BlicPL1处理后使苎麻纤维表面光滑、分散性提升,且未破坏纤维素晶体结构(结晶度从 74.59%升至 79.69%),说明其特异性降解苎麻胶质的同时保留其纤维完整性。综上所述,果胶裂解酶BlicPL1在苎麻脱胶工艺中具有较大应用潜力,进一步推进了将果胶裂解酶运用到工业苎麻脱胶工艺,为工业上利用生物酶脱胶解决苎麻脱胶问题奠定了基础。
参考文献
[1]CHENG LF, DUAN SW, FENG XY, ZHENG K, YANG Q, XU H, LUO W, PENG YD. Ramiedegumming methodologies: a short review[J]. Journal of Engineered Fibers and Fabrics, 2020, 15: 558925020940105.
[2]DING JX, ZHOU C, DONG Z. Trend of ramie industry development: a review of green D egumming and the utilization of processing residues[J]. Journal of Cleaner Production, 2023, 384: 135487.
[3]LI PD, SHU T, LIU TT, YU TY, WANG HH, FU CH, YU LJ. Visually revealing the ramie bast structure and its obstructive effects on bio-degumming[J]. Industrial Crops and Products, 2024, 221: 119301.
[4]SADRMANESH V, CHEN Y. Bast fibres: structure, processing, properties, and applications[J]. International Materials Reviews, 2019, 64(7): 381-6.
[5]LI ZF, LI ZL, DING RY, YU CW. Composition of ramie hemicelluloses and effect of polysaccharides on fiber properties[J]. Textile Research Journal, 2016, 86(5): 451-460.
[6]WANG JC, LIU ZX, LI XZ, LIU GD, ZHAO J. Elucidating structure of pectin in ramie fiber to customize enzyme cocktail for high-efficiency enzymatic degumming[J]. Carbohydrate Polymers, 2023, 314: 120954.
[7]MAO KW, CHEN HG, QI HH, QIU ZD, ZHANG L, ZHOU JG. Visual degumming process of ramie fiber using a microbial consortium RAMCD407[J]. Cellulose, 2019, 26(5): 3513-3528.
[8]HUANG H, TANG Q, LIN GY, LIU Y, YU JY, DING B, LI ZL. Anthraquinone-assisted deep eutectic solvent degumming of ramie fibers: Evaluation of fiber properties and degumming performance[J]. Industrial Crops and Products, 2022, 185: 115115.
[9]TANG Q, CHEN YX, DU MJ, YU JY, LI ZL, DING B. Research progress in Ramie fiber extraction: Degumming method, working mechanism, and fiber performance[J]. Industrial Crops and Products, 2024, 222: 119876.
[10]CUI Y, JIA ML, LIU L, ZHANG RY, CHENG LD, YU JY. Research on the character and degumming process of different parts of ramie fiber[J]. Textile Research Journal, 2018, 88(17): 2013-2023.
[11]SHADHIN M, RAHMAN M, JAYARAMAN R, CHEN Y, MANN D, ZHONG W. Natural biomass & waste biomass fibers–Structures, environmental footprints, sustainability, degumming methods, & surface modifications[J]. Industrial Crops and Products, 2023, 204: 117252.
[12]ZHOU C, XUE YF, MA YH. Characterization and overproduction of a thermo-alkaline pectate lyase from alkaliphilic Bacillus licheniformis with potential in ramie degumming[J]. Process Biochemistry, 2017, 54: 49-58.
[13]YUAN Y, ZHANG XY, ZHAO Y, ZHANG H, ZHOU YF, GAO J. A novel PL9 pectate lyase from Paenibacillus polymyxa KF-1: cloning, expression, and its application in pectin degradation[J]. International Journal of Molecular Sciences, 2019, 20(12): 3060.
[14]HUGOUVIEUX-COTTE-PATTAT N, CONDEMINE G, SHEVCHIK VE. Bacterial pectate lyases, structural and functional diversity[J]. Environmental Microbiology Reports, 2014, 6(5): 427-440.
[15]LIU SJ, QIN Y, WANG QY, ZHANG J, ZHOU J, HE BX, LIANG XQ, XIAN L, WU JH. A novel pectate lyase with high specific activity from Bacillus sp. B58-2: Gene cloning, heterologous expression and use in ramie degumming[J]. Enzyme and Microbial Technology,2024, 175: 110395.
[16]JOHN J, SURENDRANATHAN KAIMAL KK, SMITH ML, RAHMAN PKSM,
CHELLAM PV. Advances in upstream and downstream strategies of pectinase bioprocessing: a review[J]. International Journal of Biological Macromolecules, 2020, 162: 1086-1099.
[17]AMIN F, BHATTI HN, BILAL M. Recent advances in the production strategies of microbial pectinases: a review[J]. International Journal of Biological Macromolecules, 2019, 122: 1017 -1026.
[18]YADAV K, PATEL K, VARGHESE M, MANI A, YADAV S, YADAV D. Molecular modelling and docking of cloned pectin lyases from Fusarium species[J]. Biologia, 2025, 80(6): 1495-1512.
[19]SUHENDAR D, DIMAS AZKA MAULANA M, SUTRISNO A, WALTAM DR, YOHAN ABIDIN K, NANDYAWATI D, NURHAYATI N, WIDYASTI E, WAHJONO E, ADIHAYYU MONCONEGORO D, HANIYYA H, SRIHERWANTO C, LAKSAMANA PUTRA N, RAHMANI N. Optimization of enzymatic ramie degumming using thermoalkaliphilic xylanase from Bacillus halodurans CM1 and a commercial pectinase[J]. Biocatalysis and Biotransformation, 2024, 42(6): 749-759.
[20]KAUR D, SHARMA V, JOSHI A, BATRA N, RAMNIWAS S, SHARMA AK. Pectinases as promising green biocatalysts having broad-spectrum applications: Recent trends, scope, and relevance[J]. Biotechnology and Applied Biochemistry, 2023, 70(5): 1663-1678.
[21]CHANDEL V, BISWAS D, ROY S, VAIDYA D, VERMA A, GUPTA A. Current advancements in pectin: extraction, properties and multifunctional applications[J]. Foods, 2022, 11(17): 2683.
[22]ZHOU C, XUE YF, MA YH. Cloning, evaluation, and high-level expression of a thermoalkaline pectate lyase from alkaliphilic Bacillus clausii with potential in ramie degumming[J]. Applied Microbiology and Biotechnology, 2017, 101(9): 3663-3676.
[23]PAVLOVI? M, ŠOKARDA SLAVI? M, KOJI? M, MARGETI? A, RISTOVI? M, DRULOVI? N, VUJ?I? Z. Unveiling novel insights into Bacillus elezensis 16B pectin lyase for improved fruit juice processing[J]. Food Chemistry, 2024, 456: 140030.
[24]ZHANG CJ, YAO J, ZHOU C, MAO LW, ZHANG GM, MA YH. The alkaline pectate lyase PEL168 of Bacillus subtilis heterologously expressed in Pichia pastoris is more stable and efficient for degumming ramie fiber[J]. BMC Biotechnology, 2013, 13: 26.
[25]LI XM, WANG HL, ZHOU C, MA YH, LI J, SONG JN. Cloning, expression and characterization of a pectate lyase from Paenibacillus sp. 602 in recombinant Escherichia coli[J]. BMC Biotechnology, 2014, 14: 18.
[26]ZHENG XY, ZHANG YM, LIU XX, LI C, LIN Y, LIANG SL. High-level expression and biochemical properties of a thermo-alkaline pectate lyase from Bacillus sp. RN1 in Pichia pastoris with potential in ramie degumming[J]. Frontiers in Bioengineering and Biotechnology, 2020, 8: 850.
[27]ZOU MY, LI XZ, SHI WJ, GUO FF, ZHAO J, QU YB. Improved production of alkaline polygalacturonate lyase by homologous overexpression pelA in Bacillus subtilis[J]. Process Biochemistry, 2013, 48(8): 1143-1150.
文章摘自:周雪婷,朱泂婷,李华南,等.新型果胶酶的发掘、性质表征及其在苎麻脱胶中的应用[J/OL].生物工程学报,1-21[2026-01-29].https://doi.org/10.13345/j.cjb.250756.