摘 要:为挖掘亚麻抗倒伏关键位点与基因,在5个环境下,对269份亚麻种质资源抗倒伏相关10个性状进行鉴定及其全基因组关联分析,结果表明亚麻抗倒伏性状存在广泛的表型变异,鲜重的变异系数最大(54.94%),株高变异系数最小(13.68%),亚麻抗倒伏10个性状的表型变异系数依次排序为鲜重>茎秆断裂力>茎秆抗折力>木质素>纤维素>穿刺强度>抗压强度>重心高度>茎粗>株高。5个环境下该群体木质素的广义遗传力最大,达到了65.72%,重心高度的广义遗传力最小,为46.12%,广义遗传力依次排序为木质素>纤维素>鲜重>茎秆断裂力>抗压强度>穿刺强度>茎秆抗折力>茎粗>株高>重心高度;亚麻抗倒伏性状的全基因组关联分析获得了显著关联的SNP位点61个,表型解释率为6.45%~12.35%。在显著关联的SNP位点(-log10≥5.1)上下游10kb范围内注释到72个候选基因,其中2个环境下共同检测到的基因4个,分别为8号染色体上SNP(group8:26914041)位点下游1815bp处检测到的细胞壁合成候选基因LUSG00009364,表型解释率为10.89%,3号染色体上SNP(group3:153196)位点上游6843bp处检测到的DHHC棕榈酰转移酶基因LUSG00009997,表型解释率为12.35%,11号染色体上SNP(group11:23793243)位点上游6043bp处检测到的Ppx/GppA磷酸酶基因LUSG00003108和12号染色体上SNP(group12:11454172)位点下游6189bp处检测到的泛酸磷酸化酶基因LUSG00023784,表型解释率分别为12.57%和11.56%;3个环境下共同检测到的基因2个,为13号染色体上SNP(group13:21285679)位点上游7626bp处检测到的NPH3家族基因LUSG00005402和SNP(group13:17033610)位点下游9888bp处检测到的细胞过渡金属离子相关基因LUSG00004744,表型解释率为11.24%和12.57%。该6个候选基因在抗倒伏和易倒伏材料的茎和根部位的表达量差异显著,推测这些关键候选基因与亚麻抗倒伏密切相关,为亚麻抗倒伏分子育种提供参考价值。
关键词:亚麻;倒伏;关联分析;SNP;基因
亚麻(Linum usitatissimum L.)属于亚麻科亚麻属,是一年生、长日照、自花授粉植物。亚麻籽中含有α-亚麻酸、膳食纤维、木酚素等可以增强人体免疫力、预防疾病的成分;而亚麻茎秆出麻率在12%~17%,被用作纸浆、塑料合成物、地膜等原材料。由于亚麻具有耐寒及耐旱性,与水稻或小麦等重要作物相比,亚麻通常被种植在贫瘠地、盐碱地以及气候比较恶劣的地区。受环境的影响,亚麻在生长期间易发生倒伏行为,这一行为会使亚麻籽中脂肪酸的合成以及含油量降低,还会使纤维质量下降[1];在亚麻收获期间,倒伏会造成收获的困难,因此需要投入更多的精力与劳动力,导致生产成本增加。因此,倒伏限制了亚麻的生产潜力,阻碍了亚麻产业的发展。
亚麻茎秆作为植株重量的物理支撑部位,其形态和机械属性都直接与亚麻抗倒伏性能相关,例如株高、茎粗、抗折力、断裂力等在产生茎倒伏阻力方面起着至关重要的作用。作物株高在一定范围内,株高越高,倒伏率越高,同时,茎秆越粗,植株越不易倒伏[2]。据研究较短的基茎的水稻,具有较高的抗倒伏能力[3]。对苦荞茎秆形态进行研究后,发现株高、节间数、第一节间长度、第一节间直径、第一侧根数和根系体积是影响苦荞倒伏发生率的关键特征[4]。在玉米中也有同样的情况,玉米株高、穗高、重心高度和茎直径和倒伏百分比呈极显著负相关[5]。由此可见,茎形态指标是倒伏倾向的可靠预测指标,结构弯曲强度提高的茎秆抗倒伏能力[6]。
目前,已有大量作物株高、壁厚、茎秆抗折力、木质素含量、纤维素含量[7-12]等性状相关的全基因组关联分析,挖掘获得了大量的抗倒伏相关的基因[13],这些基因的挖掘为作物抗倒伏分子育种提供了一定的理论基础。卫冰雁等人通过基因分型测序(GBS)定位到Glyma.02G167100、Glyma.02G167700、Glyma.02G168100、Glyma.02G168500和Glyma.02G169000抗倒伏基因[14]。吴家辉在小麦2D染色体筛选到了木质素合成相关的T2DG3基因[15]。张德新利用小麦235对SSR分子标记对其进行关联分析,获得了株高、茎秆强度和倒伏指数关联的标记有6个[16]。王剑锋以105份小麦品种组成的自然群体为试验材料,于2014~2016连续三年测定其茎秆强度,结合SSR标记和90KSNP芯片扫描的基因型,将控制小麦茎秆强度定位到2B、3B、4A、6B、7B等染色体上[17]。刘荣华通过对苦荞差异表达基因及转录本SNPs分析,获得了苦荞矮化性状相关TTG1、Wdr70、ABCG21、ABC1和UGT92A1的候选基因[18]。以上研究均说明作物抗倒伏相关QTL研究可以获得关键基因,但亚麻抗倒伏相关基因挖掘研究鲜见。本研究测定了269份亚麻种质资源在2年5个环境下的10个性状,并对性状进行全基因组关联分析,定位与亚麻抗倒伏性相关联的SNP位点,挖掘与抗倒伏相关的候选基因,为亚麻抗倒伏育种提供理论基础。
1材料与方法
1.1试验材料及试验地概况
供试材料为国内外收集的269份亚麻种质资源,其中124份材料来自国内,145份材料来自国外,具体材料名称,编号以及来源详见附表1(见首页OSID码)。将材料于2022和2023年种植于内蒙古呼和浩特市内蒙古农业大学试验基地(东经111°41′、北纬40°48′,属中温带大陆性季风气候,年均温7.70℃,年均降水量335.2~534.6mm,无霜期145d左右)、内蒙古呼和浩特市和林格尔县盛乐镇哈拉沁村(东经111.79′、北纬40.45′,年平均气温6.2℃,年均降水量375~415mm);于2022年种植于内蒙古乌兰察布市集宁区乌兰察布市农林科学研究院试验基地(东径113°10′,北纬40°01′,年平均气温4.5℃,年平均降水量369mm)。
1.2田间种植及抗倒伏性状测定方法
试验采用完全随机区组设计,每个环境下每份材料种植3行,行长1m,每行种200粒种子,行距0.25m,3次重复。播种期为5月上旬,各个环境水肥管理条件一致。于亚麻青果期随机取长势一致的植株10株,使用SY-S03植物茎秆强度测量仪对亚麻主茎茎秆基部进行测定茎秆抗折力、茎秆断裂力、茎秆抗压力、茎秆穿刺力。株高采用直尺度量亚麻植株从子叶痕到一级分枝顶部的距离,用游标卡尺度量亚麻植株主茎茎秆基部的茎粗。重心高度采用直尺测量植株两端重量相等时与基部的距离。在亚麻青果期,使用百分之一天平测定鲜重,使用ELISA试剂盒测定纤维素含量和木质素含量。使用EXCEL进行数据统计及整理,使用SPSS.26进行描述性统计分析,使用Chiplot进行相关性分析。
1.3全基因组关联分析
1.3.1基因型检测与连锁不平衡分析
本研究对269份亚麻自然群体进行全基因组重测序,与亚麻内亚九号组装获得参考基因组(https://doi. org/ 10. 5281/zenodo. 7811972)比对,获得了8079106万个SNP标记。再使用Plink软件去除缺失率>0.2和最小等位基因频率MAF<0.05的SNP后,得到了2114424万个高质量SNP。使用“--ld”命令计算不同SNPs之间等位基因频率相关性的平方(r2),用于评估连锁不平衡(LD)水平。将LD衰减程度定义为r2下降到最大值的一半时在染色体上对应的物理距离。
1.3.2群体结构分析使用过滤后得到的高质量
SNP依次进行群体结构、系统发育树构建和主成分分析。使用Admixture(v1.3.0)软件进行群体结构分析,群体的数量K值被预先设置为1~15,选择变异系数误差最小的K值作为最优种群数。最后使用Plink软件进行主成分分析(PCA),PCA的前两个轴用于绘制散点图,用于可视化基因型之间的遗传分化。
1.3.3全基因组关联分析及候选基因检测
用GEMMA(http://www. xzlab. org/software. html)软件的混合线性模型(MixedLinearModel,MLM)方法,以主成分分析和亲缘关系矩阵做协变量与5个环境获得的表型数据和BLUP值进行分别GWAS分析,显著性P的阈值为依据本研究获得的具体实验结果(-log10(0.05/标记数)确定为P=5.1。曼哈顿图和分位数-分位数(Q-Q)图是使用CMplotR软件生成的。候选基因的区域由平均LD衰减距离定义,在与某个性状显著相关的SNP标记两侧的10kb区域内搜索候选基因。使用Eggnog和Pfam软件对基因进行功能注释。
1.4 qRT-PCR技术
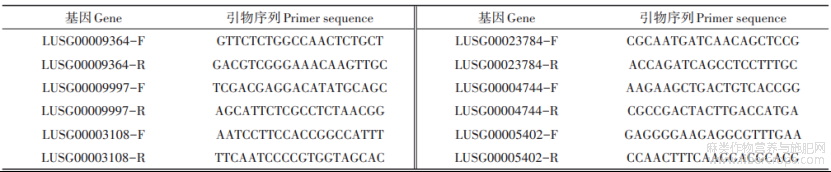
以青果期的亚麻根及茎秆为材料,选取抗倒伏相关基因,根据其全长cDNA序列,用PrimerPremier5.0设计引物(表1),以EF1A为内参基因。使用MightyScript对提取的RNA进行gDNA去除以及第一链cDNA合成。参照SGExcel FastSYBR qPCR 预混液试剂盒的反应体系和反应程序进行qRTPCR分析,按照2-ΔΔCT法计算基因的相对表达量,3个生物学重复。
表1 用于qRT-PCR检测的目的基因与引物序列
2结果与分析
2.1 269份亚麻种质资源茎秆性状的表型变异分析
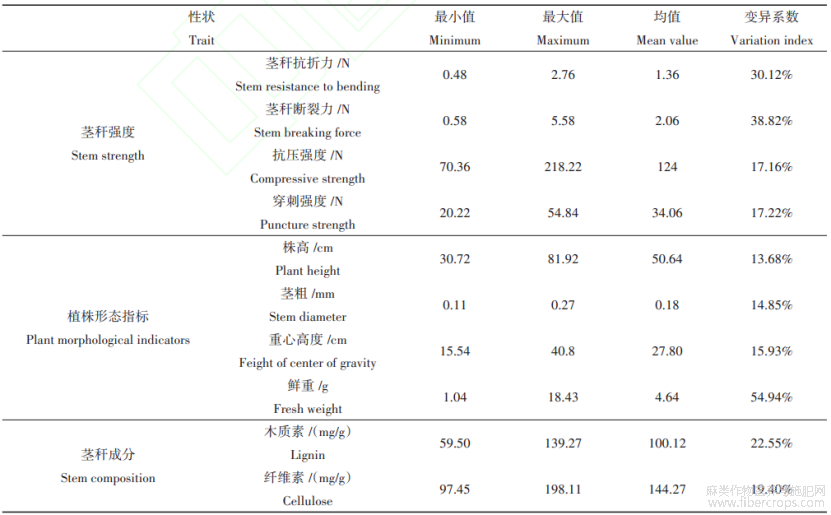
对269份亚麻种质资源抗倒伏相关性状的表型变异分析结果表明(表2),亚麻抗倒伏性状存在广泛的表型变异,其中鲜重的变异系数最大(54.94%),分布范围为1.04~18.43g,其平均值为4.64g;株高变异系数最小(13.68%),分布范围为30.72~81.92cm,其平均值为50.64g。亚麻抗倒伏10个性状的表型变异系数依次排序为鲜重>茎秆断裂力>茎秆抗折力>木质素>纤维素>穿刺强度>抗压强度>重心高度>茎粗>株高。从正态分布检验结果发现,10个抗倒伏相关性状均呈现正态分布的趋势(图1),说明亚麻倒伏相关性状主要受基因的影响。对不同环境抗倒伏相关性状分别进行统计分析结果表明,在2022年呼和浩特市(Hohhot)、2022年呼和浩特市和林县、2022年集宁(Jining)、2023年呼和浩特市(Hohhot)、2023年呼和浩特市和林县等5个环境下该群体木质素的广义遗传力最大,达到了65.72%,表型变异范围分别表现为100.316±9.46mg/g、97.753±7.26mg/g、100.670±8.36mg/g、100.564±8.66mg/g、101.294±8.76mg/g;重心高度的广义遗传力最小,为51.12%,表型变异范围分别表现为34.6±1.26cm、25.7±3.56cm、23.7±4.26cm、30.0±2.56cm、25.0±3.86cm。10个性状的广义遗传力依次排序为木质素>纤维素>鲜重>茎秆断裂力>抗压强度>穿刺强度>茎秆抗折力>茎粗>株高>重心高度(表3)。说明10个抗倒伏相关性状中木质素受环境影响较小,主要由基因控制,而重心高度受环境影响较大。木质素、纤维素、鲜重、株高、重心高度等4个性状与亚麻抗倒伏密切相关,影响亚麻倒伏的主要性状。在5个环境下,2022年和2023年呼和浩特市种植的亚麻种质的鲜重、株高、重心高度平均值均大于其他2个地区,但茎秆强度、木质素和纤维素含量相对低于其他2个地区,说明呼和浩特市种植的亚麻更容易倒伏的风险。
表2 269份亚麻种质资源抗倒伏指标评价
表3 269份亚麻种质资源5个环境的抗倒伏指标评价

2.2亚麻茎秆性状的相关性分析
对269份亚麻种质资源抗倒伏10个性状进行相关性分析,有21对性状之间呈极显著正相关关系,有5对性状之间呈显著正相关关系(图2)。茎秆抗折力与茎秆穿刺强度、株高、茎粗、鲜重呈极显著正相关关系,其中与茎秆断裂力的相关性最大(R2=0.30,P<0.0001);茎秆断裂力与茎秆穿刺强度、茎秆抗压强度、株高、茎粗、鲜重、抗倒伏指数呈极显著正相关关系,其中茎秆穿刺强度的相关系数最大(R2=0.75,P<0.0001);茎秆穿刺强度与茎秆抗压强度、株高、茎粗、重心高度、鲜重、抗倒伏指数呈极显著正相关关系,其中株高相关系数最大(R2=0.21,P<0.001);为了进一步解析抗倒相关的10个性状与抗倒伏指数(重心高度×地上部鲜重/茎秆抗折力)之间的相关性分析结果表明,鲜重、茎粗、株高、茎秆抗压强度、茎秆断裂力与倒伏指数呈极显著正相关,其中鲜重的相关系数最大(R2=0.86,P<0.0001),其次茎粗(R2=0.43,P<0.0001)说明鲜重和茎粗是影响亚麻倒伏的主要因子。
2.3基因型数据分析
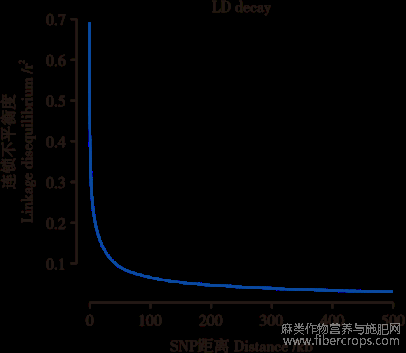
对269份亚麻种质资源材料中发现了807.9106万个SNP标记。基于缺失率>0.2且MAF小于0.05的标准得到了211.4424万个SNP标记。使用过滤后的SNP的等位基因频率相关性的平方值(r2)评估全基因组的LD衰减水平。r2在269份亚麻种质资源中呈现出明显的LD衰变模式。由图3所示,r2=0.34确定为LD衰减的阈值,LD衰减距离约为10Kb(图3)。

图1 亚麻种质资源10个抗倒伏相关性状正态分布图

图2 269份亚麻种质资源相关性图
注:****、***、**和*分别表示在0.0001、0.001、0.01和0.05水平相关性显著;橙色为正相关,淡蓝色为负相关;SBR:茎秆抗折力;SBF:茎秆断裂力;SCS:抗压强度;SPS:穿刺强度;PH:株高;SD:茎粗;CGH:重心高度;FW:鲜重;LC:木质素;CC:纤维素
图3 连锁不平衡衰减图
2.4茎秆强度相关性状的全基因组关联分析
对茎秆强度相关性状的全基因组关联分析获得了显著关联的SNP位点24个,表型解释率为6.45%~12.35%。在显著关联的SNP位点(-log10≥5.1)上下游10kb范围内注释到27个候选基因(表4)。茎秆抗折力(SBR)显著关联的SNP位点5个(附图1,见首页OSID码),其中group10:12389910位点上游7818bp处获得了PAS结构域基因LUSG00016089,group3:2898917位点下游5213bp处获得了金属-烟碱转运体基因LUSG00027295,group4:23614816位点上游8978bp处获得了富含脯氨酸的核受体辅激活因子基序基因LUSG00030034,group6:25009356位点下游5679bp处获得了苏氨酸蛋白基因LUSG00026454,group3:2893917位点下游9537bp处获得了谷氨酰胺合成酶基因LUSG00027297。茎秆断裂力(SBF)显著关联的SNP位点7个(附图2,见首页OSID码),其中group14:11195182位点上游750bp处获得了亮氨酸拉链蛋白基因LUSG00034335,group7:11360538位点下游22331bp处获得了磷酸盐转运蛋白基因LUSG00032829,group12:11454172位点下游61891bp和上游2649处分别获得了泛酸磷酸化酶LUSG00023784和γ-分泌酶复合物的亚基基因LUSG00023779,group2:13794458位点下游8533bp处获得了肌动蛋白结合酶LUSG00014741,group6:14290811位点上游7648bp处获得了催化脯氨酸酰亚胺肽键的顺反异构酶LUSG00024976,group8:15947472位点上游577bp处获得了2-羟基酸脱氢酶LUSG00007996,group5:16247423位点下游2226bp和上游8446bp处分别获得了组蛋白去乙酰化酶LUSG00019742和蛋白磷酸酶1调节亚基基因LUSG00019740。
抗压强度(SCS)显著关联的SNP位点3个(附图3,见首页OSID码),其中group12:15245742位点上游1593bp处获得了岩藻糖苷酶基因LUSG00024319,group11:17347671位点上游3402bp处获得了脱氧辅蛋白基因LUSG00002253,group3:513196位点上游6843bp处获得了DHHC棕榈酰转移酶基因LUSG00009997。穿刺强度(SPS)显著关联的SNP位点9个(附图4,见首页OSID码),其中group2:13109345位点上游1346bp处获得了半胱氨酸合酶基因LUSG00014630,group11:13767327位点下游8905bp处获得了锌指蛋白基因LUSG00001816,group4:17641615位点上游2484bp处获得了类囊体lumel15.0kDa蛋白LUSG00029714,group13:19366811位点下游1803bp、10961bp、7458bp处分别获得了铁抗坏血酸依赖的氧化还原酶基因LUSG00005072和LUSG00005073、真核核糖体蛋白基因LUSG00005078,group6:26282607位点上游5128bp处获得了Zf-MYND-like锌指蛋白基因LUSG00026640,group2:5770899位点上游9586bp处获得了原叶绿素酸酯依赖的转位子LUSG00013359,group11:9112265位点下游2357bp处获得了GDT1-like蛋白质LUSG00001050,group5:9968933位点上游5164bp处获得了丝氨酸合成酶基因LUSG00018805。茎秆强度相关的24个基因中LUSG00023784和LUSG00009997基因在2个环境中均获得,其余基因均在单个环境中获得。
表4 茎秆强度相关性状关联的候选基因

2.5亚麻植株形态指标相关性状的全基因组关联分析
对亚麻植株形态指标相关性状的全基因组关联分析检测到了显著关联的SNP位点27个,表型解释率为6.45%~12.57%。在显著关联的SNP位点(-log10≥5.1)上下游10kb范围内注释到30个候选基因(表5)。株高(PH)显著关联的SNP位点2个(附图5,见首页OSID码),其中group2:5959692位点上游6730bp处获得了MACPF结构域蛋白基因LUSG00013396,group13:29065848位点下游92bp、4830bp、8926bp和上游4382bp处分别获得了非选择性阳离子通道的谷氨酸门控受体LUSG00006690、适应蛋白结合蛋白基因LUSG00006693、植物细胞内ras-group相关LRR蛋白LUSG00006694和核糖核酸内切酶LUSG00006691;茎粗(SD)显著关联的SNP位点9个(附图6,见首页OSID码),其中group12:11807295位点上游8294bp处获得了泛素蛋白连接酶基因LUSG00023844,group3:15168367位点下游3017bp处获得了组蛋白赖氨酸合成基因LUSG00012298,group7:17013538位点下游6668bp处获得了液泡蛋白分选相关蛋白基因LUSG00033454,group11:20210365位点上游1508bp处获得了DUF620基因LUSG00002695,group1:21761917位点下游4981bp处获得了锌指蛋白质基因LUSG00032398,group13:27928462位点下游660bp处获得了病原体防御和叶片细胞死亡的调节基因LUSG00006492,group12:8492939位点上游9237bp处获得了锌指LUSG00023269,group13:24322786位点上游326bp处获得了耐盐蛋白质LUSG00005970;group13:24322804位点下游1320bp处获得了过氧化物酶LUSG00005968;重心高度(CGH)显著关联的SNP位点6个(附图7,见首页OSID码),其中group8:10834361位点上游1320bp处获得了氨基酰基tR合成酶复合物相互作用的多功能醇蛋白基因LUSG00007479,group7:10941614位点上游249bp和下游3536处分别获得了50S核糖体蛋白LUSG00032802和G3BP-like蛋白质基因LUSG00032804,group1:13728227位点上游8184bp处获得了蛋白酶基因LUSG00031777,group9:11847572位点下游4683bp处获得了吡哆醛-磷酸依赖性转氨酶基因LUSG00020455,group13:25661080位点上游7719bp和下游4383处分别获得了转运的胞质蛋白复合物LUSG00006238和甲基化转移酶蛋白基因LUSG00006240;鲜重(FW)显著关联的SNP位点9个(附图8,见首页OSID码),其中group8:11994840位点上游7750bp处获得了βef1-δ家族基因LUSG00007580,Group8:26914041位点上游1815bp处获得了CBS结构域蛋白基因LUSG00009364,group4:16820064位点下游825bp处获得了富含甘氨酸的r结合蛋白基因LUSG00029650,group13:17033610位点下游9888bp处获得了细胞过渡金属离子LUSG00004744,group13:21285679位点上游7626bp处获得了NPH3家族基因LUSG00005402,group15:2301925位点上游4137bp处获得了WAT1-related蛋白质LUSG00027185,group4:23614816位点上游8979bp处获得了富含脯氨酸的核受体辅激活子基序LUSG00030034,group11:12847015位点上游3581bp处获得了碱基-抗坏血酸转运蛋白LUSG00001655,group8:26914041位点下游5482bp处获得了小核核糖核蛋白LUSG00009366。在亚麻植株形态指标关联的27个基因中LUSG00009364在2个环境下共同获得,表型解释率为10.89%;LUSG00004744和LUSG00005402基因在3个环境下共同获得,表型解释率分别为12.57%和11.24%。
2.6茎秆木质素和纤维素含量的关联分析
对木质素(LC)和纤维素(CC)含量的全基因组关联分析获得了显著关联的SNP位点11个,表型解释率为6.45%~12.57%。在显著关联的SNP位点(-log10≥5.1)上下游10kb范围内注释到14个候选基因(表6),木质素(LC)显著关联的SNP位点4个(附图9,见首页OSID码),其中group11:23793243位点下游6776bp和上游6043处分别获得了锌指基质型蛋白基因LUSG00003112和Ppx/GppA磷酸酶家族基因LUSG00003108,group9:25929843位点下游4686bp处获得了syptobrevin家族LUSG00022421,group11:7172073位点下游1715bp处获得了泛素蛋白连接酶LUSG00000723、group10:9203827位点下游2211bp和9248bp处分别获得了snR输入到原子核基因LUSG00015637和WD重复蛋白基因LUSG00015639;纤维素(CC)显著关联的SNP位点7个(附图10,见首页OSID码),其中group5:16889181位点下游2400bp处获得了MIP水通道蛋白LUSG00019841,group10:17820887位点下游4583bp处获得了乙酸丁酸-辅酶a连接酶LUSG00016994,group4:19111672位点下游9726bp处获得了富含AT的结构域蛋白基因LUSG00029794,group2:19950076位点下游2145bp处获得了糖酵解第一步果糖1,6-二磷酸LUSG00015046,group7:22705565位点上游3493bp处获得了钙调素依赖丝氨酸苏氨酸蛋白LUSG00034181,group1:4758532位点下游5707bp处获得了耐受蛋白LUSG00031462,group12:5654614位点上游2736bp和下游4477处分别获得了泛素E3连接酶LUSG00022777和C4HC3型锌指结构LUSG00022778。木质素和纤维素含量相关的14个候选基因中LUSG00003108在2个环境中均获得,表型解释率为12.57%。
表5 亚麻植株形态指标相关的候选基因

2.7抗倒相关基因的表达验证
通过GWAS分析获得的6个关键候选基因(LUSG00009364、LUSG00009997、LUSG00003108、LUSG00023784、LUSG00004744、LUSG00005402)进行RT-qPCR表达验证(图4)。结果表明,细胞壁合成相关的基因LUSG00009364在抗倒亚麻材料乌42(238号)的茎秆中表达量显著高于易倒材料晋亚6号(265);DHHC棕榈酰转移酶基因LUSG00009997在抗倒材料根系中表达量显著高于易倒材料;Ppx/GppA磷酸酶基因LUSG00003108、泛酸磷酸化酶基因LUSG00023784和NPH3家族基因LUSG00005402均在易倒伏材料茎秆中的表达量显著高于抗倒材料;细胞过渡金属离子相关基因LUSG00004744在易倒材料茎秆中表达量显著高于抗倒材料。说明以上6个基因通过不同代谢通路的调控作用来参与亚麻抗倒伏相关性状的形成。
表6 茎秆木质素和纤维素含量相关的候选基因


图4 6个候选基因的RT-qPCR表达分析
3结论与讨论
亚麻种质资源抗倒伏评价对于提供多样化育种亲本、扩大品种遗传基础、挖掘有效等位基因等方面都是非常重要的。抗倒伏性状能直接反映亚麻的倒伏特征,它是基因和环境互作的表现形式。植物基因比较稳定,因此作物在不同自然环境中产生倒伏差异主要是环境导致的。随着种质资源的表型组学研究迅速发展,基于作物表型关联分析的研究日益增多。种质资源表型数据的准确性直接影响关键基因挖掘,为了降低环境对植物表型数据产生的干扰,有必要在不同环境下鉴定表型数据。本研究对269份亚麻种质资源进行2年5个环境的抗倒伏相关性状测定与评价结果表明,亚麻抗倒伏性状存在广泛的表型变异,鲜重的变异系数最大(54.94%),株高变异系数最小(13.68%),亚麻抗倒伏10个性状的表型变异系数依次排序为鲜重>茎秆断裂力>茎秆抗折力>木质素>纤维素>穿刺强度>抗压强度>重心高度>茎粗>株高。5个环境下该群体木质素的广义遗传力最大,达到了65.72%,重心高度的广义遗传力最小,为46.12%。
广义遗传力依次排序为木质素>纤维素>鲜重>茎秆断裂力>抗压强度>穿刺强度>茎秆抗折力>茎粗>株高>重心高度。269份亚麻种质群体的抗倒伏性状均呈现正态分布的趋势。所有性状的广义遗传力达50%以上。说明该群体遗传多样性丰富,尽管基因型和环境型有显著的互作,但所有性状主要由基因型控制。
传统抗倒伏育种主要依赖表型观察和经验选择,存在周期长、效率低、准确性不足等问题,难以满足现代农业对优良品种的需求。而抗倒伏相关候选基因的挖掘研究,为突破这一困境提供了科学路径。通过鉴定与茎秆强度、株高调控、木质素合成、细胞壁结构等性状相关的关键基因,能够揭示亚麻抗倒伏性的分子遗传机制,明确其应对风雨等环境胁迫的内在调控网络。通过全基因组关联分析挖掘抗倒伏相关的候选基因不仅为亚麻抗倒伏育种提供精准的分子标记和基因靶点,推动分子标记辅助育种、基因编辑等现代生物技术的应用,大幅缩短育种周期,提高优良品种培育效率;还能丰富植物抗逆遗传学理论,为其他作物的抗倒伏研究提供借鉴。本研究对亚麻抗倒伏性状的全基因组关联分析获得了显著关联的SNP位点61个,表型解释率为6.45~12.35%。在显著关联的SNP位点(-log10≥5.1)上下游10kb范围内注释到72个候选基因,其中2个环境下共同检测到的基因4个,分别为8号染色体上SNP(group8:26914041)位点下游1815bp处检测到的细胞壁合成候选基因LUSG00009364,表型解释率为10.89%,3号染色体上SNP(group3:153196)位点上游6843bp处检测到的DHHC棕榈酰转移酶基因LUSG00009997,表型解释率为12.35%,11号染色体上SNP(group11:23793243)位点上游6043bp处检测到的Ppx/GppA磷酸酶基因LUSG00003108和12号染色体上SNP(group12:11454172)位点下游6189bp处检测到的泛酸磷酸化酶基因LUSG00023784,表型解释率分别为12.57和11.56%;3个环境下共同检测到的基因2个,为13号染色体上SNP(group13:21285679)位点上游7626bp处检测到的NPH3家族基因LUSG00005402和SNP(group13:17033610)位点下游9888bp处检测到的细胞过渡金属离子相关基因LUSG00004744,表型解释率为11.24%和12.57%。
本研究获得亚麻抗倒伏显著关联的候选基因与氮代谢、细胞壁合成、激素调控等多种生化代谢途径,为解析亚麻抗倒伏性的分子遗传机制提供了丰富的候选资源。在茎秆抗折力相关基因中,LUSG00027297蛋白(谷氨酰胺合成酶作为氮代谢的关键酶)催化产生的谷氨酰胺是植物氮同化的核心产物,通过增加茎秆化合物增强强度,这与跨物种研究结果高度一致。杨树中该蛋白基因可增加木质部细胞壁碳水化合物[19]。转基因烟草中株系纤维素含量提升使机械强度增强,小麦中通过氮同化改善茎秆发育,均验证了谷氨酰胺合成酶家族基因在调控植物茎秆强度方面的保守功能[20]。表明亚麻LUSG00027297蛋白可能通过调控氮同化效率,影响细胞壁多糖合成,进而强化茎秆抗折能力,为氮代谢调控抗倒伏性状提供了新证据。茎秆断裂力和穿刺力相关的组蛋白脱乙酰酶基因LUSG00019742和LUSG00018805,其功能推断与拟南芥HDT1基因的研究结果相呼应,HDT1通过调控细胞分裂和次生生长影响维管组织发育,直接关联木质部次生壁形成和木质素沉积,这解释了其对茎秆机械强度的调控作用[21]。而O-甲基转移酶基因LUSG00023784在抗倒材料中的高表达特征,进一步证实了木质素合成途径在茎秆强度调控中的关键作用。该基因参与的咖啡酰辅酶A甲基化等反应,是木质素生物合成的关键步骤,与Wang等人发现的该功能基因在强茎秆中高表达的规律一致[22],凸显了木质素代谢通路作为抗倒伏育种靶点的重要价值。在株高、重心高度等株型相关性状中,候选基因功能呈现出多元化调控特征。MACPF结构域蛋白基因LUSG00013396可能通过参与生长发育调控影响株高,与拟南芥、棉花中该家族基因的功能报道相符[23]。重心高度相关的蛋白酶基因LUSG00031777和III类转氨酶基因LUSG00020455,分别通过激素调控和非生物胁迫响应影响植株生长态势,其中III类转氨酶家族在水稻抗逆中的作用[24],暗示其在亚麻抗倒伏与抗逆协同调控中的潜在价值。鲜重相关基因的功能分析揭示了细胞壁成熟对亚麻抗倒伏性的重要影响。CBS结构域蛋白基因LUSG00009364在抗倒材料中的高表达,与其在拟南芥中参与细胞壁成熟的功能一致[25];WAT1相关蛋白基因LUSG00027185则可能通过调控纤维次生壁形成影响茎秆机械强度,这与苎麻中该基因正向选择提升纤维产量的研究结果形成功能关联[26]。纤维素相关基因LUSG00016994和LUSG00034181通过钙信号调控细胞壁多糖合成,进一步完善了细胞壁—机械强度调控网络。此外,E3泛素连接酶基因LUSG00000723和DHHC棕榈酰转移酶基因LUSG00009997的发现,拓展了抗倒伏调控的新视角。前者可能通过调控次生壁合成基因表达影响细胞壁厚度,后者在抗倒材料根系中的上调表达,暗示根系发育与茎秆抗压强度的协同调控机制。
参考文献
[1]王斌, 赵利, 王利民, 等. 胡麻种质资源主要品质性状的分析与评价[J]. 中国油料作物学报, 2018, 40(6): 785-792.
[2]欧巧明, 叶春雷, 李进京, 等. 油用亚麻品种资源主要性状的鉴定与评价[J]. 中国油料作物学报, 2017, 39(5): 623-633.
[3]Zhang J, Li G H, Song Y P, et al. Lodging resistance characteristics of high-yielding rice populations[J]. Field Crops Res, 2014, 161: 64-74. DOI:10.1016/j. fcr.2014.01.012.
[4]Xiang D B, Zhao G, Wan Y, et al. Effect of planting density on lodging-related morphology, lodging rate, and yield of Tartary buckwheat (Fagopyrum tataricum)[J]. Plant Prod Sci, 2016, 19(4): 479-488.
[5]Ahmad I, Batyrbek M, Ikram K, et al. Nitrogen manage?
ment improves lodging resistance and production in maize (Zea mays L.) at a high plant density[J]. J Integr Agric, 2023, 22 (2): 417-433. DOI:10.1016/j. jia.2022.08.074.
[6]Hu H X, Liu W X, Fu Z Y, et al. QTL mapping of stalk bending strength in a recombinant inbred line maize population[J]. Theor Appl Genet, 2013, 126(9): 22572266. DOI:10.1007/s00122-013-2132-7.
[7]Zhao Z C, Duan S, Hao J M, et al. The dwarf gene Rht15 improved lodging resistance but differentially affected agronomic and quality traits in durum wheat[J]. Field Crops Res, 2021, 263: 108058.
[8]Xiang D B, Song Y, Wu Q, et al. Relationship between stem characteristics and lodging resistance of Tartary buckwheat (Fagopyrum tataricum)[J]. Plant Prod Sci, 2019, 22(2): 202-210. DOI:10.1080/1343943X.201 9.1577143.
[9]Kato S, Samanfar B, Morrison M J, et al. Genome-wide association study to identify soybean stem pushing resistance and lodging resistance loci[J]. Can J Plant Sci, 2021, 101(5): 663-670.
[10]Galinousky D V, Anisimova N V, Raiski A P, et al. Cellulose synthase genes that control the fiber formation of flax (Linum usitatissimum L.)[J]. Russ J Genet, 2014, 50(1): 20-27. DOI:10.1134/s1022795414010050.
[11]Wang J, Zhu J M, Lin Q Q, et al. Effects of stem structure and cell wall components on bending strength in wheat[J]. Sci Bull, 2006, 51(7): 815-823. DOI: 10.1007/s11434-006-0815-z.
[12]赵小红, 白羿雄, 姚有华, 等 . 禾谷类作物茎秆特性与茎倒伏关系的研究[J]. 植物生理学报, 2021, 57(2): 257-264. DOI:10.13592/j.cnki.ppj.2020.0343.
[13]卫冰雁 . 大豆重要农艺性状 QTL 定位及应用[D]. 泰安: 山东农业大学, 2023. DOI:10.27277/d.cnki.gsdnu.2023.000996.
[14]侯辛未. 玉米抗倒伏性状QTL基因的精细定位与挖掘[D]. 泰安: 山东农业大学, 2022. DOI:10.27277/d. cnki.gsdnu.2022.001240.
[15]吴家辉 . 小麦茎秆强度 SSR相关位点分析[D]. 合肥:安徽农业大学, 2018.
[16]张德新. 小麦倒伏相关性状的关联分析[D]. 合肥: 安徽农业大学, 2015.
[17]王剑锋 . 小麦茎秆强度相关位点鉴定及 CAPS标记开发[D]. 合肥: 安徽农业大学, 2019. DOI:10.26919/d. cnki.gannu.2019.000773.
[18]刘荣华. 苦荞矮化突变体矮化特征及差异表达基因分析[D]. 太谷: 山西农业大学, 2019. DOI:10.27285/d. cnki.gsxnu.2019.000113.
[19]Coleman H D, Cánovas F M, Man H M, et al. Enhanced expression of glutamine synthetase (GS1a) confers altered fibre and wood chemistry in field grown hybrid poplar (Populus tremula X alba) (717-1B4)[J]. Plant Biotechnol J, 2012, 10(7): 883-889.
[20]Ambavaram M M R, Krishnan A, Trijatmiko K R, et al. Coordinated activation of cellulose and repression of lignin biosynthesis pathways in rice[J]. Plant Physiol, 2011, 155(2): 916-931.
[21]Zhang Y Z, Yin B, Zhang J X, et al. Histone deacetylase HDT1 is involved in stem vascular development in Arabidopsis[J]. Int J Mol Sci, 2019, 20(14): 3452. DOI:10.3390/ijms20143452.
[22]Wang X Q, Chen Y N, Sun X, et al. Characteristics and candidate genes associated with excellent stalk strength in maize (Zea mays L.)[J]. Front Plant Sci, 2022, 13: 957566.
[23]Yu L J, Liu D, Chen S Y, et al. Evolution and expression of the membrane attack complex and perforin gene family in the Poaceae[J]. Int J Mol Sci, 2020, 21(16): 5736. DOI:10.3390/ijms21165736.
[24]Sharma P, Gayen D. Plant protease as regulator and signaling molecule for enhancing environmental stress-tolerance[J]. Plant Cell Rep, 2021, 40(11): 2081-2095. DOI:10.1007/s00299-021-02739-9.
[25]Sinharoy S, Liu C W, Breakspear A, et al. A Medicago truncatula cystathionine- β -synthase-like domain-containing protein is required for rhizobial infection and symbiotic nitrogen fixation[J]. Plant Physiol, 2016, 170 (4): 2204-2217.
[26] Li P, Liu Y R, Tan W Q, et al. Brittle culm 1 encodes a COBRA-like protein involved in secondary cell wall cellulose biosynthesis in sorghum[J]. Plant Cell Physiol, 2019, 60(4): 788-801.