摘 要:InDel标记已广泛用于作物种质资源遗传多样性分析和分子标记辅助育种等研究领域,但在亚麻(Linum usitatissimum)相关研究中报道甚少。本研究利用2份亚麻材料重测序数据鉴定出的InDel位点,开发多态性InDel标记并对93个亚麻育成品种进行基因分型,研究亚麻育成品种遗传多样性、亲缘关系和群体遗传结构特点。结果显示,2份亚麻材料重测序共鉴定出66851个InDel位点,选择15条染色体上120个InDel位点设计引物,筛选出26对多态性好的标记,分析了93个亚麻育成品种的遗传多样性和群体结构。结果表明,26对InDel标记在93个亚麻品种中共检测出80个主要等位变异;基因多样性指数为0.1696~0.6942,平均为0.4852;多态性信息含量(PIC)为0.1615~0.6369,平均为0.4150。经聚类分析全部品种被划分为纤维用亚麻品种群体和油用亚麻品种群体2个类群;群体结构分析将全部品种分为4个类群,主成分分析可将纤维用亚麻品种和油用亚麻品种分开;分子方差分析(AMOVA)表明,亚麻品种遗传变异主要发生在个体间和个体内,种群间也存在一定的变异。聚类分析与主成分分析结果较一致,纤维用亚麻群体遗传多样性高于油用亚麻群体,油用亚麻育成品种的遗传变异与地理来源无关。本研究在一定程度上揭示了93个亚麻育成品种的亲缘关系,在亚麻品种鉴定、亚麻资源亲缘关系分析以及分子辅助育种方面具有一定的理论意义和应用价值。
关键词:亚麻;InDel标记;遗传多样性;群体结构分析
亚麻(Linum usitatissimum)是我国北方重要的油料作物之一,按用途可分为油用亚麻和纤用亚麻,油用亚麻主要种植于我国甘肃、内蒙古、宁夏、河北、新疆、山西六个产区,目前陕西和青海也有少面积种植;纤维用亚麻主要分布在东北的黑龙江、吉林等地(王斌等,2022)。亚麻育种仍然以常规育种技术为主,育种周期长,效率低。采用分子标记辅助选择技术对目标基因进行鉴定,更为准确、简便,且不受环境影响,效率较高。现有技术中亚麻品种及纯度鉴定的方法主要是田间表型鉴定,由于亚麻种质资源的相互交流,部分亚麻品种的亲缘关系较近,难以用形态标记快速地鉴别品种的真实性及纯度。目前,亚麻种质资源研究常用的分子标记有AFLP、RAPD、SSR、SNP等标记。李明(2011)利用AFLP标记分析了亚麻种质资源遗传多样性与亲缘关系,郝荣楷等(2014)利用RAPD标记分析了我国油用亚麻育成品种的遗传多样性,安泽山等(2014)利用SRAP标记分析了油用亚麻种质资源遗的传多样性,吴建忠等(2013)利用SSR标记构建了亚麻遗传连锁图谱,王斌等(2018)利用SSR标记分析了8个地方野生亚麻的遗传多样性,Zhang等(2018)利用SNP标记构建了亚麻遗传连锁图并定位了株高QTL位点。已开展的遗传多样性研究结果显示,亚麻种质资源遗传基础狭窄,遗传多样性不高,可能是用于亚麻遗传多样性研究的分子标记较少且多态性不高,不能准确反映亚麻不同材料间的遗传多样性。随着测序技术的发展,通过高通量基因组数据开发InDel标记已经越来越多(蒲伟军等,2023;陶顺玉等,2023;王瑞等,2022;陈正杰等,2021;徐婷婷等,2022;陆海燕等,2019)。InDel分子标记具有多态性高、重复性好、易于检测等优点,目前已在许多作物相关研究中得到应用,如水稻(Oryza sativa) DNA 指纹图谱的构建 ( 赵春芳等 , 2022)、谷子(Setaria italica)株高 QTL 定位(杜晓芬等, 2024)、甘薯(Ipomoea batatas)育成品种遗传多样性分析(唐芬等, 2024)、花生(Arachis hypogaea)含油量 QTL 定位(陶顺玉等, 2023)、甘蓝型油菜(Brassica napus)白花基因 InDel 连锁标记开发(王瑞等, 2022)、海岛棉(Gossypium barbadense)纤维强度 QTL 紧密连锁 InDel 分子标记开发(吕涛等, 2025)、番茄 (Solanum lycopersicum)果实性状相关候选基因的挖掘(董洁等,2020)。利用InDel标记在亚麻品种鉴定、种质资源遗传多样性分析、指纹图谱构建等方面的研究还没有相关报道。因此,开发亚麻InDel标记,对亚麻育种具有重要的意义。本研究利用开发的26对InDel标记分析了93个亚麻育成品种的遗传多样性,并解析不同类型亚麻育成品种的亲缘关系、群体遗传结构和遗传背景,旨在为亚麻品种改良、种质创新和种质资源有效利用提供参考。
1 材料与方法
1.1实验材料
重测序亚麻(Linum usitatissimum)材料为'1S'和'陇亚10号',其中'陇亚10号'为甘肃农业科学院作物研究所选育的大面积推广品种,'1S'是诱变常规品种选育的温敏雄性不育系(党占海等,2002)。选择亚麻主要产区代表性品种及部分国外资源共93份,其中63份为油用亚麻(Pop1:序号1~63),30份纤维用亚麻(Pop2:序号64~93),以上材料保存于甘肃农业科学院作物研究所(表1)。
1.2亚麻基因组重测序和InDel位点分析
2024年利用培养皿发芽,取幼嫩叶片采用CTAB法提取亚麻基因组DNA。由南京集思慧远生物科技有限公司完成'1S'和'陇亚10号'基因组重测序,与已公布的亚麻参考基因组(https://www.ncbi. nlm. nih. gov/datasets/genome/? taxon=4006 (longya 10))进行比对,在参考基因组上获取InDel位点的位置信息。
1.3筛选InDel位点和设计引物
随机选择分布于亚麻参考基因组15条染色体上120个插入/缺失碱基数大于10的InDel位点,使用PrimerPremier5.0设计引物,由擎科生物技术有限公司(西安)合成引物。
1.4 InDel标记有效性验证
以8份不同地理来源的亚麻品种('陇亚10号','伊亚4号','宁亚21号','坝亚6号','轮选3号','晋亚6号','黑亚23号','Bisow'),验证120对InDel引物的PCR扩增有效性,筛选出26对多态性好的引物,对93个品种进行遗传多样性分析。PCR扩增体系为20μL,包括4.0μL基因组DNA(50ng/μL),10.0μL2×PCRMix,上、下游引物(10μmol/L)各2μL,2.0μLddH2O。扩增程序为:94℃预变性5min;94℃变性30s,55~60℃退火30s(根据不同引物设置退火温度),72℃延伸45s,35个循环;72℃延伸5min。6%PAGE凝胶电泳(1×TBE缓冲液)检测PCR扩增产物,银染并进行拍照。
1.5数据统计分析
统计PCR扩增产物条带,使用PowerMarker3.25和GenAlEx6.51软件计算包括主要等位基因频率、等位基因数量、基因多样性、杂合性、多态信息含量等遗传多样性指标。使用PowerMarker3.25计算各材料间的Nei's遗传距离,随后基于邻接法在MEGA5上构建聚类图。使用Structure2.3.3进行群体结构的分析。通过GenAlEx6.51进行主成分分析和分子变异分析(AMOVA),利用这些数据评估各种群间的遗传分化和遗传变异特征。
2 结果与分析
2.1亚麻InDel标记的开发和有效性鉴定
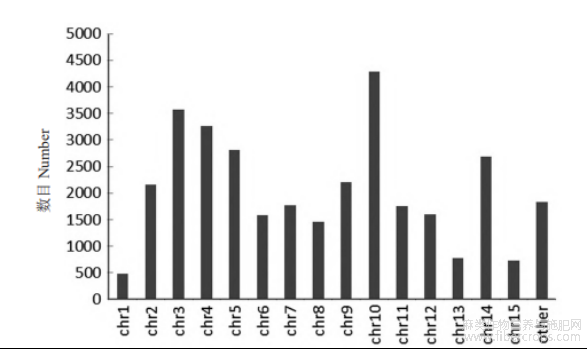
与亚麻参考基因组序列比对,2份亚麻材料重测序数据共有66851个InDel位点分布于不同染色体上(图1)。选取15条染色体上120个InDel位点设计引物,以8份不同地理来源的亚麻品种验证InDel引物PCR扩增的有效性,筛选出26对多态性好的引物。分布于亚麻15条染色体的11条染色体上,扩增片段大小在100~300bp之间(表2)。
2.2 InDel标记遗传多样性分析
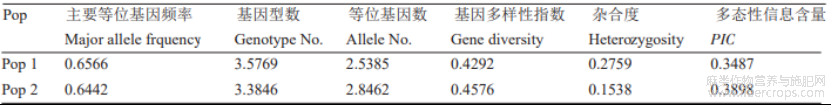
26对InDel标记在93份亚麻育成品种检测结果表明(表3),主要等位基因频率在0.3871~0.9086之间,平均为0.6059;基因型数在3~7之间,平均为4.1154;等位基因数在2~4之间,平均为3.0769;基因多样性指数为0.1696~0.6942,平均为0.4852;杂合度为0.0108~0.8710,平均为0.2366;多态性信息含量(polymorphic information content, PIC)值在0.1615~0.6369之间,平均为0.4150;26个InDel标记中有23个PIC值较大(PIC>0.200),多态性较好,只有IDE23、IDE45和IDE693个标记PIC值较小。Pop1为油用亚麻群体,Pop2为有纤维用亚麻群体,从表4可以看出,纤维用亚麻群体等位基因数、基因多样性指数、PIC值高于油用亚麻群体,油用亚麻群体等位基因频率、基因型数、杂合度高于纤维用亚麻群体。
2.3聚类分析
基于Nei1973遗传距离邻接法对93个亚麻品种进行的聚类分析,结果(图2)表明,全部品种被划分为2个类群。群Ⅰ中有31个品种,其中纤维用亚麻品种28个,油用品种3个;群Ⅱ共计62个品种,60个油用亚麻品种,2个纤维用亚麻品种;基本可以按用途分为油用亚麻品种和纤维用亚麻品种2个群。
2.4群体结构分析和主成分分析
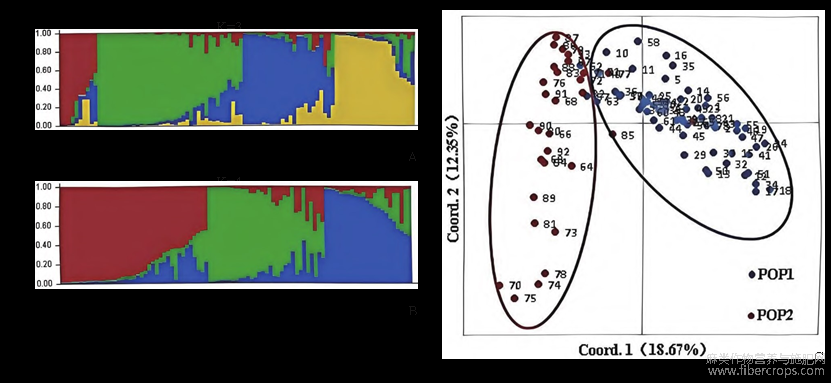
对93份亚麻育成品种进行群体遗传结构分析,结果表明,当K=4时,DeltaK出现明显峰值,将供试材料划分为4个类群(图3A)。其中红色群(10个)为油用亚麻品种,河北、甘肃、山西、内蒙、宁夏都有品种;绿色群(38个)为9个油用亚麻品种,29个纤维用亚麻品种;蓝色组为24个油用亚品种,黄色组为20个油用亚麻品种和1个纤维用亚麻品种。当K=3时,也表现较高的DeltaK值,此情况下红色组39个品种组成,30个纤维用亚麻品种,9个油用亚麻品种(图3B),绿色组和蓝色组均为油用亚麻品种。可见,不同群体结构分析方法,纤维用亚麻品种都聚类在一起,2个类群有少部分品种不能明显分开。
表1 93个亚麻育成品种信息

图1 InDel位点在亚麻基因组上的分布
表2多态性InDel标记的信息

表3 26个InDel标记多态性信息

表4 26个InDel标记在2个群体间的多态性信息
为了更好地验证油用亚麻品种和纤维用亚麻品种之间的遗传关系,对93个亚麻品种进行主成分分析。从图3C上可以看出,Pop1油用亚麻品种分布在一起,油用亚麻育成品种间的亲缘关系与地理来源没有相关性;不同产区亚麻品种基因交流频繁,表明油用亚麻育成品种的亲缘关系比较近,遗传基础狭窄。Pop2纤维用亚麻品种聚在一起,只有少部分几个品种不能分开,这与聚类分析结果基本一致。
图2 93份亚麻品种基于Nei遗传距离的聚类
图3供试亚麻品种的群体结构图和主成分分析
2.5亚麻种群变异分布
对2个种群进行了分子方差分析(AMOVA)。结果(表5)表明,个体内变异可解释总变异的43%,个体间的变异占38%,种群间的变异占18%。个体内遗传变异大于个体间遗传变异,个体间遗传变异大于种群间遗传变异,说明亚麻品种群体间存在一定程度的遗传变异,但主要变异存在于个体间和个体内。
3讨论
3.1亚麻全基因组InDel变异特征和标记开发
目前,亚麻常用的分子标记有AFLP、RAPD、SSR以及SNP。最早在亚麻种质资源遗传多样性分析、亲缘关系鉴定中应用较多的是AFLP和RAPD标记,这2个标记因操作繁琐、周期长,重复性差等原因已被SSR、SNP等标记替代。SSR和SNP广泛存在于亚麻基因组,目前通过高通量测序,利用生物信息学方法分析挖掘更多有效的变异位点,在亚麻高密度遗传图谱构建、全基因组关联分析(genome-wide association study, GWAS)、QTL定位等研究中广泛应用(姜慧等,2021)。然而,SNP检测成本高,要求的仪器设备需求无法大范围普及和推广。InDel标记在基因组内分布广、密度高、变异稳定、多态性强、检测成本低,操作简单易行;在许多作物上取得了较大的研究进展,但在亚麻相关研究中还没有相关报道。
本研究基于2份亚麻材料重测序数据,共检测到66851个InDel位点,远低于陈正杰等(2021)基于18份大豆(Glycinemax)种质资源重测序获得的InDel位点数量和吉康娜等(2019)在2份茄子(Solanum melongena)重测序获得的InDel位点数量。可能与不同物种中InDel的位点数量存在较大差异有关,另外,测序群体大小以及设置的InDel过滤阈值不同也能造成InDel的位点数量差异。本研究筛选分布于全基因组的120个InDel位点,利用8份不同地理来源的国内外亚麻品种进行检测,其中26对标记具有多态性,多态性比例为21.67%;低于徐婷婷等(2020)在大麦(Hordeum vul?gare)中开发InDel标记筛选出的多态性比例,也低于茄子多态率(34.7%)(吉康娜等,2019)。本研究使用非变性聚丙烯胺凝胶电泳分析,与吉康娜等(2019)等选择InDel的选择标相同,都大于10bp,从而增加假阳性,导致亚麻InDel标记的多态率没有其他作物高。下一步研究对测序数据进行深度分析,筛选质量更高的InDel位点,以便尽可能多的筛选出多态性亚麻InDel标记。Soto-Cerda等(2012)用150个SSR标记研究60份亚麻种质的遗传多样性,平均等位基因数为2.72,基因多样性指数平均为0.34,表明遗传多样性比较低;本研究开发的In‐Del标记平均等位基因数量和平均基因多样性高于SSR标记。Soto-Cerda等(2013)用448个SSR标记分析了407个全球分布的亚麻种质,观察到丰富的遗传多样性,平均每个位点5.32个等位基因,高于本研究开发的InDel标记,可能与选择的材料少有关。Hoque等(2020)利用6200个SNP标记分析了350个全球分布的亚麻材料的遗传多样性,表现出中等遗传多样性(平均H=0.22,PIC=0.34);而本研究开发的InDel标记平均PIC值高于SNP标记。通过与亚麻上常用的SSR和SNP标记比较,本研究开发的InDel标记平均等位基因数、基因多样性和PIC值高于SSR和SNP。研究表明,亚麻品种亲缘关系比较近,遗传基础狭窄有关,研究材料选择的多少以及选择范围,影响InDel标记的遗传多样性信息;本研究选择的材料多数来源于国内,而且多为栽培种,应增加国内外种质资源,方能客观准确的评价InDel标记遗传多样性信息。
3.2分子标记在亚麻种质资源研究中的应用
我国亚麻主要分为油用亚麻种植区和纤维用亚麻种植区,油用亚麻主要种植于西北和华北,纤维用亚麻主要分布在东北及云南等南方冬播地区。不同亚麻产区品种,不同种质资源间的遗传多样性以及遗传关系的研究国内外都有报道。Fu等(2005)利用RAPD标记对来自63个国家的2727个亚麻栽培种进行遗传多样性分析表明,所有品种根据地理区域划分12个群体,只有15.8%的RAPD变异存在于不同国家的种群间,大多数RAPD变异(84.2%)存在于品种间;本研究表明,个体内变异占总变异的43%,个体间变异占总变异的38%,种群间变异只有18%;李明(2011)对85份不同国家的亚麻种质资源利用AFLP标记进行遗传多样性分析,结果表明,纤维用亚麻的遗传多样性远低于油用亚麻;而我国纤维用亚麻品种相似度极高,遗传背景狭窄。本研究对油用和纤维用亚麻品种群体进行比较,纤维用亚麻群体遗传多样性高于油用亚麻群体,与李明(2011)研究结果不一致。郝荣楷等(2014)利用SRAP标记对我国不同地区油用亚麻品种进行分析,来自不同地区品种之间没有很大的差异,与地域的联系并不紧密,与本研究油用亚麻品种聚类结果一致,说明与不同地区栽培种存在着广泛引种有关系。Soto-Cerda等(2012)用150个SSR标记来评估60份亚麻种质的遗传多样性,分子变异分析表明,78.32%的遗传变异存在于群体内部,21.68%存在于群体之间,与本研究结果基本相同;Hoque等(2020)利用6200个SNP标记分析了350个全球分布的亚麻种质资源的遗传多样性,将所有的品种聚类成7个亚群;AMOVA显示,亚群之间的方差覆盖了总变异的28%,其余72%则解释了亚群内个体之间的方差,与本研究结果基本相同。本研究聚类分析和群体结构分析表明,大部分油用亚麻品种的遗传差异与地理来源无关,油用品种没有明显的区域特征,可能是不同亚麻主产区品种经过多次基因重组所致;纤维用亚麻品种遗传多样性高于油用亚麻品种,可能与纤维用亚麻品种中有10个外国资源有关;聚类分析和主成分分析基本可区分油用亚麻品种和纤维用亚麻品种。这些结果为亚麻新品种选育过程中挖掘优良基因和拓宽遗传基础提供重要的利用价值,也有助于对栽培亚麻的种内多样性进行分类,并为亚麻品种的遗传改良提供新的候选分子标记。
4 结论
本研究利用重测序数据检测到的66851个InDel位点,开发出26对多态性InDel标记,对93份亚麻品种进行遗传多样分析。共检测出80个主要等位变异,平均每个位点3.08个;平均基因多样性指数和多态性信息含量分别为0.4852和0.4150。聚类分析和主成分分析把全部品种被划分为纤维用亚麻品种和油用亚麻品种2个类群,纤维用亚麻群体遗传多样性高于油用亚麻群体,油用亚麻育成品种间的亲缘关系与地理来源没有相关性。群体结构分析将全部品种分为4个类群,分子方差分析表明亚麻品种个体间和个体内遗传变异高于种群间。本研究开发的26对InDel标记可用于亚麻遗传图谱构建及基因定位、种质资源评价、新品种鉴定和保护,为亚麻分子标记辅助育种增加了新的分子标记种类和数量。
参考文献
[1] 安泽山, 严兴初, 党占海, 等. 2014. 利用 SRAP标记分析胡麻资源遗传多样性[J].西南农业学报, 27(2): 530-534. (An Z S, Yan X C, Dang Z H, et al. 2014. Genetic diversities analysis of Flax based on SRAP[J]. Southwest China Journal of Agricultural Sciences, 27 (2): 530-534.)
[2] 陈正杰, 宛永璐, 钟文娟, 等. 2021. 基于大豆基因组重测序的 InDel 标记开发及应用[J]. 植物遗传资源学报, 22(3): 815-833. (Chen Z J, Wan Y L, Zhong W J, et al. 2021. Development and application of soybean InDel markers based on whole-genome resequencing datasets[J]. Journal of Plant Genetic Resources, 22(3): 815-833.)
[3] 党占海, 张建平, 佘新成. 2002. 温敏型雄性不育亚麻的研究[J]. 作物学报, (6): 861-864+874. (Dang Z H, Zhang J P, She X C. 2002. Studies on thermo-sensitivity male-sterile Flax[J]. Acta Agronomica Sinica, (6): 861-864+874.)
[4] 董洁, 王娜, 火顺利, 等. 2024. 基于InDel标记挖掘与番茄果实性状相关的候选基因[J]. 农业生物技术学报, 32(7): 1494-1503. (Dong J, Wang N, Huo S L, et al. 2024. Identification of candidate genes related to fruit traits using InDel markers of Tomato (Solanum lycopersicum)[J]. Journal of Agricultural Biotechnology, 32(7): 1494~1503.)
[5] 杜晓芬, 钱枰励, 唐楚楚, 等. 2024. 基于InDel标记的谷子株高 QTL 定位[J]. 核农学报, 38(2): 217-230. ( Du X F, Qian P L, Tang C C, et al. 2024. QTL mapping for plant height in foxtail millet based on InDel markers[J]. Journal of Nuclear Agricultural Sciences, 38(2): 217-230.)
[6] 郝荣楷, 严兴初, 党占海, 等. 2014. 我国胡麻育成品种的遗传多样性分析[J]. 中国油料作物学报, 36(3): 334-342.(Hao R K, Yan X C, Dang Z H, et al. 2014. Morphological and SRAP markers on genetic diversity of flax cultivars in China[J].Chinese Journal of Crop Sciences, 36(3): 334-342.)
[7] 姜慧, 潘根, 常丽, 等. 2021. 亚麻分子标记辅助育种研究进展[J]. 植物遗传资源学报, 22(4): 910-920. (Jiang H,Pan G, Chang L, et al. 2021. Advances in molecular marker assisted breeding of flax[J]. Journal of Plant Genetic Resources, 22(4): 910-920.)
[8] 李明. 2011. 亚麻种质资源遗传多样性与亲缘关系的AFLP 分析[J]. 作物学报, 37(4): 635-640. (Li M. 2011. AFLP analysis of genetic diversity and genetic relationship of flax germplasm resources[J]. Acta Agronomica Sinica,37(4): 635-640 .)
[9] 陆海燕, 陈璐, 王显生, 等. 2019. 基于高通量测序的棉花InDel 标记开发及其应用[J]. 棉花学报, 31(4): 297-306. (Lu HY,Chen L, Wang X S, et al. 2019. Development and application of cotton InDel markers based on high throughput sequencing[J]. Cotton Science, 31(4): 297-306.)
[10] 吕涛, 孙国清, 郭栋材, 等. 2025. 海岛棉纤维强度QTL紧密连锁InDel分子标记开发及效应评价[J]. 中国农业科学, 58 (9): 1684-1713. (Lv T, Sun G Q, Guo D C, et al. 2025. Development and effectiveness evaluation of InDel molecular markers closely linked to fiber strength QTL in Gossypium barbadense[J]. ScienTia Agricultura Sinica, 58(9): 1684-1713.)
[11] 蒲伟军, 谭冰兰, 贺丹晨, 等. 2023. 利用重测序技术开发高粱InDel分子标记[J]. 生物技术进展, 13 (05): 730-741.(PuW J, Tan B L, He D C, et al. 2023. Development of InDel molecular markers in sorghum using re-sequencing technology[J].Current Biotechnology, 13(5): 730-741.)
[12] 唐芬, 赵路宽, 苏一钧, 等. 2024. 基于InDel标记分析305份中国甘薯登记品种遗传多样性[J]. 植物遗传资源学报, 25(4): 576-591. (Tang F, Zhao L K, Su Y J, et al. 2024. Genetic diversity analysis of 305 registered sweet potato varieties in China based on InDel markers[J].Journal of Plant Genetic Resources, 25 (4): 576-591.)
[13] 陶顺玉, 吴贝, 刘念, 等. 2023. 花生InDel标记开发及其在含油量QTL定位中的应用[J]. 作物学报, 49 (05): 1222-1230. (Tao S Y, Wu B, Liu N, et al. 2023. Development and employment of InDel marker in peanut QTL mapping of content[J]. Acta Agronomica Sinica, 49(5):1222-1230.)
[14] 王斌, 赵利, 侯静静. 2022. 国内亚麻育成品种品质性状的分析与评价[J]. 中国粮油学报, 37(6): 135-140. (Wang B,Zhao L, Hou J J. 2022. Quality traits analysis and evaluation of flax cultivars in China[J]. Journal of the Chinese Cereals and Oils Association, 37(6): 135-140.)
[15] 王斌, 赵利, 赵玮. 2019. 8个地方野生亚麻资源发掘及遗传多样性分析[J]. 分子植物育种, 17(11): 3755-3760.(Wang B, Zhao L, Zhao W. 2019. Exploration and genetic diversity analysis of 8 local wild flax germplasm resources[J]. Molecular Plant Breeding, 17(11): 3755-3760)
[16] 王瑞, 陈雪, 郭青青, 等. 2022. 甘蓝型油菜白花基InDel连锁标记开发[J]. 作物学报, 48(03): 759-769. (Wang R,Chen X, Guo Q Q, et al. 2022. Development of linkage InDel markers of the white petal gene based on wholegenome re-sequencing data in Brassica napus L.[J]. Acta Agronomica Sinica, 48(3): 759-769.)
[17] 吴建忠, 黄文功, 康庆华, 等. 2013. 亚麻遗传连锁图谱的构建[J]. 作物学报, 39(6): 1134-1139. (Wu J Z, Huang W G, Kang Q H, et al. 2013. Construction of flax genetic linkage map[J]. Acta Agronomica Sinica, 39(6): 1134-1139.)
[18] 徐婷婷, 汪巧玲, 邹淑琼, 等. 2020. 基于高通量测序的大麦InDel标记开发及应用[J]. 作物学报, 46(9): 1340-1355.(Xu T T, Wang Q L, Zou S Q, et al. 2020. Development and application of InDel markers based on high throughput sequencing in barley[J]. Acta Agronomica Sinica, 46(9): 1340-1350.)
[19] 赵春芳, 赵庆勇, 吕远大, 等. 2022. 半糯粳稻品种核心标记的筛选及DNA指纹图谱的构建[J]. 中国农业科学, 55(23): 4567-4582. (Zhao C F, Zhao Q Y, Lv Y D, et al.2022. Screening of core markers and construction of DNA fingerprints of semi-waxy japonica rice varieties [J]. Scientia Agricultura Sinica, 55(23): 4567-4582.)
[20] Fu Y B. 2005. Geographic patterns of RAPD variation in cultivated Flax[J]. Crop Science, 45(3): 1084-1091.
[21] Hoque A, Fiedler J D, Rahman M. 2020. Genetic diversity analysis of a flax (Linumusit atissimum) global collection[J]. BMC Genomics, 21: 557.
[22] Soto-Cerda B J, Diederichsen A, Ragupathy R,et al.2013.Genetic characterization of a core collection of flax (Linum usitatissimum L.) suitable for association mapping studies and evidence of divergent selection between fiber and linseed types[J]. BMC Plant Biology, 13: 78.
[23] Soto-Cerda B J, Maureira-ButlerI, Muñoz G, et al. 2012. SSRbased population structure, molecular diversity and linkage disequilibrium analysis of a collection of flax (Linum usitatissimum L.) varying for mucilage seed-coatcontent[J]. Mol Breeding, 30:875-888.
[24] Zhang J P, LongY, Wang L M, et al. 2018. Consensus genetic linkage map construction and QTL mapping for plant height-related traits in linseed flax (Linum usitatissimum L.)[J]. BMC Plant Biology, 18(1): 160..
文章摘自:王斌,刘杰,侯静静,等.基于重测序的亚麻InDel标记开发及亚麻育成品种遗传多样性和群体结构分析[J].农业生物技术学报,2026,34(01):78-87.