摘 要:胡麻是我国干旱地区的主要油料作物,其中甘肃是我国胡麻主要产区之一。研究甘肃胡麻地方品种的遗传多样性和群体结构,对于挖掘丰富优异等位变异、提高胡麻育种效率具有重要意义。利用SSR分子标记对甘肃省13个县(市、区)的94份胡麻地方品种资源进行遗传多样性和群体结构分析。结果表明,21对SSR引物共扩增出46个等位基因,每对引物有2~3个等位基因;多态信息含量为0.158~0.522,平均为0.343;观测杂合度为0.060~0.957,平均0.448;期望杂合度为0.126~0.569,平均0.408;香农多样性指数为0.267~0.860,平均0.613;以遗传距离0.028为阈值,94份胡麻地方品种被聚为4个类群。由此表明,甘肃胡麻地方品种间遗传差异较小、遗传距离相近、遗传背景狭窄。
关键词:胡麻;地方品种;遗传多样性;群体结构
胡麻产业面临着种植面积连续萎缩、生产成本高、单产低、对国际市场依赖度高的现实困境,但胡麻在油料作物中的地域优势、文化优势、品质优势具有重要且无法替代的价值[1]。因此进一步提高胡麻的产量及品质、提升其种质资源的丰富度是首要选择。高丰富度的种质资源是作物育种与开发的重要基础[2-3]。目前种质资源的遗传多样性分析主要是在农艺性状和分子标记水平进行研究。由于农艺性状易受环境及生产栽培的影响,且鉴定周期较长,植物种质的遗传多样性研究及鉴定多采用分子标记法,主要包括ISSR(inter-simple sequence repeat)[4]、SSR(simple sequence repeats)[5-6]、RAPD(random amplified polymorphic DNA)[7-8]、SRAP(sequence-related amplified polymorphism)[9-10]、AFLP(amplified fragment length polymorphism)[11]、SNP(single nucleotide polymorphism)[12]等。其中,SSR以其遗传稳定性好、重复性高、易于分析等优点,在植物种质资源的群体结构分析及遗传多样性评估中广泛应用[13]。
邓欣[14]对535份亚麻材料表型性状和遗传多样性进行分析,将其划分为油用亚麻和纤用亚麻2个类群,并构建了亚麻种质的初级核心种质库。Cloutier等[15]对23份亚麻种质资源进行遗传多样性分析发现,这23份品种间遗传背景较狭窄。Wu等[16]利用62对SSR引物将48份亚麻品种分为纤用亚麻和油用亚麻2大类群,且同一类群中的种质遗传相似性较高。
地方品种作为现代育种品种重要的基础资源,对当地的地理与气候环境具有良好的适应性,兼具有抗逆性强、遗传变异丰富等特点,可用于拓宽现代育种品种的遗传背景,在种质改良和创新上具有重要价值[17]。甘肃是我国胡麻的主要产区之一,境内胡麻地方品种繁多,种质资源丰富。但地方资源存在良种互相引种的现象,且在引种过程中经常会出现“异种同名”和“异名同种”等问题,严重影响了地方资源的保护和利用。因此,开展地方资源遗传多样性研究非常重要。
本研究基于SSR分子标记对甘肃13个县(市、区)的94份胡麻地方品种资源进行遗传多样性和群体结构分析,以揭示甘肃胡麻地方种质资源的群体结构与遗传多样性,为进一步研究甘肃胡麻地方种质资源及其开发利用提供参考。
1 材料与方法
1.1试验材料
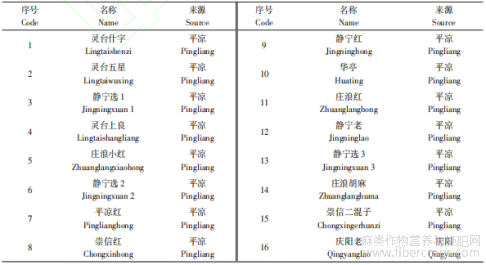
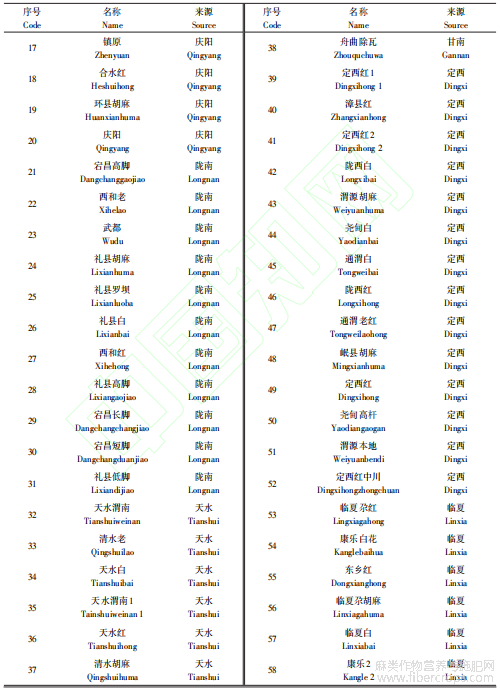
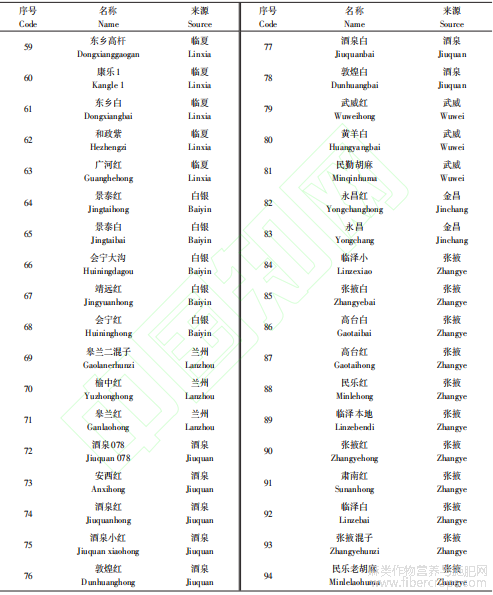
选取甘肃13个县(市、区)的94份胡麻地方品种资源为试验材料,其中平凉16份、定西14份、陇南11份、张掖11份、临夏10份、酒泉7份、天水6份、庆阳5份、白银5份、武威3份、兰州3份、金昌2份、甘南1份(表1),均由甘肃省农业科学院作物研究所提供。
表1 94份甘肃地方种质信息
按照甘肃省农作物种植区划和各种植区域的生态条件、品种特性、气候类型、生产实际及耕作制度等,将甘肃省划分为陇南(LN)、陇东(LD)、中部(ZB)和河西(HX)4个地区,其中陇东地区包括庆阳和平凉;陇南地区包括甘南、天水和陇南;中部地区包括定西、兰州、白银和临夏;河西走廊包括酒泉、嘉峪关、武威、张掖和金昌。试验材料来源也按此标准进行分类。
1.2试验方法
1.2.1 DNA提取与检测
每份种质随机选取50粒种子室温条件下于培养皿发芽,2周后采集幼苗叶片,用改良版CTAB法[18]提取基因组DNA。使用1%琼脂糖凝胶电泳和超微量分光光度计检测DNA质量,并调节至50 ng·μL−1,置于−20℃冰箱保存备用。
1.2.2 SSR引物筛选
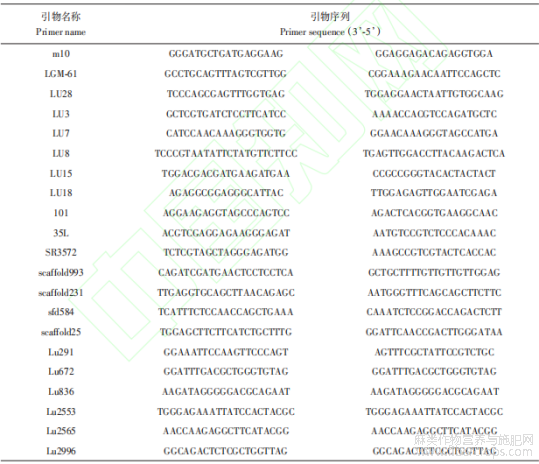
课题组前期从国内外文献查找亚麻SSR引物1 015对;利用EST序列(美国NCBI数据库下载)设计引物282对;利用亚麻基因组序列(https://www.ncbi.nlm.nih.gov/genome/?term=flax)设计SSR引物29对;共计1 326对。通过生物信息筛选,最后筛选得到282对引物进行序列合成。利用6大胡麻主产区审定的8个胡麻品种对282对合成的引物进行初步筛选,得到多态性引物54对;然后利用来自胡麻主生态区的58个胡麻品种对筛选的引物进行评价,得到扩增条带清晰、稳定性和重复性好的引物32对。进一步挑选6份不同来源的甘肃胡麻地方种质对筛选的32对引物进行验证,挑选出21对引物(表2)。其中7对引物来源于Deng等[19];4对来源于Saha等[20];3对来源于Cloutier等[15];3对来源于Bickel等[21],1对来源于Roose-Amsaleg等[22];2对来源于Soto-Cerda等[23-24];1对为本课题组前期开发所得。所有引物由北京擎科生物科技有限公司合成。
表1 94份甘肃地方种质信息
1.3 PCR体系与程序
PCR体系共10μL,包括DNA模板2μL,正、反向引物各1μL,PCR Mix 5μL,ddH2O 1μL。PCR扩增程序:94℃预变性5min,94℃变性30 s,65℃复性45 s,72℃延伸30 s,12个循环;94℃变性30 s,55℃复性45 s,72℃延伸30 s,24个循环;72℃延伸10 min,4℃保存。PCR扩增产物用8%聚丙烯酰胺凝胶电泳(200 V,1.5 h)检测,银染显色后进行统计。
1.4数据分析
采用人工读取条带的方法统计扩增结果,将清晰条带记为“1”,同一位置无条带记为“0”,编辑为(0,1)矩阵。利用DataFormater[25]实现数据格式转换,利用PopGen32软件计算有效等位基因数(Ne)、观测等位基因数(Na)、期望杂合度(He)、观测杂合度(Ho)、Shannon多样性指数(I)、基因流(Nm)、遗传分化系数(Fst)等指标,利用PowerMarker 3.25计算引物多态信息含量(polymorphism information content,PIC),用MEGA11软件基于非加权成对算术平均法(unweighted pair-group method with arithmetic means,UPGMA)进行聚类分析,用iTOL在线网站对聚类图进行美化。应用Structure 2.3.4[26]软件对94份胡麻种质进行群体结构分析。
2结果与分析
2.1 SSR多态性位点分析
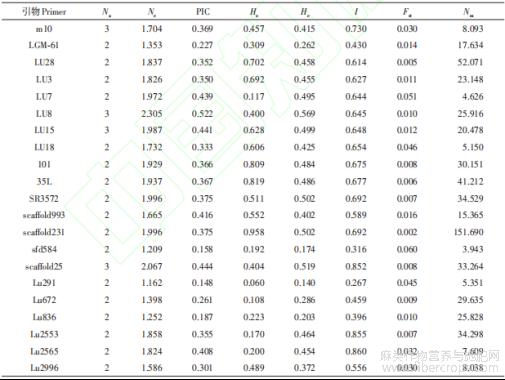
由表3可知,94份胡麻种质共检测出46个等位变异,每对引物2~3个等位变异,平均2.191个等位变异。Ne表示群体遗传变异大小,21对SSR引物的Ne为1.162~2.305,平均1.743。PIC是引物多态性指标,当0.25<PIC≤0.50,表示该位点为中度多态性位点[27]。21对引物的PIC为0.158~0.522,平均0.343,表明这21对SSR引物为中度多态性标记,可以较好地分析胡麻种质的遗传多样性。
表3 21对SSR引物的遗传多样性参数
注:Ne —有效等位基因数;Na —观测等位基因数;He —期望杂合度;Ho —观测杂合度;I— Shannon多样性指数;Nm—基因流;Fst—遗传分化系数。
2.2群体遗传多样性分析
He是衡量群体的遗传多样性高低的指标,He越高,表示群体的遗传一致性越低,其遗传多样性越丰富。当Ho>He时,说明位点的杂合子较多[28-29]。由表3可知,21对引物的Ho为0.060~0.957,平均0.448;He为0.126~0.569,平均0.408,Ho平均值大于He平均值,在21对引物中有14对引物的Ho>He,说明这些位点杂合子较多。I反映遗传多样性程度,I越大,代表群体的离散度越高,遗传多样性越丰富。21对引物的I为0.267~0.860,平均0.613,进一步说明94份胡麻地方种质的遗传多样性较低。
Fst代表不同群体间的遗传分化水平,Fst取值为0~1,当Fst<0.05时,表示不同群体间的遗传分化水平较低[30]。94个供试样品的Fst平均为0.020,说明94份胡麻地方品种种质的遗传分化水平较低。
Nm是指生物个体从其发生地分散出去而导致不同种群之间基因交流的过程,可发生在同种或不同种生物种群之间[31]。基因的交流可以降低种群之间的遗传差异,是影响种群遗传结构的重要因素。当Nm>1时,表明基因流可防止遗传漂变导致的种群遗传分化。94份胡麻种质的Nm平均值为27.525,说明94份胡麻地方种质间的基因交流水平较高,可以防止遗传漂变导致的种群遗传分化。
2.3聚类分析
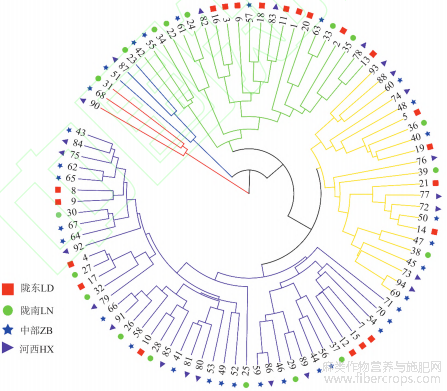
利用21对SSR标记基于UPGMA法对94份胡麻地方种质进行聚类分析,结果(图1)表明,94份种质间的遗传距离为0.042~0.822,平均0.283。其中,礼县罗坝(甘肃陇南)与渭源本地(甘肃定西)间遗传距离最远,为0.822;灵台上良(甘肃平凉)与镇原(甘肃庆阳)及灵台上良(甘肃平凉)与西和红(甘肃陇南)间遗传距离最近,均为0.042。
注:种质编号同表1。
图1 94份胡麻地方种质的聚类分析
在遗传距离为0.028处,94份胡麻地方种质被划分4个类群,其中,类群1有3份种质;类群2有3份种质;类群3有22份种质;类群4有66份种质。在遗传距离0.035处,类群4又可划分为2个亚群。从种质来源分析,来自陇东和陇南的胡麻种质主要分布在类群3和类群4的第2亚群,说明陇东和陇南的种质亲缘关系较近;来自中部地区和河西走廊的种质主要分布在类群4,说明中部地区和河西走廊的种质间亲缘关系较近。从品种表型分析,80%的白籽胡麻种质分布在类群4;高杆的胡麻种质中除宕昌高脚和尧甸高杆分布在类群4的第1亚群,其余均分布在类群4的第2亚群。
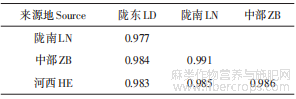
由表4可知,不同地区群体间的遗传相似性系数为0.977~0.991,平均0.984,遗传距离为0.009~0.023,平均0.016。这说明甘肃省内不同地方胡麻地方品种的遗传差异较小,遗传背景狭窄。
表4 不同地区种质间的相似性系数
2.4群体结构划分
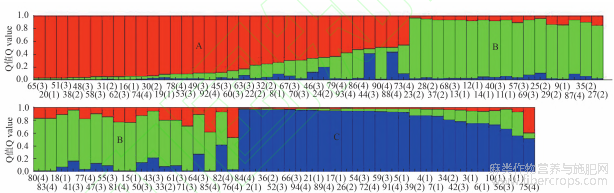
由图2可知,94份胡麻地方种质可划分为3个群体,群体A包括33份种质,其中陇东4份、陇南6份、中部14份、河西9份;群体B包括35份种质,其中陇东7份、陇南8份、中部12份、河西8份;群体C包括26份种质,其中陇东10份、陇南4份、中部6份、河西6份。来自陇东的种质主要分别在群体B和群体C中;来自陇南、中部和河西的种质主要分别在群体A和群体B中。当Q<0.6时,认为种质具有混合来源[32]。94份种质中有12份种质属于混合来源。且94份种质均有不同程度的基因交流,表明甘肃省内不同地区的地方种质间遗传距离相近,遗传背景狭窄。
注:小括号外的序号为表1中94份种质序号;括号内的1~4分别表示陇东、陇南、中部、河西。
图2 94份胡麻地方种质的群体结构
3讨论
物种的遗传多样性分析是其评价种质资源和利用杂种优势的基础。Cloutier等[15]利用275个EST-SSRs标记检测到635个等位基因,PIC为0.08~0.92,平均0.35。本研究表明,21对SSR引物的PIC为0.158~0.522,平均0.343,表明这21对SSR标记为中度多态性标记,可较好地用于遗传多样性分析。高凤云等[5]筛选出的30对SSR引物,在230份胡麻种质中共扩增出365个等位基因,PIC为0.248 9~0.625 7,平均0.432 2,与本研究一样,均属于中多态性标记。
伊六喜等[13]基于SRAP标记对161个胡麻种质资源进行了遗传多样性分析发现,来源地相同的胡麻品种多聚成一类,与本研究相似一致;且161份胡麻种质的I为0.521,与本研究的0.613相近,表明同一地域内的胡麻种质遗传多样性较低,遗传背景较窄。
UPGMA聚类分析显示,在遗传距离0.028处,94份胡麻地方种质被划分为4个类群,从地理距离来看,距离较近的陇东与陇南种质被聚为同一类群,河西与中部地区的种质聚为同一类群;从品种表型来看,80%的白籽胡麻种质被划分在类群4中;高杆的胡麻种质中除了宕昌高脚和尧甸高杆分布在类群4的第1亚群中,其余高杆种质均分布在类群4的第2亚群中。
随着育种目标的趋同,人工选择优良性状,导致地方品种的遗传基础日益狭窄[33]。本研究结果表明,甘肃省胡麻地方品种的遗传差异较小、遗传距离相近、遗传背景狭窄,这可能与省内各市区间相互引种有关。胡麻是自花授粉植物,因此建议在选育胡麻新品种时,采用人工授粉的方法进行杂交,以增加胡麻遗传变异,同时可选择遗传背景差异较大的种质进行杂交配组,以丰富甘肃省胡麻地方品种的遗传背景,提高遗传多样性。同时在亲本选配可通过分子标记,挖掘亲缘关系相对较远的核心种质,从而有利于选育出遗传多样性丰富的胡麻新品种。
参考文献
[1]马慧敏,任红燕,张振宇,等.山西胡麻产业高质量发展对策研究[J].山西农经,2024(5):152-156.
[2]崔翠,周清元,王利鹃,等.亚麻种质主要农艺性状主成分分析与综合评价[J].西南大学学报(自然科学版),2016,38(12):10-18.CUI C,ZHOU Q Y,WANG L J,et al..The cluster analysis and comprehensive evaluation of flax germplasm based on principal components of agronomic traits[J].J.Southwest Univ.(Nat.Sci.),2016,38(12):10-18.
[3]张丽丽,刘晶晶,乔海明,等.从俄罗斯引进亚麻种质资源的农艺性状评价[J].中国油料作物学报,2017,39(5):698-703.ZHANG L L,LIU J J,QIAO H M,et al..Evaluation of flax germplasm introduced from Russia by agronomic traits[J].Chin.J.Oil Crop Sci.,2017,39(5):698-703.
[4]HAO D M,QIU C S,LONG S H,et al..An overview on molecular biology of flax(Linum usitatissimum L.)in China[J].Agric.Sci.Technol.,2016,17(8):1767-1772.
[5]高凤云,斯钦巴特尔,周宇,等.基于SSR标记的胡麻粗脂肪及脂肪酸组分的关联分析[J].作物杂志,2022(1):44-49.GAO F Y,Siqinbateer,ZHOU Y,et al..Association analysis of crude fat and fatty acid components in flax based on SSR markers[J].Crops,2022(1):44-49.
[6]潘慧云.亚麻表型性状与SSR标记的关联分析[D].呼和浩特:内蒙古大学,2019.PAN H Y.Association analvsis of flax phenotypic traits using SSR markers[D].Hohhot:Inner Mongolia University,2019.
[7]何东锋.亚麻RAPD和微卫星标记开发与遗传多样性分析[D].长沙:湖南农业大学,2009.HE D F.Flax RAPD and microsatellite marker development and genetic diversity analysis[D].Changsha:Hunan Agricultural University,2009.
[8]李秋芝,宋鑫玲,曹洪勋,等.100份亚麻种质资源遗传多样性及亲缘关系的RAPD分析[J].现代农业科技,2015(24):65-67,71.LIQ Z,SONG X L,CAO H X,et al..Genetic diversities and phylogenetic relationships of 100 flax germplasm resources based on RAPD[J].Mod.Agric.Sci.Technol.,2015(24):6567,71.
[9]王斌.亚麻EST-SSR和SRAP标记研究[D].兰州:甘肃农业大学,2010.WANG B.Studies on EST-SSR and SRAP markers in flax[D].Lanzhou:Gansu Agricultural University,2010.
[10]李闻娟,齐燕妮,王利民,等.运用SRAP分子标记对胡麻杂交种纯度的鉴定研究[J].甘肃农业科技,2019(9):59-62.
[11]李丹丹.部分胡麻种质资源主要农艺性状和AFLP分子标记的遗传多样性分析[D].呼和浩特:内蒙古农业大学,2015.LID D.Genetic diversity of agronomic traits and aflp marker of flax germplasm[D].Hohhot:Inner Mongolia Agricultural University,2015.
[12]SINGH N,AGARWAL N,YADAV H K.Genome-wide SNPbased diversity analysis and association mapping in linseed(Linum usitatissimum L.)[J/OL].Euphytica,2019,215(8):139[2024-02-10].https://doi.org/10.1007/s10681-019-2462-x.
[13]伊六喜,斯钦巴特尔,高凤云,等.内蒙古胡麻地方品种资源遗传多样性分析[J].作物杂志,2018,34(6):53-57.YI L X,Siqinbateer,GAO F Y,et al..Genetic diversity of flax germplasm resources in Inner Mongolia[J].Crops,2018,34(6):53-57.
[14]邓欣.亚麻分子标记的开发及产量相关性状的关联分析[D].北京:中国农业科学院,2013.DENG X.Development of molecular markers and association analysis of yield related traits in flax(Linum usitatissimum L.)[D].Beijing:Chinese Academy of Agricultural Sciences,2013.
[15]CLOUTIER S,NIU Z,DATLA R,et al..Development and analysis of EST-SSRs for flax(Linum usitatissimum L.)[J].Theor.Appl.Genet.,2009,119(1):53-63.
[16]WU J,ZHAO Q,WU G,et al..Development of novel SSR markers for flax(Linum usitatissimum L.)using reducedrepresentation genome sequencing[J].Front.Plant Sci.,2017,7(7):22-30.
[17]李清超,张登峰,李春辉,等.西南地区玉米地方种质资源遗传多样性分析及综合评价[J].作物杂志,2024(4):24-32.LIQ C,ZHANG D F,LI C H,et al..Genetic diversity analysis and comprehensive evaluation of maize landraces in southwest China[J].Crops,2024(4):24-32.
[18]李荣华,夏岩石,刘顺枝,等.改进的CTAB提取植物DNA方法[J].实验室研究与探索,2009,28(9):14-16.LIR H,XIA Y S,LIU S Z,et al..CTAB-improved method of DNA extraction in plant[J].Res.Explor.Lab.,2009,28(9):14-16.
[19]DENG X,LONG S H,HE D F,et al..Isolation and characterization of polymorphic microsatellite markers from flax(Linum usitatissimum L.)[J].African J.Biotechnol.,2011,10:734-739.
[20]SAHA D,RANA R S,DAS S,et al..Genome-wide regulatory gene-derived SSRs reveal genetic differentiation and population structure in fiber flax genotypes[J].J.Appl.Genet.,2019,60(1):13-25.
[21]BICKEL C L,GADANI S,LUKACS M,et al..SSR markers developed for genetic mapping in flax(Linum usitatissimum L.)[J].Res.Reports Biol.,2011,2:23-29.
[22]ROOSE-AMSALEG C,CARIOU-PHAM E,VAUTRIN D,et al..Polymorphic microsatellite loci in Linum usitatissimum[J].Mol.Ecol.Notes,2006,6(3):796-799.
[23]SOTO-CERDA B J,CARRASCO R A,ARAVENA G A,et al..Identifying novel polymorphic microsatellites from cultivated flax(Linum usitatissimum L.)following data mining[J].Plant Mol.Biol.Report.,2011,29(3):753-759.
[24]SOTO-CERDA B J,CLOUTIER S.Outlier loci and selection signatures of simple sequence repeats(SSRs)in flax(Linum usitatissimum L.)[J].Plant Mol.Biol.Rep.,2013,31(4):978-990.
[25]樊文强,盖红梅,孙鑫,等.SSR数据格式转换软件DataFormater[J].分子植物育种,2016,14(1):265-270.FAN W Q,GAI H M,SUN X,et al..DataFormater,a software for SSR data formatting to develop population genetics analysis[J].Mol.Plant Breeding,2016,14(1):265-270.
[26]YAN W,KARIKARI B,CHANG F,et al..Genome-wide association study to map genomic regions related to the initiation time of four growth stage traits in soybean[J/OL].Front.Genet.,2021,12:715529[2024-02-10].https://doi.org/10.3389/fgene.2021.715529.
[27]HIETER P A,HOLLIS G F,KORSMEYER S J,et al..Clustered arrangement of immunoglobulin lambda constant region genes in man[J].Nature,1981,294(5841):536-540.
[28]汤存伟,余雄,刘武军,等.新疆13个绵羊群体遗传多样性及遗传分化的研究[J].家畜生态学报,2011,32(1):13-19.TANG C W,YU X,LIU W J,et al..Stndy on genetic diversity and genetic differentiation of 13 sheep populations in Xinjiang uygur autonomous region[J].J.Domest.Anim.Ecol.,2011,32(1):13-19.
[29]WRIGHT S.Variability within and among Natural Populations[M].Chicago:University of Chicago Press,1978:1-256.
[30]黄燕,朱振东,段灿星,等.灰葡萄孢蚕豆分离物的遗传多样性[J].中国农业科学,2014,47(12):2335-2347.HUANG Y,ZHU Z D,DUAN C X,et al..Genetic diversity of Botrytis cinerea isolates from broad bean[J].Sci.Agric.Sin.,2014,47(12):2335-2347.
[31]穆小婷,廖侦成,凌彩金,等.基于SSR标记的55份清远野生茶树种质资源遗传多样性和亲缘关系分析[J].中国茶叶,2023,45(2):36-43.MU X T,LIAO Z C,LING C J,et al..Genetic diversity and relationship analysis of 55 wild tea germplasm from Qingyuan based on SSR markers[J].China Tea,2023,45(2):36-43.
[32]罗凯,卢会翔,吴正丹,等.中国西南地区甘薯主要育种亲本的遗传多样性及群体结构分析[J].中国农业科学,2016,49(3):593-608.LUO K,LU H X,WU Z D,et al..Genetic diversity and population structure analysis of main sweet potato breeding parents in southwest China[J].Sci.Agric.Sin.,2016,49(3):593-608.
[33]王欣,李强,曹清河,等.中国甘薯产业和种业发展现状与未来展望[J].中国农业科学,2021,54(3):483-492.WANG X,LI Q,CAO Q H,et al..Current status and future prospective of sweetpotato production and seed industry in China[J].Sci.Agric.Sin.,2021,54(3):483-492.
文章摘自:刘杰,王斌,侯静静,等.甘肃胡麻地方品种遗传多样性和群体结构分析[J/OL].中国农业科技导报(中英文),1-10[2025-08-19].https://doi.org/10.13304/j.nykjdb.2024.0222.