摘 要:探究适用于对苎麻(Boehmeria nivea)进行低温脱胶的酶可以节约能源,降低对环境的影响。本研究从土壤中筛选出果胶酸裂解酶(Pectate lyase,Pel)产生菌,并通过基因克隆表达获得该菌的重组Pel,测定重组Pel的酶学性质后将其应用于苎麻的低温脱胶中。结果表明,从土壤中获得一株短小芽孢杆菌Bacillus pumilus B.15-1,该菌株在液体培养时可产酶0.12U/mL。将其Pel基因克隆并在大肠杆菌中表达,用Ni+-亲和层析纯化获得分子量为42.56kDa的重组酶。该酶的最适作用pH为8.5,在pH8.5和30℃孵育24h后酶活力无损失;最适作用温度为55℃,在30℃较稳定。rBpumPel-Ec可在30℃对苎麻进行单独脱胶,当用酶量为25U/mL时得苎麻失重率为18.65%。rBpumPel-Ec亦能通过与NaOH联用的方式提升碱法脱胶的效果,当将25U/mL rBpumPel-Ec与0.5%NaOH联用时,苎麻的失重率(30.20%)高于单独使用0.5%NaOH时所得的失重率(23.54%)。本研究结果表明rBpumPel-Ec在苎麻的低温脱胶方面具有潜在应用价值。
关键词:果胶酸裂解酶;基因克隆表达;酶学性质;短小芽孢杆菌;苎麻脱胶;清洁生产
苎麻(Boehmeria nivea)是一种苎麻属(Boehmeria)的多年生植物,其主要被种植于热带、亚热带和部分温带国家或地区[1]。中国每年生产50万吨纤维,该产量占世界总产量的97%,其中苎麻产量最大,又被称为“中国草”[2]。在所有植物中,苎麻韧皮部所含纤维的长度最长,韧性最高,因此苎麻韧皮纤维被广泛用于纺织和服装等行业中[3]。苎麻韧皮部的成分复杂,以此制备韧皮纤维的工艺依次为“剥皮”(从植株上分离获得韧皮部)[4]、“刮麻”(亦称“刮青”或“刮背”,即把青色的表皮刮掉)[5]和“脱胶”(将果胶、木聚糖、甘露聚糖、阿拉伯聚糖、蛋白质、脂肪和灰分等去除),其中“脱胶”步骤决定了制备所得韧皮纤维的质量,因此该步骤对苎麻的应用有重要意义。
传统脱胶方法为化学脱胶,即采取化学物进行脱胶,最常用的做法为用NaOH在高于100℃进行脱胶[6],然而所获效果不够理想。有研究者尝试使用其他化学物,例如Lin等[7]使用共晶溶剂(deep eutectic solvent)进行脱胶,发现在160℃煮沸2.5h能获得最佳效果;Qu等[8]分别使用甘油、乙二醇、1,2-丙二醇和1,4-丁二醇进行脱胶,发现使用甘油在200℃处理1h能获得最佳效果,然而由于采用的温度高,导致能耗高,因此工业上仍然采用NaOH进行脱胶。
生物脱胶包括微生物脱胶和酶法脱胶,其中微生物脱胶是将“刮麻”后的苎麻韧皮部作为微生物生长的基质,使用微生物在生长过程中产的果胶酶和半纤维素酶进行脱胶。此法可在较低温度(30-35℃)进行微生物的培养和产酶,但此法往往需要较长时间,致使制作工艺过长[9]。例如Shu等[10]采集107份土壤、田间腐烂苎麻和脱胶池腐烂物等类型样品,从中筛选获得一株能高效脱胶的细菌HG-49,但该菌的脱胶过程需要16h。Yang等[11]从环境中筛选获得5株菌株 Dickeya dadantii DCE-01、Bacillus subtilis 1101、Bacillus sp.B6、B. cereus B7和B. cereus B8,将它们两两组合进行共培养后发现 Dickeya dadantii DCE01+B.cereus B7的共培养组合所获的粗酶用于脱胶可获得最佳效果,但脱胶时间长达12h。
酶法脱胶是将微生物的生长产酶与脱胶分开,直接使用酶进行脱胶,其优点是耗时短于微生物脱胶[12]。酶法脱胶所用的果胶酶以内切果胶酸裂解酶(endo-Pectate Lyase,EC:4.2.2.2)最为常见,该酶通过β-消除反应随机裂解果胶主链的α-(1,4)-糖苷键,比外切果胶酸裂解酶(exo-Pectate Lyase,EC:4.2.2.9,通过β-消除反应裂解果胶主链末端的α-(1,4)-糖苷键)能更快实现对果胶的解聚合[13]。虽然该法用时比微生物脱胶短,但所用温度高于微生物脱胶所用温度,致使能耗稍高。例如Zhao等[14]采用来自 Paenibacillus polymyxaKF-1的酶PpPel10a进行脱胶,脱胶时间为1h,脱胶温度为50℃;Zhen等[15]采用来自Paenibacillus sp.0602的酶pelNK93I进行脱胶,脱胶时间为1h,脱胶温度为60℃;Xu等[16]采用来自Dickeya dadantiiDCE-01的酶PelG403进行脱胶,脱胶时间为2h,脱胶温度为50℃;Xu等[17]采用来自Dickeya dadantiiDCE-01的酶PelG419经蛋白质工程获得的突变体进行脱胶,脱胶时间为2h,脱胶温度为55℃、Liu等[18]采用来自Bacillus sp.B58-2的酶rBvelPL1-Ec进行脱胶,脱胶时间为4h,脱胶温度为50℃;Zheng等[19]采用来自Bacillus sp.RN1的酶BspPel进行脱胶,脱胶时间为4h,脱胶温度为50℃。
目前关于将果胶酸裂解酶用于苎麻的酶法低温脱胶的研究较少。因此,本研究拟通过基因克隆表达的方法获得来自土壤微生物的果胶酸裂解酶,将其进行酶学性质测定后应用于苎麻韧皮部的低温脱胶,并评估该酶单独使用及与NaOH联用所得效果,以期为该酶的工业化应用研究提供理论指导。
1 材料与方法
1.1 材料
1.1.1 样品处理
将新鲜桔子皮置于50℃的烘箱中干燥至恒重,再使用小型植物组织粉碎机粉碎,使用60目分样筛将粉碎物分样后,所获粉末用于制作培养基。苎麻韧皮部产自江西宜春市上高县,将苎麻韧皮部“刮青”后日晒所获干品再于50℃烘箱中干燥至恒重,备用。
1.1.2 药品和试剂
聚半乳糖醛酸购买自上海阿拉丁生化科技股份有限公司,所有化学药品均为分析纯。使用天根生化科技(北京)有限公司生产的基因组DNA提取试剂盒进行细菌基因组DNA的提取,使用南京诺唯赞生物科技股份有限公司所产的2×Phanta Max Master Mix试剂扩增细菌16SRNA的部分编码区序列。
1.1.3 菌株
细菌B.15-1为本研究从土壤中筛选得到,大肠杆菌(Escherichia coli)菌株DH5α和Rossetta均存于广西科学院生物科学与技术研究所微生物与酶工程创新团队实验室。
1.1.4 主要仪器与设备
Infinite 2000酶标仪(瑞士Tecan公司),OSE-GP-01 PCR仪[天根生化科技(北京)有限公司],MIN QUAN MQD-B3R恒温摇床(上海旻泉仪器有限公司),Centrifuge5415D小型常温离心机(德国Eppendorf公司),HICLAVE高压灭菌锅(日本Saitama-ken公司),超净工作台(苏州净化设备有限公司),KS-1000ZDN超声波细胞粉碎机(昆山洁力美超声仪器有限公司)。
1.1.5 培养基
分离培养基的配方为(g/L):桔子皮粉10g,胰蛋白胨10g,酵母提取物5g,NaCl10g,pH7.0,琼脂粉20g。筛选培养基为不含琼脂粉的分离培养基。进行种属鉴定时所用菌体以不含杂质为佳,故所用培养基为不含桔子皮粉的筛选培养基。
1.2 微生物的分离筛选
土壤样品采自广西大学农场木薯地。将1g土壤样品置于装有10mL无菌水的指形瓶中,在200r/min震荡10min。将悬浊液用无菌水以10倍为梯度进行逐步稀释,后将稀释液涂布于分离培养基平板上,置于37℃培养2d。将平板上生长的菌落接种至筛选培养基中(250mL三角瓶装培养基50mL),置于37℃、200r/min的摇床中培养2d,然后将培养液在10000r/min离心1min,测定上清液中的果胶酸裂解酶活力。
1.3 种属鉴定
使用液体培养基对菌株进行培养,将所得培养物在10000r/min离心1min获得菌体。使用基因组DNA提取试剂盒提取DNA作为模板,使用通用引物(27F:5’-AGAGTTTGATCCTGGCTCAG-3’;1492R:5’-TACGACTTAACCCCAATCGC-3’)进行16SRNA部分编码区序列的扩增。所得扩增产物送上海生工生物工程技术服务有限公司进行测序,所得序列拼接后进行BLASTN(N为nucleotide)搜索。使用MEGA软件,取芽孢杆菌属内subtilis分枝(Clade)所含物种模式菌株的16SRNA部分编码区序列与待鉴定物种的序列进行发育建树,根据亲缘关系进行物种鉴定。
1.4 酶活力测定
将40μL适当稀释后的酶液与280μL含0.15%PGA的Tris-HCl缓冲液(0.2M,pH8.5)混合,50℃下孵育10min后加入640μL30mmol/L H3PO4溶液终止反应,在235nm处测量不饱和键来反映果胶酸裂解酶的活力。一个单位的酶活力定义为每分钟产生1µmol不饱和键所需的酶量,其中235nm处的分子消光系数为4600M-1cm-1。将酶液沸水浴5min灭活,其余操作同上作为对照。
1.5 基因克隆与表达
在公布于NCBI(National Center for Biotechnology Information,https://www.ncbi.nlm.nih.gov/)的短小芽孢杆菌菌株基因组中寻找被预测为果胶酸裂解酶(pectate lyase)的假定蛋白质(Putative protein),使用SMART在线软件(http://smart.embl-heidelberg.de/)预测该假定蛋白质的信号肽和催化功能域,以决定基因的克隆区域。从菌株B.15-1的基因组中扩增对应基因,将其与载体pET30a(+)进行双酶切后连接并转化至大肠杆菌DH5α。将构建成功的质粒pET30a(+)-BpumPel转化至大肠杆菌Rossetta中表达,诱导条件为20℃、100r/min、1mmol/LIPTG(异丙基-β-D-硫代半乳糖苷)/12h。
使用Clustal X和GENEDOC软件将克隆所得基因片段编码的氨基酸序列与芽孢杆菌属的家族1果胶酸裂解酶进行多序列比对,所用的酶分别为P147(登录号BAB40336)[20]、BacPelA(登录号KR819891)[21]、Bsp165PelA(登录号GU088530,已通过蛋白质工程确认活性中心内催化氨基酸)[22]、PelE(登录号AAA24844)[23]、BliPelA(登录号KX440958)[24]和BvelPL1(登录号OR233722)[18]。
1.6 蛋白质纯化和功能鉴定
诱导后所得细胞悬浮于吸附液(NaH2PO4 50mmol/L,NaCl 300mmol/L,咪唑10 mmol/L,pH 8.0),经超声波破碎后所得总蛋白质在10000r/min离心1min,所得上清液为总可溶性蛋白质。将上清液用0.45μm的水性滤膜去除杂质颗粒后,再与Ni+填料混匀并包裹在冰中,放置于50r/min的往复式摇床中吸附1h后装柱。依次使用含有10、35、60、100、200、300、400和500mmol/L咪唑的吸附液润洗Ni+柱,每个咪唑梯度均润洗3倍柱体积,所得洗脱液以SDS-PAGE电泳[25]的方式检查蛋白质纯度,并以考马斯亮蓝法(Bradford法)[26]测定蛋白质的量。
将纯化后的重组酶(0.1mg)与含有0.3%聚半乳糖醛酸(PGA)的Tris-HCl缓冲液(1mL,pH8.5)混合,在35℃分别孵育10min和24h,用Aminex HPX 87H柱(Bio Rad,USA)在235nm波长处对其催化产物进行分析,柱温为50℃,流动相为5mmol/L H2SO4,流速为1mL/min,标准品为不饱和半乳糖醛酸(u1)、不饱和双半乳糖醛酸(uG2)和不饱和三半乳糖醛酸(uG3)。
1.7 酶学性质
1.7.1 最适作用 pH 值
测定酶在不同pH值中的活力,其中pH4.0-7.0用0.1mol/L柠檬酸-Na2HPO4缓冲液,pH7.0-9.0用0.1mol/L Tris-HCl缓冲液,pH9.0-11.0用0.1mol/L甘氨酸-NaOH缓冲液。酶活力最高处为100%,其他酶活力计为相对值。
1.7.2 pH 稳定性
将酶置于0.1mol/L缓冲液中,在30℃孵育24h后测定其酶活力。以未孵育的酶液作为标准(酶活力100%),酶液孵育后的活力计为相对值。
1.7.3 最适作用温度
分别测定酶在30、35、40、45、50、55、60、65、70℃的活力,其中酶活力最高处为100%,其他酶活力计为相对值。
1.7.4 热稳定性
将酶置于30、35、40、45、50、55、60℃孵育15、30、45和60min后测定其酶活力。以未孵育的酶液作为标准(酶活力100%),酶液孵育后的活力计为相对值。
1.7.5 金属离子
在酶反应体系中分别添加0.5、1.0或2.5mmol/L金属盐(LiCl、NaCl、MgCl2、KCl、CoCl2、FeCl2和MnCl2)后测定酶活力,以不额外添加金属盐时测定所得的酶活力作为标准(酶活力100%),其余酶活力计为相对值。
1.8 苎麻脱胶
1.8.1 酶的用量对脱胶的影响
将1.00g苎麻韧皮部缠绕成直径为5.0cm的小圈(苎麻圈),按照固液比1:20浸入20mL不同酶浓度(0.0、0.5、1.0、2.5、5.0、10.0、15.0、20.0、25.0 U/mL)的Tris-HCl缓冲液中(pH8.5),在30℃孵育6h后用超纯水充分浸洗,沥干后置于100℃烘干至恒5重。
失重率(%)=(脱胶前的重量-脱胶后的重量)×100/脱胶前的重量[16]。
1.8.2 NaOH 浓度对脱胶的影响
将苎麻圈浸入20mL不同浓度的NaOH溶液(0.010%、0.020%、0.030%、0.040%、0.05%、0.075%、0.100%、0.250%、0.500%),在30℃孵育6h(或在105℃的灭菌锅中孵育2.5h[18])后用超纯水充分浸洗,沥干后于100℃烘干至恒重,计算失重率。
1.8.3 酶和 NaOH 联合脱胶
先用25U/mL rBpumPel-Ec(单独脱胶时失重率达到最高值所用的量)对苎麻圈进行脱胶,再将苎麻圈洗涤和烘干后使用0.5%NaOH(单独脱胶时失重率达到最高值所用的量)在105℃的灭菌锅中孵育2.5h,计算苎麻圈的失重率。
1.9 数据处理
每个实验数据点由3个平行数值得来,使用Excel软件计算平均值及其标准偏差,使用Excel 2016或Origin 2018软件作图,使用SPSS 27.0软件分析显著性差异。
2 结果与分析
2.1 菌株筛选和种属鉴定
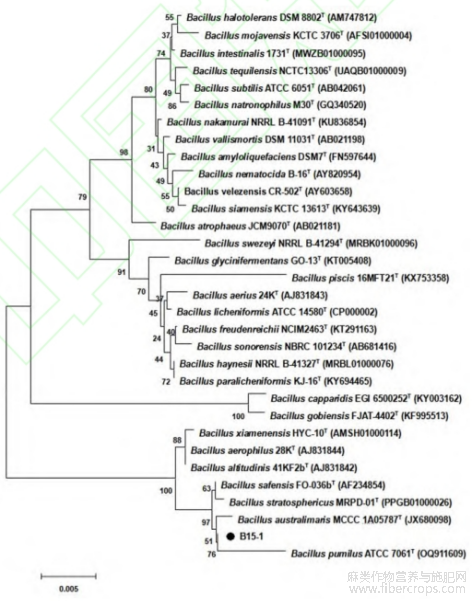
菌株B.15-1在使用筛选培养基培养时表现出果胶酸裂解酶活力(0.12U/mL),因此选择其进行进一步研究。在枯草杆菌属subtilis分枝所含物种中,短小芽孢杆菌的16S RNA序列与菌株B.15-1的相应序列拥有最近的亲缘关系,因此菌株B.15-1被鉴定为短小芽孢杆菌(图1)。
图1 B.15-1 与枯草杆菌属 subtilis 分枝代表性菌株 16S RNA 部分编码区序列的发育树
2.2 基因克隆与表达及蛋白质纯化
在短小芽孢杆菌3-19的基因组中找到一个被注释为假定pectate lyase的蛋白质,其登录号为QKN79634,将其氨基酸序列提交SMART进行预测后发现其第3-32位氨基酸构成信号肽,因此克隆其第33-347位氨基酸的编码基因序列。使用上游引物5’-CCCAAGCTTGGGAGGCAAAAGCGGCAG-3’(下划线处为HindⅢ识别位点)和下游引物5’-CCGCTCGAGAGGGTTTACCTTTCCAACACC-3’(下划线处为XhoⅠ识别位点)从B.15-1基因组中扩增获得基因BpumPel。通过多序列比对,发现基因BpumPel编码的蛋白质BpumPel的催化位点为位于保守区的Arg209和Arg214(图2)。
图2 BpumPel-Ec与已报道的家族1果胶酸裂解酶的多序列比对
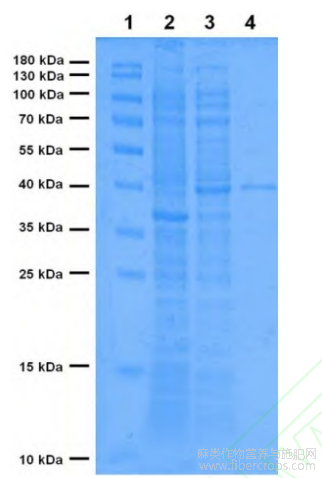
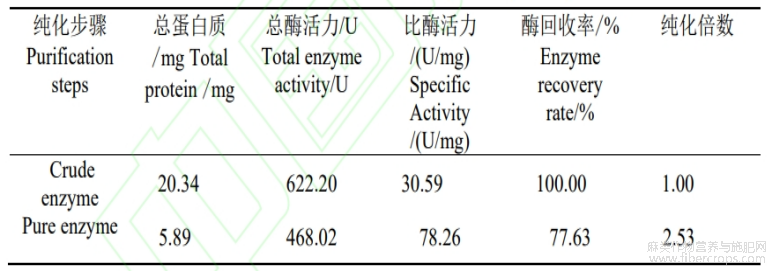
将扩增获得的基因克隆至载体pET30a(+),获得质粒pET30a(+)-BpumPel。将载体pET30a(+)和质粒pET30a(+)-BpumPel分别转化至大肠杆菌Rossetta并进行诱导表达,将细胞以超声波破碎后在10000r/min离心1min获得总可溶性蛋白质并进行SDS-PAGE电泳。由图3可见,相较于含有载体pET30a(+)的细胞(泳道2),含有质粒pET30a(+)-BpumPel的细胞可额外产生一条约40kDa的条带(泳道3),经过Ni+-亲和层析纯化后获得重组蛋白质rBpumPel-Ec(泳道4)。酶的回收率达77.63%,比酶活力为78.26U/mg(表1)。
图3 B.15-1 重组果胶酸裂解酶 rBpumPel-Ec 的纯化
Lane 1: molecular weight ladder; lane 2: total soluble protein of induced E. coli cell harboring pET30a(+); lane 3: total soluble protein of induced E. coli cell harboring pET30a(+)-BpumPel; lane 4: recombinant enzyme rBpumPel-Ec.
表1 B.15-1 重组果胶酸裂解酶 rBpumPel-Ec 的纯化
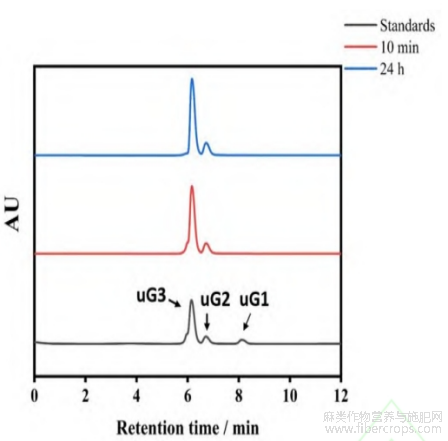
rBpumPel-Ec降解多聚半乳糖醛酸10min后的产物为双-不饱和半乳糖醛酸(uG2)和三-不饱和半乳糖醛酸(uG3),将降解时间延长至24h后亦无其他新产物(图4),因此表明双-和三-不饱和半乳糖醛酸为降解终产物。
图4 rBpumPel-Ec 降解产物分析
2.3 rBpumPel-Ec 的酶学性质
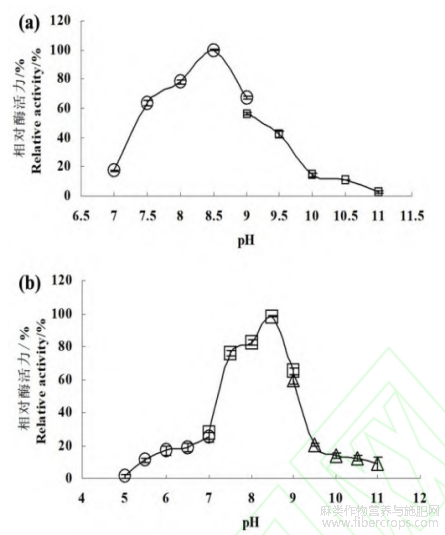
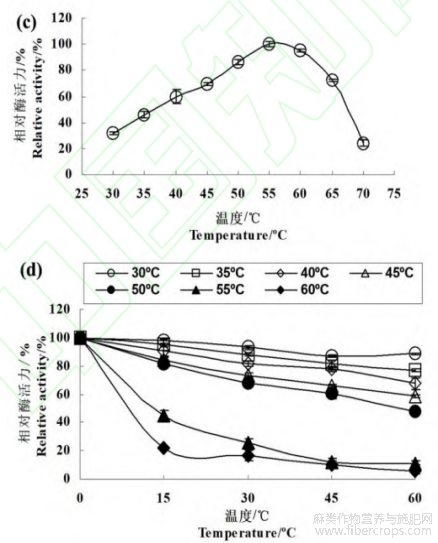
rBpumPel-Ec在酸性环境中酶活力低,其酶活力随着pH值的升高而升高,至pH值8.5时达到最高,因此最适作用pH值为8.5[图5(a)]。在30℃孵育24h后,置于pH8.5中的rBpumPel-Ec没有损失酶活力,置于其他pH中后均有不同程度的损失,因此rBpumPel-Ec在pH8.5时稳定[图5(b)]。在不同温度下,rBpumPel-Ec的酶活力随温度升高而升高,至55℃达到顶峰,因此其最适作用温度为55℃[图5(c)]。rBpumPel-Ec在30℃孵育1h后酶活力剩余88.55%,其稳定性随着温度升高而变差,其中在60℃孵育1h后酶活力剩余22.05%,延长孵育时间至1h后酶活力损失殆尽[图5(d)]。
图5 pH 值和温度对 rBpumPel-Ec 酶活力和稳定性的影响
(a) Effect of pH on the enzyme activity of rBpumPel-Ec; (b) Effect of pH on the stability of rBpumPel-Ec; (c) Effect of temperature on the enzyme activity of rBpumPel-Ec; (d) Effect of temperature on the stability of rBpumPel-Ec.
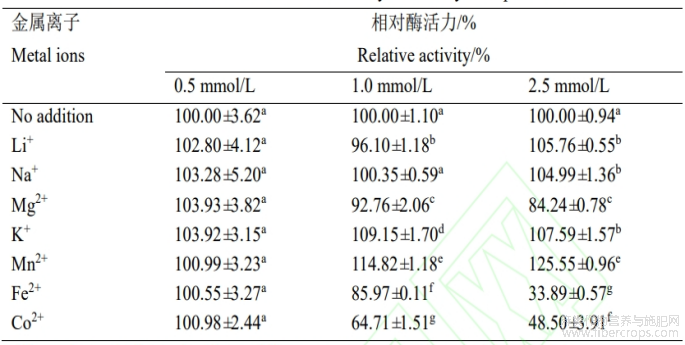
0.5mmol/L、1mmol/L和2.5mmol/L的Li+、Na+和K+对rBpumPel-Ec酶活力的影响范围均10%(表2)。0.5mmol/L的Mg2+、Fe2+和Co2+对rBpumPel-Ec酶活力基本无影响,但随着受测浓度的升高而表现出抑制作用。
表2 金属离子对 rBpumPel-Ec 酶活力的影响
Different letters indicate significant differences compared with no addition (P<0.05)
2.4 rBpumPel-Ec 用于苎麻脱胶
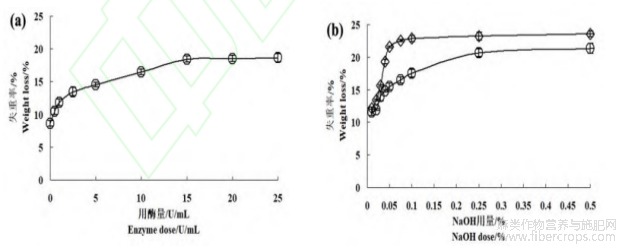
当仅以缓冲液浸泡时(即用酶量为0U/mL),苎麻的失重率为8.60%[图6(a)]。当用酶处理时,苎麻的失重率随着用酶量的上升而上升,至15U/mL时达到18.44%,继续提高用酶量至20U/mL和25U/mL时,失重率未能继续提升。当使用0.000%-0.125%NaOH在30℃脱胶6h时,失重率随用量的上升而急剧上升,当用量达到0.5%时达到顶峰,此时失重率为21.34%[图6(b),(圆形图标)];而当在105℃脱胶2.5h时,失重率的增长速度比在30℃脱胶6h时更快,并且最终获得的失重率更高(当NaOH用量达到0.5%时失重率为23.54%)[图6(b),(菱形图标)]。
图 6 rBpumPel-Ec(a)和 NaOH(b)用于苎麻脱胶
Use of NaOH for ramie degumming was conducted at 30 ℃ for 6 h (empty cycle) and at 105 ℃ for 2.5 h (empty diamond).
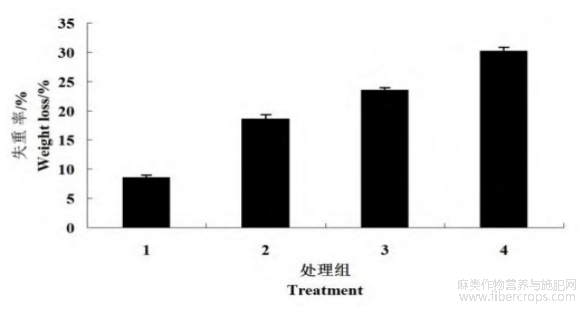
如图7所示,当使用0.5%NaOH对已使用25U/mL rBpumPel-Ec处理后的苎麻韧皮部进行脱胶时(在105℃侵泡2.5h),所得失重率(30.20%)高于25U/mL rBpumPel-Ec(21.34%)和0.5%NaOH单独脱胶时(23.54%),说明rBpumPel-Ec可提升NaOH的脱胶效果。
图7 rBpumPel-Ec 和 NaOH 单独和联用于苎麻脱胶的效果比较
Treatment one: control; Treatment two: 25 U/mL rBpumPel-Ec at 30 ℃ for 6 h; Treatment three: 0.5% NaOH at 105 ℃ for 6 h; Treatment four: combination of 25 U/mL rBpumPel-Ec and 0.5% NaOH at 105 ℃ for 6 h.
3 讨论
3.1 重组酶的纯化和功能鉴定
rBpumPel-Ec的表观分子量约为40kDa,与其理论分子量42.56kDa一致。当使用同样量的原料和能量时,表达宿主能合成更多小分子量的蛋白质,因此相比于分子量较大的内切果胶酸裂解酶,如来自B. amyloliquefaciens S6的recPELS6(分子量65.75kDa)[27]和来自B.licheniformis的PMGL-Ba(分子量54.6kDa)[28]],rBpumPel-Ec具备天然优势。
由于内切果胶酸裂解酶的作用模式为随机降解果胶内部的α-(1,4)-半乳糖醛酸键且以uG2和uG3为主要终产物,极少存在uG1和uG4,而rBpumPel-Ec的降解终产物只有uG2和uG3,因此rBpumPel-Ec为内切果胶酸裂解酶[29]。与外切果胶酸裂解酶相比,内切酶能够更快促使果胶长链的聚合度降低,更加适合被用于脱胶[30]。
3.2 酶学性质
植物组织的自然pH值普遍为弱酸性[31],因此相比于最适作用pH值更高的内切果胶酸裂解酶,如来自 Humicola insolens Y1的PLHY1(pH10.0)[32]、B.clausiiS-4的PNL(pH10.0)[21]、Paenibacillus polymyxa KF-1的PpPel9a(pH10.0)[33]、Bacillus sp.RN1的BspPel(pH10.0)[15]和Bacillus sp.strainP-4-N的Pel-4A(pH11.5)[34],rBpumPel-Ec更加适合在自然环境中用于脱胶。rBpumPel-Ec的最适作用温度低于Bacillus sp.RN1的PelSWU(90℃)[35]、Bacillus sp.RN1的BspPel(80℃)[15]和Bacillus sp.strainP-4-N的Pel-4A(70℃)[34],但rBpumPel-Ec在30℃表现出更高的相对酶活力,因而更适合用于低温脱胶。
金属离子是植物的必需元素,广泛存在于植物体内的任何组织中。当应用的对象为植物组织时,活力不受金属离子抑制的酶更具有优势。苎麻植株地上部分中的金属离子可达0.18mmol/kg(以最高者镉为例)[36],按本研究脱胶体系换算则体系内浓度为0.0088mmol/L。当额外添加的金属离子浓度为0.5mmol/L时,所有受试金属离子均不表现出抑制作用,该优良特性可确保rBpumPel-Ec在苎麻脱胶体系中正常发挥作用。
3.3 苎麻脱胶
仅以缓冲液浸泡时能使苎麻韧皮部失重8.60%,导致该结果的原因是苎麻韧皮部经“刮青”后结构严重破损,与韧皮纤维结合松散的物质在液体浸泡时自动脱离[37]。Xu等[16]、Liu等[18]和Zheng等[19]亦发现以缓冲液浸泡时能使苎麻韧皮部失重,与本研究所得结果相似。与微生物脱胶法相比,rBpumPel-Ec脱胶所用时间仅为6h,远远短于Shu等[10]使用细菌菌株HG-49脱胶所需要的16h、Cheng等[38]使用4个细菌菌株脱胶所需要的14.5h、Yang等[11]使用5个细菌菌株脱胶所需要的12h和Cheng等[39]使用细菌菌株B.cereushn1-1脱胶所需要的10h,因此rBpumPel-Ec脱胶比微生物脱胶法更省时。
虽然酶的应用前景与酶学性质有关,但植物材料成分复杂,导致酶的脱胶环境比酶学性质的测定体系复杂,因而应以脱胶效果作为酶应用前景的推断依据[37]。rBpumPel-Ec单独脱胶所得最高失重率(18.44%)远高于Zheng等[19]使用来自Bacillus sp.RN1的BspPel获得的失重率(9.20%)、Xu等[16]使用来自Dickeya dadantiiDCE-01的Pel419突变酶获得的失重率(12.89%)、Zou等[40]使用来自B.subtilis7-3-3的B-pN-pelA获得的失重率(13.50%)和Kapoor等[41]使用来自Bacillus sp.的MG-cp-2所获得的失重率(13.00%),表明rBpumPel-Ec单独使用时拥有较高的脱胶效率。
值得注意的是,虽然已有文献报道获得在联用时能提升NaOH脱胶效果的果胶酸裂解酶,但这些报道中所得失重率均未高于rBpumPel-Ec与NaOH组合脱胶时所得数值,并且rBpumPel-Ec的脱胶温度(30℃)远低于Zhao等[14](来自Paenibacillus polymyxa KF-1的PpPel10a,脱胶温度50℃)、Xu等[16](来自Dickeya dadantiiDCE-01的Pel403,脱胶温度50℃)、Liu等[18](来自Bacillus sp.B58-2的rBvelPL1-Ec,脱胶温度50℃)、Zheng等[19](来自Bacillus sp.RN1的BspPel,脱胶温度50℃)、Xu等[17](来自Dickeya dadantii DCE-01的PelG419突变酶,脱胶温度55℃)和Zhen等[15](来自Paenibacillus sp.0602的pelNK93I,脱胶温度60℃)所用的脱胶温度,因此相比于上述果胶酸裂解酶,rBpumPel-Ec表现出可以节省用于维持酶法脱胶条件所用能耗的优点。
3.4 该酶的后续研究方向
结合已的果胶酸裂解酶的相关报道,后续应对rBpumPel-Ec进行以下3方面研究。
(1)蛋白质工程。rBpumPel-Ec的比酶活力(78.26U/mg)低于PNL(来自B.clausii,297.10U/mg)[21]?EPLM(来自宏基因组文库,1524.10U/mg)[42]和rBvelPL1-Ec(来自Bacillus sp.B58-2,1524.10U/mg)[18],并且rBpumPel-Ec在30℃的热稳定性略有不足(虽然rBpumPel-Ec可实现低温脱胶,但提高rBpumPel-Ec在30℃的热稳定性则可以节约用酶成本),可通过蛋白质工程的方式解决这些问题?例如Wang等[43]将与PEL168(来自B.subtilis168,比酶活力353.6±25.6U/mg)活性相关的氨基酸进行突变,获得比酶活力提高至873.3±65.3U/mg的双突变体K47E/V132F;Xu等[17]将潜在影响PelG403(来自Dickeya Dadantii DCE-01)热稳定性的氨基酸进行突变,获得在50℃t1/2为野生型2.37倍的突变体A129V。
(2)生产成本。本研究使用大肠杆菌生产获得rBpumPel-Ec,在证实rBpumPel-Ec具有应用潜力后应着眼于降低其生产成本。丝状真菌黑曲霉[44]、米曲霉[45]和草酸青霉[46]等物种可产纤维素酶、淀粉酶、木聚糖酶等多种生物质水解酶,能通过基因组和转录组作为宿主表达外来基因[47],因此后续研究可将BpumPel转入丝状真菌表达系统以使用廉价的农副产品和农业废弃物为基质持续高产该酶。
(3)多酶联用协同脱胶。植物细胞壁最基础的结构为果胶、木聚糖、甘露聚糖、阿拉13伯聚糖等半纤维素与纤维素构成的网络结构,因此在脱胶时加入其它生物大分子降解酶可提升脱胶效果[48]。例如在微生物脱胶法中,Yang等[11]将高产果胶酸裂解酶的Dickeya dadantii菌株DCE-01与高产木聚糖酶的B.cereus菌株B7进行共培养,将苎麻失重率提升至27%。相似地,Edwards等[49]和Duan等[50]在酶法降解细胞壁生物大分子的研究中亦发现木聚糖酶和甘露聚糖酶与果胶酶之间存在协同作用,因此后续研究可将rBpumPel-Ec与其它生物大分子降解酶配合协同脱胶。
4 结论
本研究从土壤中筛选出一株产果胶酸裂解酶的细菌B.15-1,通过分析其基因组中编码16SRNA的部分基因序列将其鉴定为短小芽孢杆菌。将其基因组中1个编码假定果胶酸裂解酶的基因克隆并在大肠杆菌中表达,获得重组内切果胶酸裂解酶rBpumPel-Ec。rBpumPel-Ec的最适作用pH为8.5,最适作用温度为55℃,且具有优良的金属离子相容性。当先用25U/mL rBpumPel-Ec处理苎麻韧皮部再使用0.5%NaOH处理时,所得失重率(30.20%)高于rBpumPel-Ec(21.34%)和NaOH单独脱胶时(23.54%),说明rBpumPel-Ec可在低温下实现对苎麻韧皮部的单独脱胶,并且能提升NaOH脱胶的效果,因此rBpumPel-Ec在苎麻韧皮部脱胶方面具有潜在的应用价值。
参考文献
[1] REHMAN M, GANG D, LIU Q Q, et al. Ramie, a multipurpose crop: potential applications, constraints and improvement strategies [J]. Industrial Crops and Products,2019,137:300-307.
[2] KIPRIOTIS E, HEPING X, VAFEIADAKIS T, et al. Ramie and kenaf as feed crops [J]. Industrial Crops and Products,2015,68:126-130.
[3] KUANG X Q, GUAN S P, RODGERS J, et al. Study on length distribution of ramie fibers[J].The Journal of The Textile Institute,2017,108(11):1853-1862.
[4] 郑文龙,马兰,刘佳杰,等.苎麻剥制机械研究现状与发展趋势[J].中国麻业科学,2024,46(1):47-59.
[5] 王瑛,冯艳,谭冬梅,等.苎麻自动剥麻机设计与试验[J].农业工程,2022,12(2):98-101.
[6] CHENG L F, DUAN S W, FENG X Y, et al. Ramie-degumming methodologies: a short review[J].Journal of Engineered Fibers and Fabrics, 2020,15:1558925020940105.
[7] LIN G Y, TANG Q, HUANG H, et al. One-step extraction of ramie cellulose fibers and reutilization of degumming solution[J].Textile Research Journal,2022,92(19/20):3579-3590.
[8] QU Y S, ZHAO S Y, SHI Z H, et al. High-efficiency organosolv degumming of ramie fiber by autocatalysis of high-boiling alcohols: anevaluation study of solvents[J].Cellulose,2020, 27(8):4271-4285.
[9] TANG Q, CHEN Y X, DU M J, et al. Research progress in ramie fiberextraction: degumming method, working mechanism, and fiber performance[J]. Industrial Crops and Products, 2024, 222:119876.
[10] SHU T, BAI Y, WANG Y W, et al. A high-efficiency and eco-friendly degumming process for ramie fibers[J]. Journal of Cleaner Production, 2020, 276:124217.
[11] YANG Q, DUAN S W, CHENG L F, et al. An effective degumming technology for ramie fibers based on microbial coculture strategy[J]. Journal of Natural Fibers,2022,19(4):1555-1565.
[12] DING J X, ZHOU C, DONG Z. Trend of ramie industry development: a review of green degumming and the utilization of processing residues[J]. Journal of Cleaner Production,2023,384:135487.
[13] SHARMA N, RATHORE M, SHARMA M. Microbial pectinase: sources, characterization and applications[J]. Reviews in Environmental Science and Bio/Technology, 2013, 12(1): 45-60.
[14] ZHAO Y, YUAN Y, ZHANG X Y, et al. Screening of a novel polysaccharide lyase family 10 pectate lyase from Paenibacillus polymyxa KF-1: cloning, expression and characterization [J]. Molecules,2018,23(11):2774.
[15] ZHEN J, TAN M, FU X P, et al. High-level extracellular production of an alkaline pectate lyase in E. coli BL21 (DE3) and its application in bioscouring of cotton fabric [J].3 Biotech,2020,10(2):49.
[16] XU H, DUAN S W, FENG X Y, et al. Improving the thermo-activity and-stability of pectate lyase from Dickeya dadantii DCE-01 for ramie degumming[J]. Processes,2021,9(12):2106.
[17] XU H, FENG X Y, YANG Q, et al. Improvement on thermostability of pectate lyase and its potential application to ramie degumming[J]. Polymers,2022,14(14):2878.
[18] LIU S J, QIN Y, WANG Q Y, et al. A novel pectate lyase with high specific activity from Bacillus sp.B58-2: Gene cloning, heterologous expression and use in ramie degumming[J]. Enzyme and Microbial Technology,2024,175:110395.
[19] ZHENG X Y, ZHANG Y M, LIU X X, et al. High-level expression and biochemical properties of a thermo-alkaline pectate lyase from Bacillus sp. RN1 in Pichia pastoris with potential in ramie degumming[J]. Frontiers in Bioengineering and Biotechnology,2020,8:850.
[20] TAKAO M, NAKANIWA T, YOSHIKAWA K, et al. Molecular cloning, DNA sequence, and expression of the gene encoding for thermostable pectate lyase of thermophilic Bacillus sp. TS 47[J]. Bioscience, Biotechnology, and Biochemistry,2001,65(2):322-329.
[21] LI Z M, BAI Z H, ZHANG B G, et al. Purification and characterization of alkaline pectin lyase from a newly isolated Bacillus clausii and its application in elicitation of plant disease resistance[J].Applied Biochemistry and Biotechnology,2012,167(8):2241-2256.
[22] ZHENG Y Y, HUANG C H, LIU W T, et al. Crystal structure and substrate-binding mode of a novel pectate lyase from alkaliphilic Bacillus sp. N16-5[J]. Biochemical and Biophysical Research Communications,2012,420(2):269-274.
[23] KEEN N T, TAMAKI S. Structure of two pectate lyase genes from Erwinia chrysanthemi EC16 and their high-level expression in Escherichia coli[J]. Journal of Bacteriology,1986,168(2):595-606.
[24] ZHOU C, XUE Y F, MA Y H. Characterization and overproduction of a thermo-alkaline pectate lyase from alkaliphilic Bacillus licheniformis with potential in ramie degumming[J].Process Biochemistry,2017,54:49-58.
[25] LAEMMLI U K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4 [J]. Nature,1970,227(5259):680-685.
[26] BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding [J]. Analytical Biochemistry,1976,72:248-254.
[27] BEKLI S, AKTAS B, GENCER D, et al. Biochemical and molecular characterizations of a novel pH- and temperature-stable pectate lyase from Bacillus amyloliquefaciens S6 for industrial application[J]. Molecular Biotechnology,2019,61(9):681-693.
[28] LI J Y, YANG M L, ZHAO F G, et al. Efficient expression of an alkaline pectin lyase from Bacillus licheniformis in Pichia pastoris [J]. Bioresources and Bioprocessing,2024,11(1):37.
[29] YADAV S, YADAV P K, YADAV D, et al. Pectin lyase: a review [J]. Process Biochemistry, 2009, 44(1): 1-10.
[30] PATEL V B, CHATTERJEE S, DHOBLE A S.A review on pectinase properties, application in juice clarification, and membranes as immobilization support[J].Journal of Food Science,2022,87(8):3338-3354.
[31] GRASSIN C, COUTEL Y. Enzymes in fruit and vegetable processing and juice extraction [M].Enzymes in Food Technology, Second edition,2009:236-263.
[32] WANG Z Y, XU B, LUO H Y, et al. Production pectin oligosaccharides using Humicola insolens Y1-derived unusual pectate lyase[J]. Journal of Bioscience and Bioengineering,2020,129(1):16-22.
[33] YUAN Y, ZHANG X Y, ZHAO Y, et al. A novel PL9 pectate lyase from Paenibacillus polymyxa KF-1:cloning, expression, and its application in pectin degradation [J]. International Journal of Molecular Sciences,2019,20(12):3060.
[34] HATADA Y, KOBAYASHI T, ITO S. Enzymatic properties of the highlythermophilic and alkaline pectate lyase Pel-4B from alkaliphilic Bacillus sp. strain P-4-N and the entire nucleotide and amino acid sequences [J]. Extremophiles,2001,5(2):127-133.
[35] SUKHUMSIIRCHART W, KAWANISHI S, DEESUKON W, et al. Purification,characterization, and overexpression of thermophilic pectate lyase of Bacillus sp. RN1 isolated from a hot spring in Thailand[J]. Bioscience, Biotechnology, and Biochemistry,2009,73(2):268-273.
[36] ZHU S, SHI W, ZHANG J. Effect of different ramie (Boehmeria nivea L. Gaud) cultivars on the adsorption of heavy metal ions cadmium and lead in the remediation of contaminated farmland soils [J].Open Chemistry,2022,20(1):444-454.
[37] MUDI C, SHAW A. Investigations on the mechanical performance anddurability of ramie fibers under various environmental conditions [J].Journal of Natural Fibers,2025,22(1):2433043.
[38] CHENG L F, DUAN S W, FENG X Y, et al. Screening and identification of pectinolytic bacteria for ramie degumming[J].Textile Research Journal,2021,91(9/10):1056-1064.
[39] CHENG L F, WANG Q M, FENG X Y, et al. Screening a bacterium and its effect on the biological degumming of ramie and kenaf [J].Scientia Agricola,2018,75(5):375-380.
[40] ZOU M Y, LI X Z, SHI W J, et al. Improved production of alkalinepolygalacturonate lyase by homologous overexpression pelA in Bacillussubtilis[J]. Process Biochemistry,2013,48(8):1143-1150.
[41] KAPOOR M, BEG Q K, BHUSHAN B, et al. Application of an alkaline and thermostable polygalacturonase from Bacillus sp. MG-cp-2 in degumming of ramie (Boehmeria nivea) and sunn hemp (Crotalaria juncea) bast fibres[J]. Process Biochemistry,2001,36(8/9):803-807.
[42] SHARMA N, SAHOO D, RAI A K, et al. A highly alkaline pectate lyase from the Himalayan hot spring metagenome and its bioscouring applications [J].Process Biochemistry,2022,115:100-109.
[43] WANG X W, LU Z H, XU T, et al. Improving the specific activity and thermo-stability of alkaline pectate lyase from Bacillus subtilis 168 for bioscouring[J].Biochemical Engineering Journal,2018, 129:74-83.
[44] LI C, ZHOU J W, DU G C, et al. Developing Aspergillus niger as acell factory for food enzyme production[J].Biotechnology Advances,2020,44:107630.
[45] MACHIDA M, ASAI K, SANO M, et al. Genome sequencing and analysisof Aspergillus oryzae [J].Nature,2005,438(7071):1157-1161.
[46] YAN Y S, ZHAO S, LIAO L S, et al. Transcriptomic profiling and geneticanalyses reveal novel key regulators of cellulase and xylanase gene expression in Penicillium oxalicum [J].Biotechnology forBiofuels,2017,10:279.
[47] WANG L, ZHAO S, CHEN X X, et al. Secretory overproduction of a raw starch-degrading glucoamylase in Penicillium oxalicum using strongpromoter and signal peptide [J].Applied Microbiology and Biotechnology,2018,102(21):9291-9301.
[48] SCHELLER H V, ULVSKOV P. Hemicelluloses [J].Annual Review of Plant Biology,2010,61:263-289.
[49] EDWARDS M C, WILLIAMS T, PATTATHIL S, et al.Replacing a suite ofcommercial pectinases with a single enzyme, pectate lyase B, in Saccharomyces cerevisiae fermentations of cull peaches [J].Journalof Industrial Microbiology & Biotechnology,2014,41(4):679-686.
[50] DUAN S W, XU B R, CHENG L F, et al.Bacterial strain for bast fiber crops degumming and its bio-degumming technique [J].Bioprocessand Biosystems Engineering,2021,44(12):2503- 2512.
文章摘自:周瑨,刘思伽,秦艳,王青艳,冼亮,梁欣泉.短小芽孢杆菌果胶酸裂解酶的功能鉴定及其在苎麻低温脱胶中的应用[J/OL].广西科学.https://doi.org/10.13656/j.cnki.gxkx.20250407.001。