摘 要:DMP家族成员是植物特有的膜蛋白,在植物生殖发育及衰老过程中发挥重要作用。亚麻是我国重要的油料及经济作物,目前关于DMP基因家族在亚麻中的分布及功能研究尚未有报道。本研究利用生物信息学方法对亚麻DMP基因家族进行鉴定,对其序列特征、系统进化、复制事件进行了分析,并利用转录组数据和qRT-PCR分析其在不同组织器官及胁迫处理下的表达模式。本研究以陇亚10号基因组为参照,利用同源性分析共鉴定到17个LuDMP成员,命名为LuDMP-1~LuDMP-17,不均匀分布在9条染色体上。序列特征分析结果表明,除LuDMP-9、LuDMP-16和LuDMP-17外,其余成员均不含内含子;基因启动子区含有大量激素与逆境胁迫相关响应元件;编码的蛋白氨基酸长度为195~240aa,分子量为8700~25300Da,等电点为4.76~9.71;所有蛋白均为疏水性蛋白,含有2~5个跨膜结构域及4~8个motif,其中有4个motif存在于所有LuDMP蛋白;绝大多数蛋白定位于细胞膜。LuDMP家族共有3对串联复制事件和8对片段复制事件,进化过程中经历了强烈的纯化选择。转录组分析结果表明,大多数LuDMP成员在果实和茎中呈现特异性表达模式。系统进化分析将DMP蛋白分为5个亚族,第III亚族DMP数量最多,含有11个LuDMP成员,各亚族中DMP蛋白具有单子叶或双子叶特异性聚类模式。第IV亚族中的LuDMP-1和LuDMP-7与拟南芥及玉米单倍体诱导基因具有很近的亲缘关系,位于同一进化分支,序列同源性达到67%以上,而且该基因在成熟花药中的表达量明显高于其他组织,推测其为亚麻单倍体诱导的候选基因。本研究在陇亚10号中克隆到了LuDMP-1,全长为699bp。LuDMP-1/7启动子区域含有激素响应及逆境胁迫相关作用元件,吲哚乙酸、赤霉素、萘乙酸、聚乙二醇、高温及低温处理下,LuDMP-1/7基因表达量均有不同程度的提高,其中对低温和高温的响应最明显,分别较对照提高了约260和600倍。本研究为阐明亚麻DMP蛋白的功能奠定了基础,并为亚麻单倍体育种提供了基因资源。
关键词:亚麻;DMP基因家族;单倍体诱导;表达分析
膜蛋白在细胞增殖与分化、信号转导与识别及物质运输等众多生物过程中发挥重要作用[1-4]。Mori等[5]发现定位于精子膜的拟南芥(Arabidopsis thaliana)蛋白GEX2对精子附着并最终完成双受精是必需的。DMPs(domain of unknown function 679 membrane proteins)家族成员是一种绿色植物特有的膜蛋白,通常含有4个跨膜结构域,具有胞质氨基端和羧基端,参与植物生殖发育及衰老等各种生理过程[6]。目前,已经在拟南芥全基因组水平上鉴定到10个DMP基因家族成员,不同成员在不同组织器官中具有独特的表达模式,其中AtDMP1,AtDMP2,AtDMP3,AtDMP4和AtDMP7参与不同类型的程序性细胞死亡,包括器官衰老,角果开裂及花器官和角果的脱落,而AtDMP8和AtDMP9在双受精过程中促进配子融合[7]。Zhong等[8]利用图位克隆鉴定到玉米(Zea mays)DMP基因,并且发现DMP突变植株可诱导母本单倍体的产生。双子叶植物拟南芥(A.thaliana)AtDMP8和AtDMP9功能缺失也可诱导母本单倍体,而且ZmDMP-like蛋白在许多双子叶植物中都很保守,序列同源性高达74%,这说明DMP基因突变可以在双子叶植物中触发单倍体诱导[9]。Zhu等[10]在棉花(Gossypiumspp)中对DMP家族进行了系统分析,发现棉花DMP基因参与植物衰老及生殖过程,其中GhDMP8-A/-D和GbDMP8-A/-D可作为棉花单倍体诱导的候选基因。
亚麻(Linum usitatissimum)属于亚麻科亚麻属,是一种古老的韧皮纤维作物和油料作物[11]。作为人类最早栽培利用的农作物之一,亚麻在全世界被广泛种植,主要的种植国家有中国、加拿大、美国、阿根廷等[12]。按其经济性状与用途可将亚麻分为油用、纤维用和油纤维兼用等3种类型[13-15]。油用亚麻又称胡麻,富含α-亚麻酸、木酚素、膳食纤维、蛋白质等多种营养物质,其中α-亚麻酸含量达到55%左右,是人类摄取ω-3脂肪酸的最好来源之一。
随着测序技术的发展,玉米[16]、高粱(Sorghum bicolor)[17]、亚麻[18]、大豆(Glycine max)[19]等多种作物完成了全基因组测序,而基因家族分析也成了挖掘功能基因的有效手段。目前,LBD蛋白(lateralorgan boundaries domain)[20]、天冬氨酸蛋白酶(aspartate protease,AP)[21]、多聚半乳糖醛酸酶(polygalacturonase,PG)[22]等基因家族在很多物种中都被鉴定出来,并进行了系统分析,而DMP基因家族在各种作物中的研究还相对缓慢,亚麻作为重要的油料及经济作物,目前尚无相关报道。本研究旨在鉴定亚麻DMP基因,并对其进行生物信息学及表达模式分析,从而为亚麻育种及功能基因组研究提供基因资源。
1材料与方法
1.1试验材料
本研究选用甘肃省农业科学院育成的胡麻品种“陇亚10号”“黑亚14号”“陇亚15号”(丰产性、抗病性等综合性状优良)[23]作为试验材料。供试材料于2021年3月种植于甘肃省农业科学院兰州试验基地(36°03′N,103°40′E,海拔1500m)。在开花期进行标记,分别取开花后10、20、30、40和50d的种子及未成熟花药和成熟花药。在培养皿中萌发陇亚15号种子,10d后选取生长一致的幼苗移至1/2MS液体培养基中,3d后进行胁迫处理同时更换新的培养基。分别对亚麻幼苗进行盐胁迫(150mmol·L-1氯化钠)、干旱处理(20%聚乙二醇PEG)、吲哚乙酸处理(15μmol·L-1IAA)、萘乙酸处理(5μmol·L-1NAA)、赤霉素处理(5μmol·L-1GA3)、高温胁迫(45℃处理3h)及低温胁迫(4℃处理3h),分别在处理后0、3、6、9、12、24、48、72h及恢复后48h取其叶片。所有样品用液氮速冻后均保存于-80℃超低温冰箱备用。
1.2亚麻LuDMP基因家族的鉴定与克隆
亚麻基因组序列从NCBI下载(登录号为QMEI02000000),其他基因组注释文件下载地址为(https://doi.org/10.6084/m9.figshare.13614311.v3)。利用文献[10]报道的玉米、水稻(Oryza sativa)、葡萄(Vitis vinifera)、大豆及拟南芥DMP基因ID号,进而通过各自基因组蛋白序列提取相应的DMP序列,利用Blastp在亚麻基因组中进行同源比对,并采用Pfam(http://pfam.sanger.ac.ck/)对结构域进行确认,获得亚麻DMP-like基因。将LuDMP序列与玉米和拟南芥单倍体诱导相关DMP蛋白序列进行比对,选择同源性最高的基因作为亚麻单倍体诱导的候选基因并对其进行克隆。以陇亚10号基因组DNA和成熟花药的cDNA为模板分别进行PCR扩增,PCR扩增体系为50μL:Buffer25μL;dNTP10μL;高保真酶KOD1μL;正向引物(10μmol·L-1)1.5μL;反向引物(10μmol·L-1)1.5μL;cDNA模板(100ng·μL-1)2μL;无RNase水9μL。PCR反应条件:94℃2min;98℃10s,60℃30s,68℃50s,35个循环;72℃7min。扩增产物经1%琼脂糖凝胶电泳后酶切回收,之后用Zero Blunt® TOPO® PCR Cloning Kit连接至T载体并转化DH-5α,将阳性菌液进行测序。测序结果经比对分析后,将正确的基因序列上传至NCBI数据库。
1.3亚麻LuDMP家族序列特征分析
利用ExPASy ProtParam(http://web.wxoasy.org/protparam/)预测LuDMP蛋白的分子量和等电点;并使用Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)和ExPASy ProtScale(https://web.expasy.org/protscale/)分别预测LuDMP蛋白的亚细胞定位和亲疏水性,利用TMHMM-2.0(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)预测LuDMP蛋白的跨膜结构域。LuDMP蛋白结构预测采用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)和SWISSMODEL(https://swissmodel.expasy.org/interactive)。利用MEME(http://alternate.meme-suite.org/tools/meme)分析LuDMP蛋白的保守基序。利用PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对LuDMP基因启动子的顺式作用元件进行预测。
1.4亚麻LuDMP家族成员的染色体分布及复制分析
LuDMP基因染色体定位及可视化采用MapGene to Chromosome在线软件。利用MCScanX分析LuDMP基因的复制事件,之后采用DnaSP6计算基因对之间的非同义替换率(nonsynonymous mutation rate,Ka)、同义替换率(synonymous substitution rate,Ks)和Ka/Ks比值,利用Ka/Ks比值分析选择压力。用Mya=Ks/(2×6.1×10-9)×10-6计算基因对之间的分歧时间(million years ago,MYA)[24]。
1.5DMP蛋白的系统进化分析
利用MEGA7.0软件对亚麻、拟南芥、葡萄、大豆、水稻及玉米6个物种的DMP蛋白序列进行比对,比对结果采用邻接法(neighbor-joining;参数:Bootstrap1000)构建DMP蛋白的系统进化树。
1.6LuDMP基因的表达分析
用RNA提取试剂盒(BIOMGA)提取RNA,具体操作步骤按照说明书进行。经NanoDrop-2000进行浓度和质量检测后,利用TaKaRa反转录试剂盒将RNA反转录成cDNA。qRT-PCR采用TB GreenTM Premix Ex TaqTM II(TaKaRa)荧光定量试剂盒。PCR反应体系为20μL:2×mix10μL;上下游引物(10μmol·L-1)各1μL;cDNA模板2μL;无RNase水6μL。qRT-PCR程序为50℃2min;95℃10min;95℃15s,60℃15s,72℃15s,40个循环。每个样品3次重复,选取GAPDH为内参基因,运用2-ΔΔCt方法[25]计算基因的相对表达量。本研究所用转录组数据从NCBI下载(登录号为PRJNA505721),利用TBtools绘制热图,本研究所用引物均见表1(由于LuDMP-1与LuDMP-7序列相似性很高,故二者所用引物相同)。
表1 本研究所用引物
2结果与分析
2.1亚麻LuDMP基因家族的鉴定与系统进化分析
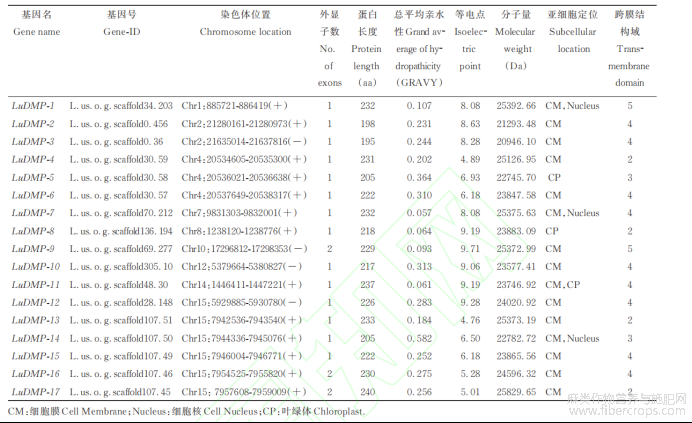
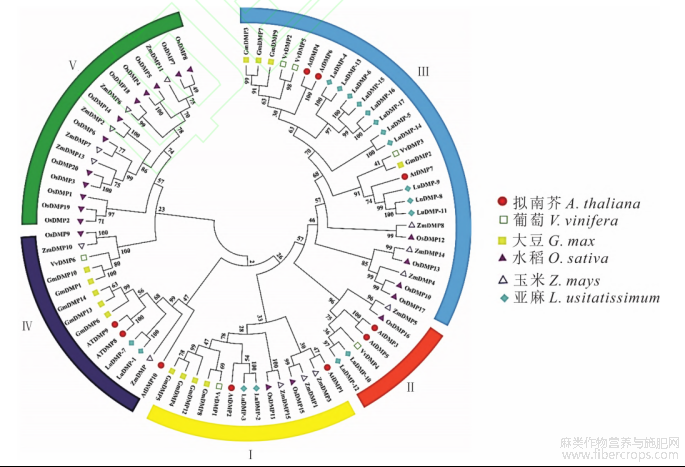
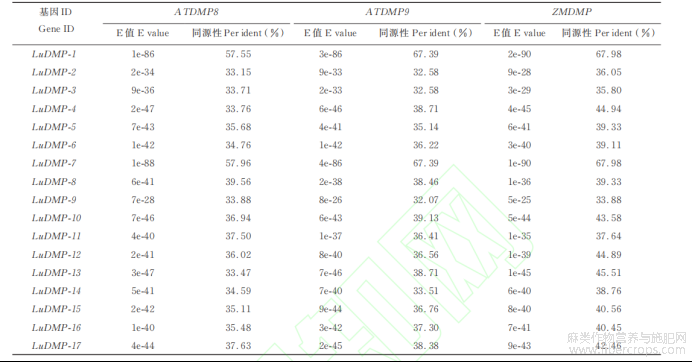
通过BlastP比对,并利用Pfam数据库进行结构域分析,最终在亚麻基因组中鉴定到17个DMP成员(表2)。根据LuDMP基因在染色体上的位置,将其依次命名为LuDMP-1~LuDMP-17。为深入了解LuDMP与其他物种DMP之间的进化关系,本研究将拟南芥、大豆、葡萄、玉米及水稻等5个物种的DMP蛋白序列与LuDMP蛋白序列进行多序列比对并构建系统进化树。结果显示,6个物种的DMP蛋白可分为I、II、III、IV和V5个亚族(图1),第III亚族DMP数量最多,包含28个基因,其中LuDMP11个,其次为第V、IV、I亚族,分别含17、14、14个基因,其中LuDMP分别为0、2、2个,第II亚族DMP数量最少,包含7个基因,其中LuDMP2个,每个亚族均含有单子叶与双子叶植物DMP,而且各亚族中的DMP蛋白具有单子叶或双子叶特异性聚类模式。和其他双子叶植物相比,亚麻DMP基因发生了显著扩张。此外,第IV亚族中的LuDMP-1和LuDMP-7与单倍体诱导基因AtDMP8、AtDMP9和ZmDMP[8-9]具有很近的亲缘关系,位于同一个进化分支,而且LuDMP-1和LuDMP-7与AtDMP9及ZmDMP蛋白的同源性达到67%以上(与AtDMP8的同源性为57%),这表明它们可能具有相同的功能,可以作为亚麻单倍体诱导的候选基因,而其余LuDMP成员与ZmDMP和AtDMP8的同源性低于46%(表3)。
表2 亚麻LuDMP成员基本特征
图1 DMP蛋白系统进化树
表3 LuDMP基因同源性比对
2.2亚麻单倍体候选基因LuDMP-1/7的克隆
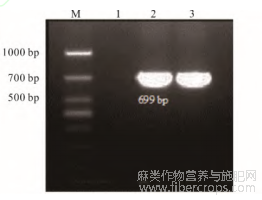
对LuDMP-1和LuDMP-7基因进行扩增,只扩增得到LuDMP-1基因的DNA序列和CDS序列,大小均为699bp,与参考序列大小相符(图2),将LuDMP-1基因序列上传至NCBI,GenBank登录号为OM234689。
图2 LuDMP-1基因全长扩增
M:DL1000marker;1:阴性对照;2:DNA扩增;3:cDNA扩增。1:Negative control;2:DNA amplification;3:cDNA amplification.
2.3亚麻LuDMP基因序列分析
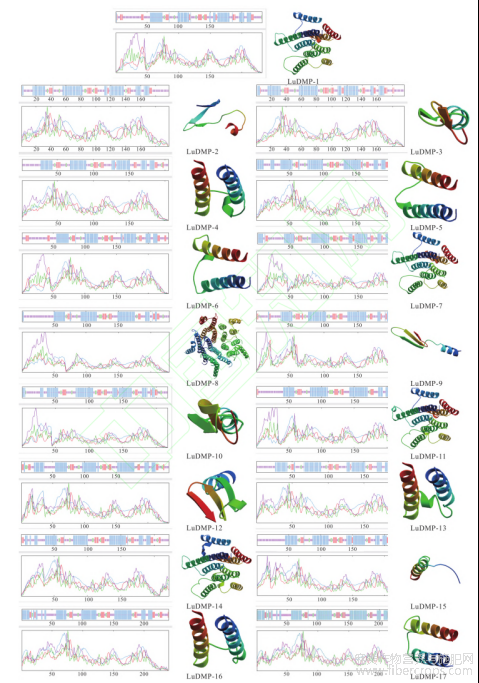
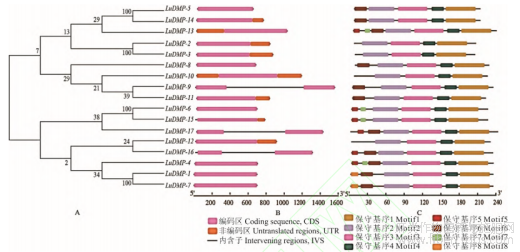
亚麻LuDMP蛋白氨基酸长度在195~240aa,分子量为20946~25830Da,等电点(isoelectric point,PI)为4.76~9.71,所有LuDMP蛋白的总平均亲水性(GRAVY)均大于0,即所有LuDMP蛋白均为疏水性蛋白(表2,图3)。亚细胞定位预测结果表明,除LuDMP-5和LuDMP-8定位于叶绿体,其余蛋白均定位于细胞膜,其中LuDMP-1、LuDMP-7和LuDMP-14定位于细胞膜和细胞核,LuDMP-11定位于细胞膜和叶绿体(表2)。此外,LuDMP蛋白均具有跨膜结构域,数量2~5个不等(表2)。LuDMP蛋白二级结构预测结果表明,所有LuDMP蛋白均含有α-螺旋、β-转角、无规则卷曲以及延伸链(图4)。LuDMP蛋白种内进化关系如图5A所示,对LuDMP进行基因结构分析,除LuDMP-9、LuDMP-16和LuDMP-17外,其余LuDMP成员均不含内含子(图5)。对17个LuDMP蛋白的保守基序进行分析,共鉴定到8个motif,其中motif1、motif2、motif3和motif4在17个LuDMP成员中高度保守,然而一些特定的基序只存在于特定的基因中,如motif5只存在于LuDMP-4、LuDMP-6、LuDMP-13、LuDMP-15、LuDMP-16及LuDMP-17;motif7只存在于LuDMP-4、LuDMP-6、LuDMP-13及LuDMP-15;motif6存在于除LuDMP-2、LuDMP-3、LuDMP-10及LuDMP-12之外的所有成员中;而motif8只存在于LuDMP-1和LuDMP-7中,推测该基序与单倍体诱导相关。
图3 LuDMP蛋白亲疏水性预测
图4 LuDMP蛋白二三级结构
图5 LuDMP基因系统进化、基因结构与保守基序分析
2.4LuDMP基因染色体定位和复制分析
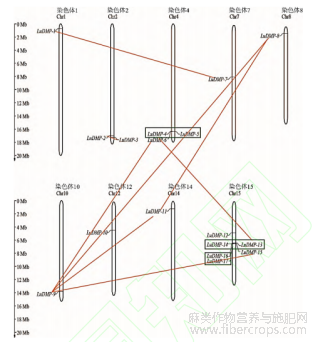
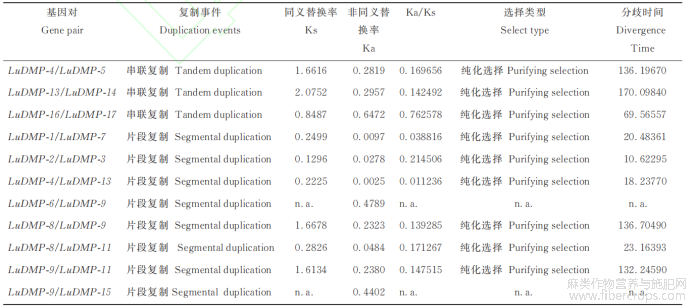
对17个LuDMP基因进行染色体定位,结果显示LuDMP-1、LuDMP-7、LuDMP-8、LuDMP-9、LuDMP-10和LuDMP-11分别位于1、7、8、10、12和14号染色体,LuDMP-2和LuDMP-3位于2号染色体,LuDMP-4、LuDMP-5及LuDMP-6位于4号染色体,LuDMP-12~LuDMP-17均在15号染色体(图6)。对亚麻LuDMP基因进行复制分析(表4),结果显示有3对基因发生串联复制事件(LuDMP-4/LuDMP-5、LuDMP-13/LuDMP-14、LuDMP-16/LuDMP-17),8对基因发生片段复制(LuDMP-1/LuDMP-7、LuDMP-2/LuDMP-3、LuDMP-6/LuDMP-9、LuDMP-4/LuDMP-13、LuDMP-8/LuDMP-9、LuDMP-8/LuDMP-11、LuDMP-9/LuDMP-11、LuDMP-9/LuDMP-15)。通过判断Ka/Ks比值进行选择压力分析,由于LuDMP-6/LuDMP-9及LuDMP-9/LuDMP-15的Ks值为n.a.,导致Ka/Ks比值及分歧时间也为n.a.,n.a.表示这两对基因几乎可发生同义突变的位点都发生了同义突变,表明序列分歧度相当大,进化距离很远。其他发生复制的基因对Ka/Ks比值均小于1,表明这些LuDMP基因在进化过程中受到纯化选择。
2.5顺式作用元件分析
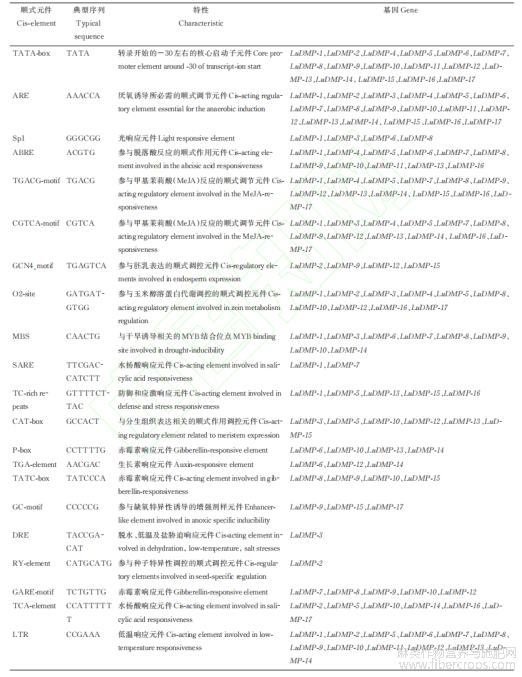
对亚麻LuDMP基因进行启动子分析,发现LuDMP基因启动子区域除含有TATA-box等基本作用元件之外,还含有多种与光响应、激素响应、非生物胁迫响应等相关的顺式作用元件(表5)。所有LuDMP基因都含有厌氧诱导必需的作用元件ARE;每个LuDMP基因至少含有一个或多个光响应元件,如Sp1等;除LuDMP-4和LuDMP-11外,其余15个LuDMP基因均有一个或多个植物激素响应元件,如参与生长素响应的TGA-element,参与赤霉素响应的TATC-box和GARE-motif,参与水杨酸反应的TCA-element,参与脱落酸响应的顺式作用元件ABRE,以及参与甲基茉莉酸(MeJA)响应的TGACG-motif和CGTCA-motif;除LuDMP-6、LuDMP-7、LuDMP-9、LuDMP-11、LuDMP-13、LuDMP-14和LuDMP-15外,其余LuDMP基因均含有参与玉米醇溶蛋白代谢调控的顺式调控元件O2-site;参与种子特异性调控的顺式调控元件RY-element只存在于LuDMP-2中;除LuDMP-3、LuDMP-4、LuDMP-15、LuDMP-16和LuDMP-17外,其他基因均含有低温响应元件LTR;参与脱水,低温,盐胁迫的顺式作用元件DRE只存在于LuDMP-3中;此外,LuDMP基因启动子区域还存在其他逆境胁迫响应元件,如参与防御和应激反应的TC-rich repeats、参与干旱诱导的MBS。
图6 LuDMP基因染色体定位
横线代表片段复制,方框代表串联复制。The horizontal line represents segmental duplication,and the box represents tandem duplication.Chr:染色体Chromosome.
表4 LuDMP基因复制分析
注:n.a.表示这两对基因几乎可发生同义突变的位点都发生了同义突变,表明序列分歧度相当大,进化距离很远。
表5 亚麻LuDMP启动子顺式元件分析
2.6LuDMP基因表达模式分析
利用陇亚10号和黑亚14号转录组数据分析LuDMP基因在不同品种、不同组织中的表达模式(图7),除LuDMP-9和LuDMP-15外,其他15个基因均具有表达量数据。由图7可知,大多数基因在两个亚麻品种中表现为组织特异性表达。其中LuDMP-2、LuDMP-4、LuDMP-5、LuDMP-6、LuDMP-8、LuDMP-11和LuDMP-16在两个品种果实中的表达量高于在茎中的表达量,尤其是LuDMP-4和LuDMP-5在油用品种陇亚10号中的表达量明显高于纤维品种黑亚14号,而LuDMP-1、LuDMP-3、LuDMP-7、LuDMP-10、LuDMP-12、LuDMP-13、LuDMP-14和LuDMP-17在两个品种茎中的表达量高于果实中的表达量,特别是LuDMP-10和LuDMP-12在黑亚14号中的表达量高于陇亚10号。
为进一步验证LuDMP-1/7的功能,本研究分析了该基因在陇亚15号不同发育时期的种子、未成熟花药及成熟花药中的表达模式。结果显示,LuDMP-1/7基因在各组织中均有表达,但在花药中的表达量明显高于其他组织,尤其在成熟花药中的表达量约为其他组织的6倍以上(图8)。此外,本研究发现LuDMP-1/7启动子存在激素响应及逆境胁迫相关元件,因此分析了LuDMP-1/7在不同胁迫处理下的表达模式。结果表明(图9),LuDMP-1/7在IAA处理后3h略有降低,6h时表达量上升了1.4倍左右,之后呈先降再升的趋势,处理48h后表达量趋于稳定;NAA处理后,LuDMP-1/7基因表达量呈先减后增再减的趋势,在24h时表达量达到最高,较对照上升1.3倍左右;GA3处理后,LuDMP-1/7基因表达量在12h时上升了约1.6倍,之后有所降低,在恢复48h时又呈上升的趋势;用NaCl处理后,不同时期基因表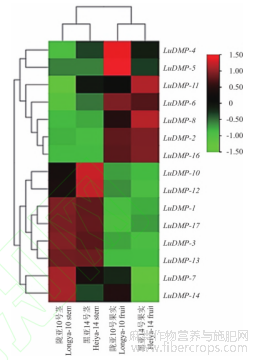 达均低于对照;PEG处理后6h表达量达到最高,约为对照的1.3倍;在低温(4℃)和高温(45℃)处理下,LuDMP-1/7基因表达量与对照相比有显著的提高,分别上升了约260倍和600倍。
达均低于对照;PEG处理后6h表达量达到最高,约为对照的1.3倍;在低温(4℃)和高温(45℃)处理下,LuDMP-1/7基因表达量与对照相比有显著的提高,分别上升了约260倍和600倍。
图7 LuDMP在不同组织中的表达量热图

图8 LuDMP-1/7基因在种子及花药中的表达模式
不同小写字母表示在0.05水平具有显著性差异。下同。Different small letters mean significant differences at the0.05level.The same below.
3讨论与结论
DMP蛋白是一种几乎只存在于绿色植物中的膜蛋白,目前已经在拟南芥[7]和棉花[10]中对其进行了系统分析,此外,在水稻(20个)、葡萄(6个)、玉米(15个)、大豆(9个)等多个物种中分别对其进行了鉴定[10]。本研究在亚麻基因组数据库中筛选获得了17个DMP基因,不均匀分布于9条染色体上。将亚麻DMP蛋白与大豆、拟南芥、葡萄、水稻、玉米等5个物种的DMP成员一起构建进化树,发现DMP蛋白可分为5个亚族,LuDMP家族成员在5个亚族中的数量差异很大,超过65%的LuDMP成员(11个)属于第III亚族。此外,DMP家族在各亚族中呈现出单子叶或双子叶特异性聚类模式,说明在单子叶植物和双子叶植物分化前DMP基因具有共同的祖先,这与Zhu等[10]的研究结果一致。串联复制和全基因组复制/片段复制是基因加倍、基因功能特异性和多样化的重要原因之一,也可能导致基因功能冗余[20,26-28]。DMP基因家族的高度扩张主要来源于几次全基因组复制,4个棉花种中只有3对基因发生串联复制,其余134对基因都经历了全基因组复制/片段复制[10]。LuDMP在亚麻进化过程中有3对基因发生串联复制,8对基因发生片段复制事件,片段复制是促进LuDMP基因家族扩张的主要原因,这源于亚麻的两次全基因组复制事件[18]。其中第III亚族的扩张最明显,有9对基因发生了复制事件,基因占比为73.3%,而亚族I和IV各自仅有1对基因发生了片段复制,说明这两个亚族在进化中更为保守。亚麻中,发生复制的LuDMP基因对Ka/Ks比值全部小于1,说明LuDMP基因在亚麻进化过程中经历了纯化选择。对LuDMP蛋白的亚细胞定位进行预测,发现大多数蛋白定位于细胞膜,与棉花DMP蛋白预测结果相似[10],拟南芥中DMP蛋白定位于液泡膜及内质网[7],这与DMP蛋白的功能一致。此外,LuDMP-1、LuDMP-7、LuDMP-11及LuDMP-14除了定位于细胞膜,还分别位于细胞核和叶绿体,这说明DMP蛋白还参与其他生物过程。基因结构分析显示,大多数LuDMP基因不含内含子,与棉花DMP基因家族研究结果相似[10],说明它们可能具有特殊的功能。保守基序分析结果表明,有4个motif存在于所有LuDMP蛋白,而其他motif只存在于特定的基因中,这些特有的motif是导致LuDMP蛋白功能分化的重要原因。亚麻LuDMP蛋白具有1个DMP结构域及2~5个跨膜结构域,与棉花中DMP蛋白的研究结果相同[10]。有研究表明玉米DMP蛋白第一个跨膜结构域发生单个氨基酸突变可诱导玉米单倍体的产生[8]。拟南芥中也有相似的研究结果,AtDMP8和AtDMP9蛋白第一个和第二个跨膜结构域发生突变可诱导拟南芥单倍体的产生[9]。因此,本研究推测与ZmDMP及AtDMP8和AtDMP9亲缘关系相近的亚麻LuDMP-1和LuDMP-7蛋白第一个和第二个跨膜结构域也具有相似的功能,可诱导亚麻单倍体的产生。由此可见,DMP蛋白跨膜结构域在植物育性中起重要作用。

图9 胁迫处理下LuDMP-1/7的表达模式
恢复48h:48hafter recovery.
与传统育种方法[29-30]相比,双单倍体育种可在两个世代内获得稳定的二倍体纯系,从而在很大程度上节约时间和成本。利用CRISPR技术对单倍体诱导相关基因PLA1/MATL和DMP进行编辑可诱导单倍体的产生,这种方法已经成功应用于玉米[31-33]、水稻[34]、小麦[35]、拟南芥[9]等多个物种。玉米单倍体的产生主要受ZmPLA1/MTL/NLD和ZmDMP的调控[8,31-33]。水稻中OsMATL基因突变可诱导水稻单倍体的产生[34],AtDMP8和AtDMP9突变可在拟南芥中诱导母本单倍体的产生[9]。在本研究中,亚麻LuDMP-1/7与AtDMP8、AtDMP9和ZmDMP具有很近的亲缘关系,位于同一进化分支,而且序列同源性达到67%以上,这说明它们可能具有相同的功能,参与植物的授粉和受精过程且能够诱导单倍体的产生。此外,利用qRT-PCR分析LuDMP-1/7基因在亚麻不同组织中的表达模式,发现它们在成熟花药中的表达量最高,进一步说明它们可作为亚麻单倍体诱导的候选基因,但还需进一步对其单倍体诱导功能进行研究。
基因表达通常受到其上游启动子区顺式元件的调控。这些位于基因转录起始位点上游非编码DNA中的顺式元件调节不同环境下基因的应激表达或组织特异性表达行为。因此,对参与LuDMP基因调控的顺式元件进行分析,有助于了解LuDMP基因的调控机制,并预测其潜在功能。在LuDMP基因启动子区存在许多与生长、逆境胁迫和植物激素响应相关的顺式元件,其中大部分顺式元件参与非生物胁迫和激素信号传导,这与棉花DMP基因顺式作用元件预测结果相似[10]。通过分析基因表达模式可初步预测基因功能。组织特异性分析结果表明,有7个LuDMP基因在果实中的表达量高于茎中的表达量,特别是LuDMP-4和LuDMP-5在油用品种中的表达量显著高于纤维品种,说明这些基因可能与果实发育或含油率相关;而其他8个基因在茎中的表达量较高,尤其是LuDMP-10和LuDMP-12在黑亚14号中的表达量高于陇亚10号,推测它们可能参与茎的生长发育(株高、纤维合成等)。此外,由于LuDMP-1/7基因启动子具有激素响应及非生物胁迫相关元件,推测其可能参与亚麻激素响应及逆境胁迫等生理过程。本研究中除NaCl胁迫外,其他处理下LuDMP-1/7基因表达量均有不同程度的提高,其中基因表达量对低温和高温的响应最明显,分别较对照提高了约260倍和600倍,表明该基因参与调控亚麻对激素和逆境胁迫的响应,但具体的功能还需进一步验证。
参考文献
[1]Yamada K,Osakabe Y,Mizoi J,et al. Functional analysis of an Arabidopsis thaliana abiotic stress-inducible facilitated diffusion transporter for monosaccharides. Journal of Biological Chemistry,2010,285(2):1138-1146.
[2]Xicluna J,Lacombe B,Dreyer I,et al. Increased functional diversity of plant K+ channels by preferential heteromerization of the shakerlike subunits AKT2 and KAT2. Journal of Biological Chemistry,2007,282(1):486-494.
[3]Chen Y,Heazlewood J. Organellar proteomic profiling to analyze membrane traf-ficking pathways. Trends in Plant Science,2020,26(3):299-300.
[4]Chen Y,Weckwerth W. Mass spectrometry untangles plant membrane protein sig-naling networks. Trends in Plant Science,2020,25(9):930-944.
[5]Mori T,Igawa T,Tamiya G,et al. Gamete attachment re-quires GEX2 for successful fertilization in Arabidopsis. Current Biology,2014,24(2):170-175.
[6]Kasaras A,Melzer M,Kunze R. Arabidopsis senescence-associated protein DMP1 is involved in membrane remodeling of the ER and tonoplast. BMC Plant Biology,2012,12:54.
[7]Kasaras A,Kunze R. Expression,localisation and phylogeny of a novel family of plant-specific membrane proteins. Plant Biology,2010,12(Supple 1):140-152.
[8]Zhong Y,Liu C X,Qi X L,et al. Mutation of ZmDMP enhances haploid induction in maize. Nature Plants,2019,5(6):575-580.
[9]Zhong Y,Chen B J,Mengran L,et al. A DMP-triggered in vivo maternal haploid induction system in the dicotyledonous Arabidopsis. Nature Plants,2020,6(5):466-467.
[10]Zhu S,Wang X,Chen W,et al. Cotton DMP gene family:Characterization,evolution,and expression profiles during development and stress. International Journal of Biological Macromolecules,2021,183(2):1257-1269.
[11]Wei Y H,Kong J P,Zhang Y H,et al. Development status,research and development trend and countermeasures of flax at home and abroad. Xinjiang Agricultural Sciences,2007,44(Supple 2):70-75.魏彦宏,孔建平,张彦红,等 . 国内外亚麻发展现状、研发趋势与对策 . 新疆农业科学,2007,44(增刊 2):70-75.
[12]Huis R,Hawkins S,Neutelings G. Selection of reference genes for quantitative gene expression normalization in flax(Linum usitatissimum L.). BMC Plant Biology,2010,10(1):71.
[13]Chytilova M,Mudronova D,Nemcova R,et al. Anti-inflammatory and immunoregulatory effects of flax-seed oil and Lactobacillus plantarum-BiocenolTM LP96 in gnotobiotic pigs challenged with enterotoxigenic Escherichia coli. Research in Veterinary Science,2013,95(1):103-109.
[14]Heller K,Sheng Q C,Guan F,et al. A comparative study between Europe and China in crop management of two types of flax:linseed and fibre flax. Industrial Crops and Products,2015,68:24-31.
[15]Liu Q,Talboot M,Llewellyn D J. Pectin methylesterase and pectin remodelling differ in the fibre walls of two Gossypium species with very different fibre properties. PLoS One,2013,8(6):e65131.
[16]Guo D,Du M,Zhou B Y,et al. Identification and bioinformatics analysis of CCT gene family in maize. Journal of Plant Genetic Resources,2019,20(4):1001-1010.郭栋,杜媚,周宝元,等 . 玉米 CCT 基因家族的鉴定与生物信息学分析 . 植物遗传资源学报,2019,20(4):1001-1010.
[17]Zheng L,Bai X T,Li H Y. Genome-wide identification and expression analysis of TCP gene family in Sorghum bicolor.Journal of Henan Agricultural Sciences,2019,48(10):30-36.郑玲,白雪婷,李会云 . 高粱 TCP 基因家族全基因组鉴定及表达分析 . 河南农业科学,2019,48(10):30-36.
[18]Yuan H,Guo W,Zhao L,et al. Genome-wide identification and expression analysis of the WRKY transcription factor family in flax(Linum usitatissimum L.). BMC Genomics,2021,22(1):375.
[19]Cai X X,Shen Y,Zhou W H,et al. Genome-wide identification and bioinformatics analysis of soybean CHX gene family.Genomics and Applied Biology,2018,37(12):5360-5369.才晓溪,沈阳,周伍红,等 . 大豆 CHX 基因家族全基因组鉴定与生物信息学分析 . 基因组学与应用生物学,2018,37(12):5360-5369.
[20]Wang X H,Xu N L,Yao M M,et al. Identification and evolution analysis of LBD gene family in Lophopyrum elongatum.Acta Agriculturae Boreali-Occidentalis Sinica,2022,31(2):202-216.王新华,许娜丽,姚明明,等 . 长穗偃麦草 LBD 基因家族的鉴定与进化分析 . 西北农业学报,2022,31(2):202-216.
[21]Huang C,Liang X M,Dai C,et al. Genome-wide identification and analysis of BnAPs gene family members in Brassica napus.Acta Agronomica Sinica,2022,48(3):597-607.黄成,梁晓梅,戴成,等 . 甘蓝型油菜 BnAPs 基因家族成员全基因组鉴定及分析 . 作物学报,2022,48(3):597-607.
[22]Chen D F,Wei X Q,Xu L,et al. Genome-wide identification and expression analysis of wax apple PG gene family. Journal of Fruit Science,2022,39(4):16.陈迪飞,魏秀清,许玲,等 . 莲雾 PG 基因家族全基因组鉴定及表达分析 . 果树学报,2022,39(4):16.
[23]Dang Z,Zhang J P,Wang L M,et al. Breeding technology report on new flax variety Longya-15. Plant Fiber Sciences in China,2020,42(4):145-149.党照,张建平,王利民,等 . 胡麻新品种陇亚 15 号选育技术报告 . 中国麻业科学,2020,42(4):145-149.
[24]Lynch M,Conery J S. The evolutionary fate and consequences of duplicate genes. Science,2000,290(5494):1151-1155.
[25]Tang Y K,Jia Y Y. Method of processing real time PCR data. Biotechnology,2008,18(3):89-91.唐永凯,贾永义 . 荧光定量 PCR 数据处理方法的探讨 . 生物技术,2008,18(3):89-91.
[26]Ren R,Wang H,Guo C,et al. Widespread whole genome duplications contribute to genome complexity and species diversity in angiosperms. Molecular Plant,2018,11(3):414-428.
[27]Zhang L,Jian H J,Yang B,et al. Identification and expression analysis of sucrose phosphate synthase(SPS)gene family members in Brassica napus. Acta Agronomica Sinica,2018,44(2):197-207.张 莉 ,荐 红 举 ,杨 博 ,等 . 甘 蓝 型 油 菜 蔗 糖 磷 酸 合 酶(SPS)基 因 家 族 成 员 鉴 定 及 表 达 分 析 . 作 物 学 报 ,2018,44(2):197-207.
[28]Soltis P S,Soltis D E. Ancient WGD events as drivers of key innovations in angiosperms. Current Opinion in Plant Biology,2016,30:159-165.
[29]Kelliher T,Starr D,Su X,et al. One-step genome editing of elite crop germplasm during haploid induction. Natur Biotechnology,2019,37(3):287-292.
[30]Jacquier N M A,Gilles L M,Pyott D E,et al. Puzzling out plant reproduction by haploid induction for innovations in plant breeding. Nature Plants,2020,6(6):610-619.
[31]Gilles L M,Khaled A,Laffaire J B,et al. Loss of pollen-specific phospholipase not like dad triggers gynogenesis in maize. The EMBO Journal,2017,36(6):707-717.
[32]Kelliher T,Starr D,Richbourg L,et al. MATRILINEAL,a sperm-specific phospholipase,triggers maize haploid induction.Nature,2017,542(7639):105-109.
[33]Liu C X,Li X,Meng D X,et al. A 4-bp insertion at ZmPLA1 encoding a putative phospholipase a generates haploid induction in maize. Molecular Plant,2017,10(3):520-522.
[34]Yao L,Zhang Y,Liu C,et al. OsMATL mutation induces haploid seed formation in indica rice. Nature Plants,2018,4(8):530-533.
[35]Liu H Y,Wang K,Jia Z M,et al. Efficient induction of haploid plants in wheat by editing of TaMTL using an optimized Agrobacterium-mediated CRISPR system. Journal of Experimental Botany,2020,71(4):1337-1349.
文章摘自:李雯,赵丽蓉,张建平,刘自刚,齐燕妮,李闻娟,谢亚萍.亚麻DMP基因家族的全基因组鉴定与分析[J/OL].草业学报:1-16[2023-01-31].http://kns.cnki.net/kcms/detail/62.1105.S.20221124.1332.008.html