摘 要: 亚麻(Linum usitatissimum L.)是重要的纤用作物,GRAS基因家族在植物生长发育中具有重要作用。研究利用生物信息学从亚麻全基因组数据库鉴定 LuGRAS 蛋白序列,经 SMART 和 Pfam-search 去除冗余序列后,通过 ExPASy ProParam、TBtools以及 MEGA7.0 等进行理化性质、染色体定位和系统进化等分析,结合转录组测序(RNA-seq)分析亚麻GRAS家族成员在不同组织、不同时期的表达模式,并用实时荧光定量PCR(qRT-PCR)技术检测部分具有显著差异的LuGRAS 基因在赤霉素处理后的表达情况。通过共表达网络对 LuGRAS 基因之间的关系进行分析。结果表明:从亚麻全基因组中鉴定出114个 LuGRAS 转录因子,根据亲缘关系远近分为10个亚族;亚麻GRAS蛋白氨基酸数介于135~1377个,分子量介于15.3~152.9kDa,平均等电点5.96,除LuGRAS114外,其他 LuGRAS 基因分布在15条染色体上;基于亚麻RNA-seq数据,可以将 LuGRAS 基因分为7组;通过qRT-PCR 验证8个表达量具有显著差异的 LuGRAS 基因,结果显示,有4个 LuGRAS 基因经赤霉素处理后不同时间段的表达量变化明显。LuGRAS50与LuGRAS94的表达模式表现出极高的正相关关系,LuGRAS38和LuGRAS50与LuGRAS94之间可能存在负调控关系。以上结果将为进一步研究 LuGRAS 基因的生物学功能及其在亚麻生长发育中的表达模式提供理论参考。
关键词:亚麻;GRAS基因家族;全基因组;生物信息学分析;表达分析
GRAS转录因子是植物生长发育中不可或缺的一类蛋白,是早期被发现并研究的转录因子家族之一。GRAS名称来源于该家族最先鉴定出的3个转录因子:GIBBERELLIC-ACIDINSENSITIVE(GAI)、REPRESSOR of GAI(RGA)和SCARECROW(SCR)[1]。GRAS蛋白的C末端区域的高度保守区域通常被称为GRAS结构域。典型的GRAS蛋白的长度为400~700个氨基酸,一般由5个典型的高度保守结构域组成:leucine-rich region I(LHRI)、VHIID、leucine-rich region II(LHRII)、PFYRE和SWA。其中,VHIID结构域是GRAS蛋白的核心结构域,几乎存在于所有的GRAS蛋白中,并且参与蛋白与DNA之间以及蛋白与蛋白之间的相互作用。GRAS蛋白的N末端是一个高度可变的区域,包含两个高度保守的蛋白结构,DELLA和TVHYNP。由于具有不同的N末端基序,GRAS蛋白可以与不同靶蛋白结合,从而增加了基因功能的多样性[1-3]。目前,在拟南芥、水稻和玉米等多个物种中分别鉴定出GRAS蛋白[4-6]。根据氨基酸序列的保守性及其功能特性的不同,GRAS蛋白家族被细分为多个亚家族,这些亚家族分别依据其代表性成员的名称或共有的关键结构域来命名,具体包括SCL3、PAT1、SHR、LISCL、DELLA、SCR、LAS、DLT、HAM和SCL4/7等[7]。不同的亚族参与的生理生化反应各不相同。赤霉素通过调节DELLA-SPL9复合物活性抑制拟南芥腋芽的形成[4]。类似地,LAM也被发现能够调节草莓腋芽形成[8]。PAT1和CONSTANS-Like13能够协同调控芥菜的分枝和开花[9]。
亚麻(Linumu sitatissimum L.)是亚麻科(Linaceae)亚麻属(Linum)一年生草本植物,是我国重要的经济作物之一[10]。研究[11]表明,向亚麻叶片喷施赤霉素后,能够显著提高亚麻的株高、工艺长度和原茎产量,从而影响亚麻的生长发育。在亚麻的快速生长期中,存在一个关键的生长点,被称为“snap point(sp)”,该点通常距离植株顶部6~8cm。在sp点之上,韧皮纤维细胞处于快速伸长阶段,这对于纤维亚麻的生长发育至关重要[12]。本研究以亚麻RNA-seq数据为参考,对亚麻中的GRAS成员进行了系统的生物信息学分析,为今后分析其功能提供依据。
1 材料与方法
1.1 植物材料
试验于2023年10—12月在南通大学啬园校区气候室进行。供试材料为黑亚16号,来源于黑龙江省农业科学院经济作物研究所,是高纤亚麻品种。取直径20cm、高22cm的塑料花盆,种植在花土和蛭石质量比为3:1的土壤中,每盆播种10~15粒,出苗一周后进行间苗,每盆留苗5株。在25℃的温室中生长,光照16h,暗处理8h,相对湿度为70%,持续30d((从枞形期到快速生长期)。在该环境下,当亚麻植株进入快速生长期时,使用浓度为100mg/L和300mg/L的赤霉素溶液,均匀喷洒在亚麻叶片上。在处理后的不同时间点(0、3、6、12、24、48h)收集亚麻主茎sp点上部(距离茎尖1~3cm)的茎组织样品。同时,生长相同时间的植株仅进行喷水处理作为对照。所有样品进行3个生物学重复的采集。采集后,立即使用液氮进行速冻,并将样品保存在—80℃的冰箱中。
1.2 亚麻 GRAS 基因家族成员的鉴定、理化性质
从 figshare 网站下载亚麻基因组蛋白文件(https://figshare.com/articles/dataset/Annotation_files_for_Longya-10_genome/13614311)。从pfam(http://pfam.xfam.org/)中下载 LuGRAS基因家族保守结构域(PF03514)的隐马尔可夫模型(HMM),对检索到的 LuGRAS 序列进行BLAST比对,参考染色体位置,利用pfam-search(http://pfam.xfam.org/search#tabview=tab1)与Smart(http://smart.embl.de/)对 LuGRAS 蛋白序列进行验证,最终保留含有 LuGRAS 结构域的蛋白。得到 LuGRAS 基因家族成员,按其在染色体中的位置依次命名。从植物TFDB(http://planttfdb.cbi.pku.edu.cn/)中获得拟南芥和水稻GRAS基因的蛋白质序列。利用在线软件ExPASy(https//web.expasy.org/protparam/)计算 LuGRAS 家族成员蛋白质的理化性质。
1.3 亚麻GRAS蛋白的系统发育分析
使用 Clustal X(2.0版)的默认参数对亚麻与拟南芥 GRAS 蛋白进行多序列比。使用 MEGA(7.0版) 和1000个 bootstrap 重复,采用 Neighbor Joining(NJ)方法制作亚麻和拟南芥的系统发育进化树。
1.4 保守基序和基因结构
使用基因结构显示服务器(GSDS:http://gsds.cbi.pku.edu.cn)获得基于CDS和基因序列相对应的内含子和外显子的基因结构。利用MEME(https://meme-suite.org/meme/tools/meme)(设定基序数目为3个)对亚麻的114个 LuGRAS 蛋白序列进行保守基序分析。使用 TBtools(https://www.tbtools.com/)将基因结构、保守基序和亚麻系统发育树绘制成图。
1.5 亚麻GRAS染色体定位及共线性分析
从 figshare 网站下载亚麻基因组注释gff3文件(https://figshare.com/articles/dataset/Annotation_files_for_Longya-10_genome/13614311)。利用TBtools(https://www.tbtools.com/)绘制染色体定位图。通过 TBtools 的(“高级circus”模块分析了 LuGRAS 家族114个成员的种内共线性,并将其可视化。利用(“Dual system plot for MCscanx”模块,对水稻、拟南芥和亚麻的GRAS基因的种间共线性进行了分析和可视化。
1.6 亚麻GRAS基因家族的表达模式及qRT-PCR分析
选取3个时期的茎组织、花后不同时期的种子、花瓣的RNA-seq数据通过微生信在线软件(https://www.bioinformatics.com.cn/)绘制成 LuGRAS 基因家族的表达热图,RNA-seq数据来源于本试验室先前测得的试验数据(未发表),转录丰度表示为每千碱基片段的外显子模型每百万映射读取(FPKM)值。使用联川生物在线软件(https://www.omicstudio.cn/tool)绘制 LuGRAS 基因互作网络图。按照说明书,采用 RNAprep Pure 多糖多酚植物总RNA提取试剂盒(天根)提取总RNA,使用 FastKing cDNA 第一链合成试剂盒(去基因组)(天根)反转录为cDNA。用PrimerPremier5.0软件对选定的 LuGRAS 基因进行特异性引物设计,内参基因为LuGAPDH,引物信息见表1。按照天根 SuperReal PreMix Plus(SYBR Green)说明书中的指导,配置20μL的反应体系进行qRT-PCR,并在7500 Real Time PCR System(BIO-RAD,USA)上进行操作。反应程序为95℃15min;95℃10s,60℃32s,循环40次,根据2-ΔΔCt方法计算 LuGRAS 相对表达水平。使用 GraphPad Prism9 软件进行数据分析并作图。
表1 qRT-PCR引物信息
2 结果与分析
2.1 亚麻GRAS家族成员的全基因组鉴定
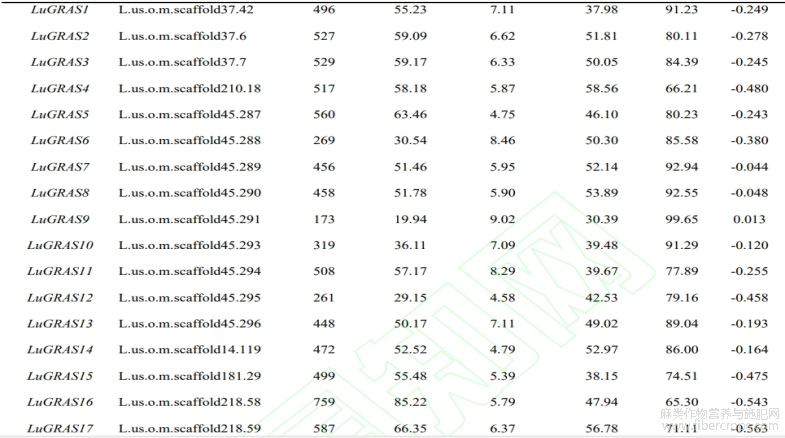
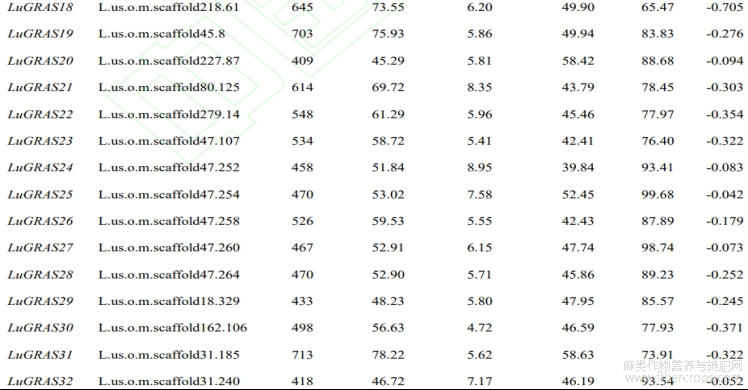
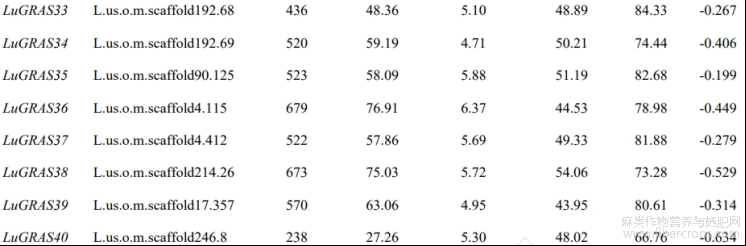
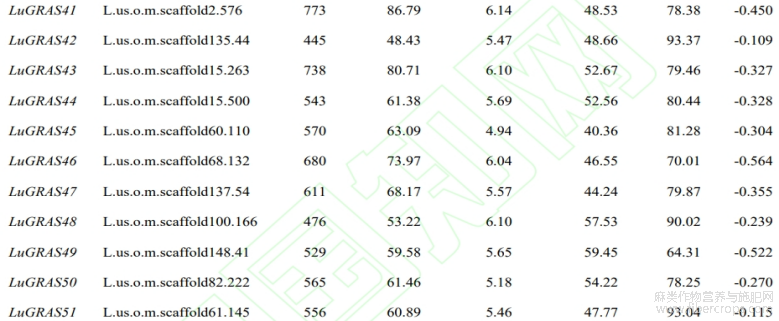
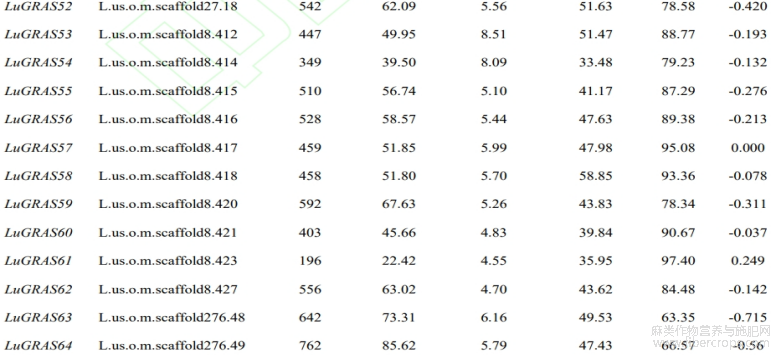
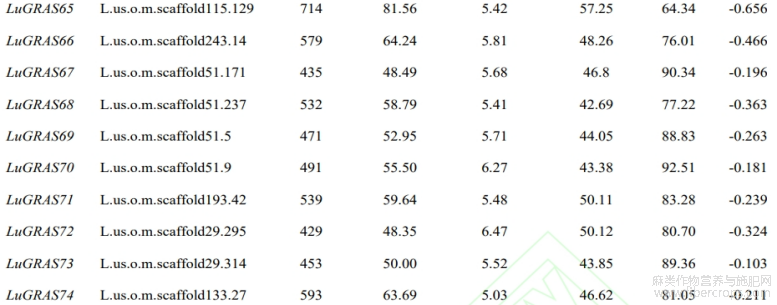
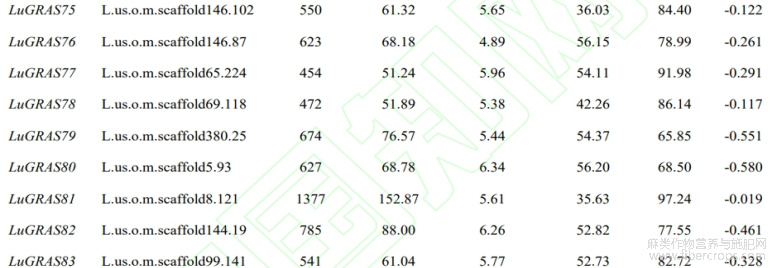
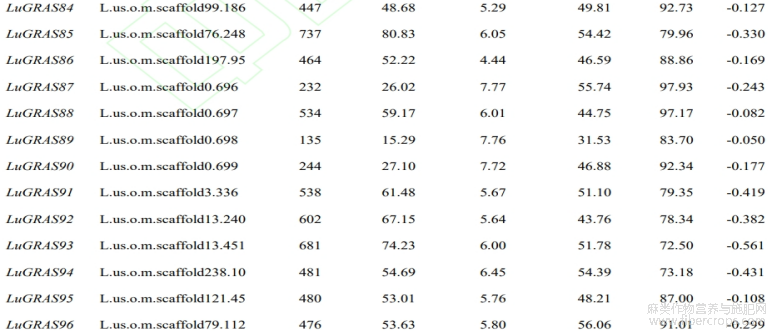
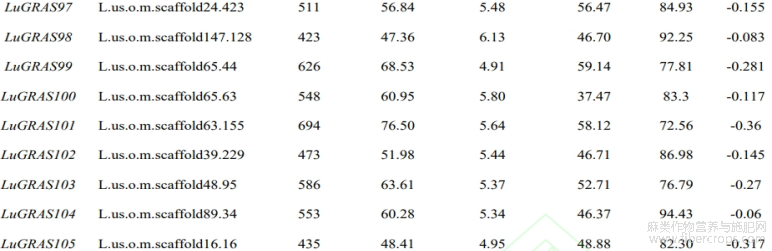
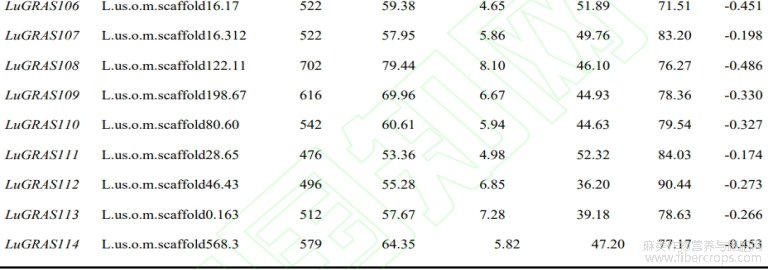
从亚麻基因组中鉴定出114个 LuGRAS 基因,根据其在染色体中的位置顺序将它们重命名为 LuGRAS1~LuGRAS114 (表2)。114个 LuGRAS 蛋白的氨基酸数为135个(LuGRAS89)~1377(LuGRAS81)个。 LuGRAS 蛋白的等电点为4.44(LuGRAS86)~9.02(LuGRAS9),平均等电点为5.96,表明大多 LuGRAS 蛋白是酸性的。LuGRAS蛋白的平均疏水系数都小于0,表明这些蛋白质是亲水的。
表2 亚麻GRAS基因家族的基本理化性质
2.2 系统发育分析
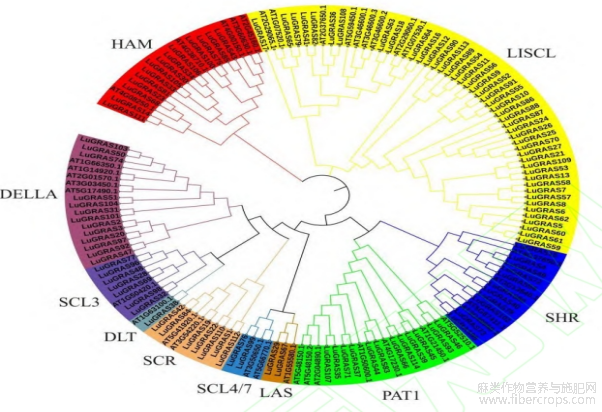
将114个 LuGRAS 蛋白和37个 AtGRAS 蛋白的氨基酸序列构建系统发育树(图1)。根据与 AtGRAS 蛋白的同源性,将114个 LuGRAS 蛋白分为10个亚家族:LAS、SCL4/7、HAM、SCR、DLT、SCL3、DELLA、PAT1、SHR和LISCL。LISCL亚家族成员数量最多,共有45个 LuGRAS 蛋白。SCL4/7和LAS亚家族包含两个成员,SCR和SCL3中包含7个成员,在HAM、DELLA、PAT1、SHR和DLT中分别有14、13、12、11和1个 LuGRAS 成员。拟南芥共有37个 AtGRAS 蛋白,分布在LISCL亚家族的成员最多,共9个,最少的LAS、DLT和SCL3亚家族,只有1个成员。说明亚麻GRAS蛋白与拟南芥GRAS蛋白在不同亚族分布上存在差异。
图1 亚麻(Lu)、拟南芥(At)系统发育树
2.3 亚麻GRAS基因家族的基因结构和基序组成
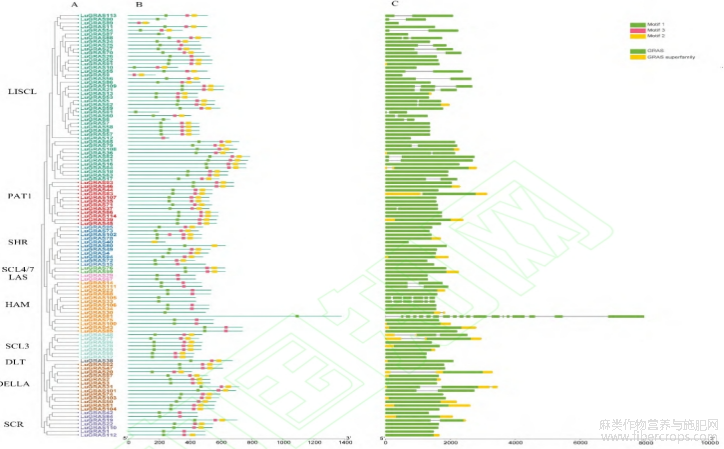
为了探究 LuGRAS 基因的进化历程,通过 cDNA 与基因组序列比对揭示了其外显子/内含子结构(图2C)。在114个 LuGRAS 基因中,78个(68.42%)无内含子,而36个(31.58%)含内含子,其中多数仅含1~2个,仅少数(2个)含5个。此外,LuGRAS81含18个内含子,暗示其可能经历了独特的进化历程。为了进一步研究 LuGRAS 蛋白的特征区域,使用在线MEME分析了114种 LuGRAS 蛋白质的基序,共发现了3个不同的保守基序(图2B),86个 LuGRAS 蛋白(75.44%)含有基序1、基序2和基序3。有9个 LuGRAS 蛋白只含有基序1,LuGRAS40只含有基序2,LuGRAS12只含有基序3。结合系统进化分类来看,同一亚家族的成员具有相似的基因结构和基序组成。例如,LAS和HAM亚族大部分只含有基序1和基序2,不含基序3。
图2 亚麻GRAS蛋白系统进化树(A)、蛋白保守基序(B)、基因结构(C)
2.4 亚麻GRAS基因的染色体分布和同源性分析
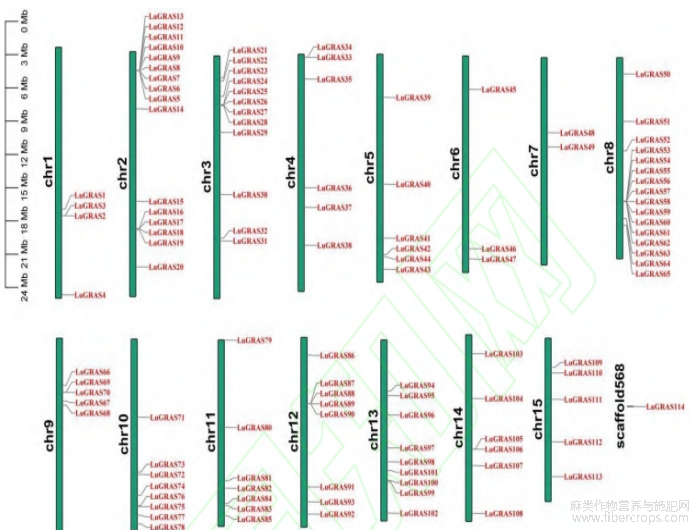
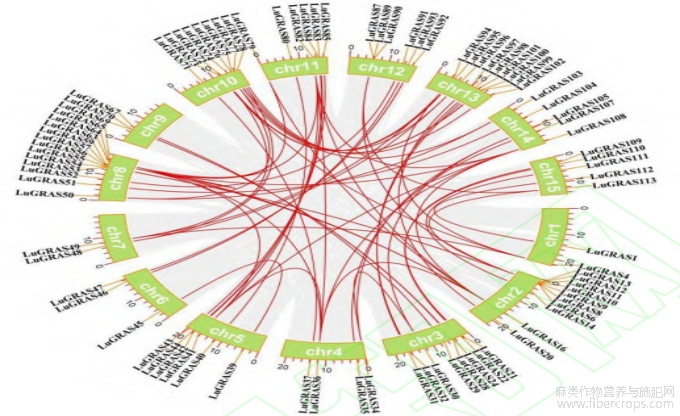
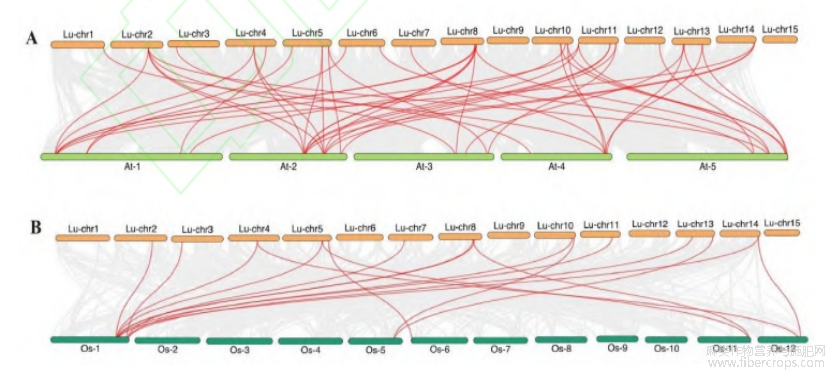
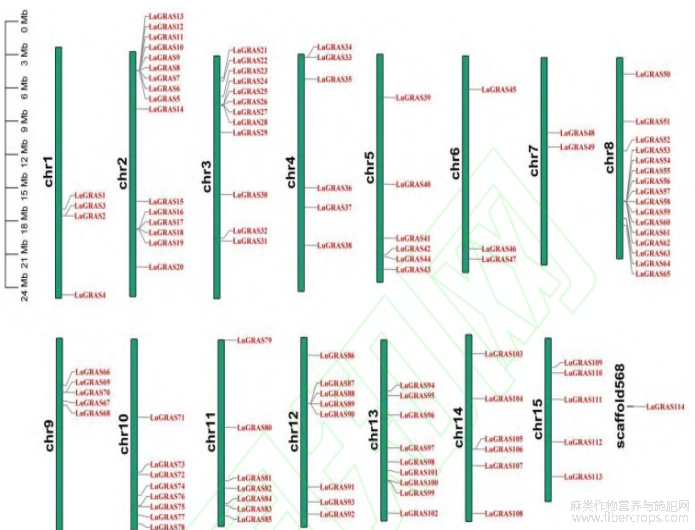
除了位于未定位在染色体上的1个基因外,其余113个 LuGRAS 基因在15条染色体中分布不均(图3)。Chr2和Chr8含有 LuGRAS 基因(16个基因,约14.04%)数量最多,其次是Chr3上的12个基因,以及Chr13上的9个基因。除了Chr1、Ch5、Chr6、Chr7、Chr10和Chr15,在其他染色体中均存在串联重复序列。例如Chr1的LuGRAS2/LuGRAS3、Chr4的LuGRAS33/LuGRAS34。此外,在染色体之间检测到70对节段重复基因(图4),其中Chr10有11对节段重复基因,Chr11 有13对节段重复基因。这些结果表明,串联和节段重复可能是 LuGRAS 家族进化的主要驱动力。为了进一步推断 LuGRAS 基因的进化关系,对亚麻与双子叶植物(拟南芥)和单子叶植物(水稻)的GRAS基因家族成员进行研究(图5),结果发现,亚麻与拟南芥之间的同源GRAS基因对为44对(图5A),与水稻之间的同源GRAS基因对为18对(图5B),说明双子叶植物之间的亲缘关系更近。LuGRAS16、LuGRA36、LuGRAS41、LuGRAS43、LuGRAS63、LuGRAS64、LuGRAS65、LuGRAS82和LuGRAS108在拟南芥和水稻中都具有同基因座,表明这些在基因进化中发挥了重要作用。
图3 亚麻GRAS家族成员染色体定位分析
图4 亚麻 GRAS 基因的种内共线图
图5 亚麻(Lu)与拟南芥(At)、水稻(Os)GRAS 基因种间共线图
2.5 亚麻GRAS基因在不同组织不同时期的表达分析
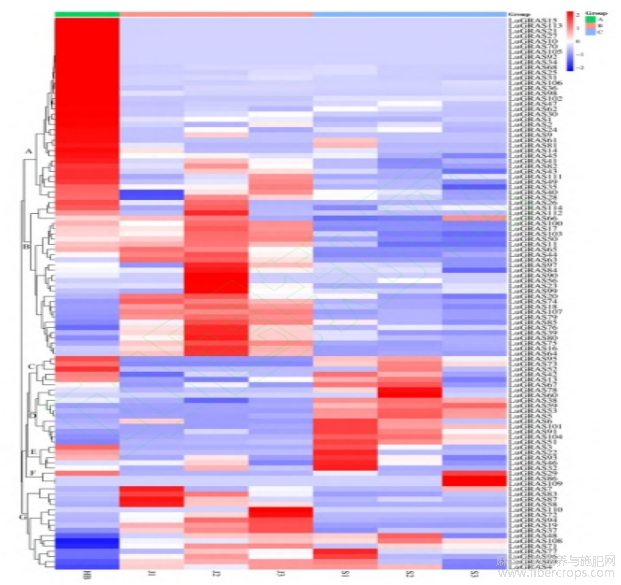
基于 RNA-seq 数据绘制 LuGRAS 基因表达热图(图6),分析 LuGRAS 基因在不同组织不同时期的表达量变化模式。其中8个 LuGRAS 基因在花瓣、茎和种子中均没有表达,这可能意味着它们在亚麻中存在组织特异性表达的现象。而其中106个 LuGRAS 基因则显示出了特定的组织表达特性。通过聚类分析,这些表达的 LuGRAS 基因被分为了7个不同的组(A至G)。A组基因主要在花瓣中表达量较高,而在茎和叶组织中的表达量则较低或几乎不表达。B组包含30个 LuGRAS 基因,这些基因在J2时期表达量较高。C组中的6个 LuGRAS 基因,其中5个在花瓣中表达,并且这些基因在S2时期的表达量相对较高。D组中的12个 LuGRAS 基因中,有7个在S1、S2、S3这3个时期均有表达。表明D组基因可能与种子的生长发育密切相关。E组的5个 LuGRAS 基因在S1时期的表达量尤为突出。F组中的3个LuGRAS 基因在S3时期的表达量较高。最后,G组中的16个 LuGRAS 基因在花瓣中的表达量较低,但其中有4个在J1时期高表达,2个在J3时期高表达。以上结果表明 LuGRAS 基因可能在亚麻生长发育过程中发挥着不同的作用。
注:HB代表花瓣,J1、J2、J3分别代表茎的快速生长期、花期、工艺成熟期,S1、S2、S3分别代表花后10、20、30d的种子。
图6 亚麻 GRAS 基因在不同组织不同时期的表达热图
2.6 亚麻 GRAS 基因在不同浓度赤霉素处理后的表达模式
在深入研究亚麻和拟南芥的GRAS家族成员间的进化关系后,挑选了8个具有潜在研究价值的 LuGRAS 基因,采用 qRT-PCR 技术进行表达模式分析。结果表明,在赤霉素的作用下, LuGRAS 基因的表达呈现出复杂而多变的模式。具体来说,在100mg/mL的赤霉素浓度下,LuGRAS49、LuGRAS94和LuGRAS99在3h时显著上调,表明这些基因对赤霉素的响应较为迅速。此外,LuGRAS36、LuGRAS49、LuGRAS50、LuGRAS94和LuGRAS99在48h时也呈现上调趋势,表明这些基因的表达可能受到赤霉素的长时间影响。当赤霉素浓度提升至300mg/mL时, LuGRAS 基因的表达变化更为复杂。LuGRAS36、LuGRAS50、LuGRAS94、LuGRAS97和 LuGRAS99 基因在初始阶段表现出上调趋势,随后又出现下调,最终再次上调,这种先升后降再升的模式可能反映了这些基因在应对高浓度赤霉素时的复杂调控机制。尤其值得关注的是LuGRAS50在3h显著上调,LuGRAS36、LuGRAS49、LuGRAS94和LuGRAS99 在12h显著上调,这些时间点可能是这些基因响应赤霉素的关键阶段。
图7 在100mg/mL和300mg/mL赤霉素处理后不同时间段亚麻 GRAS 基因的表达
2.7 亚麻 GRAS 基因共表达网络分析
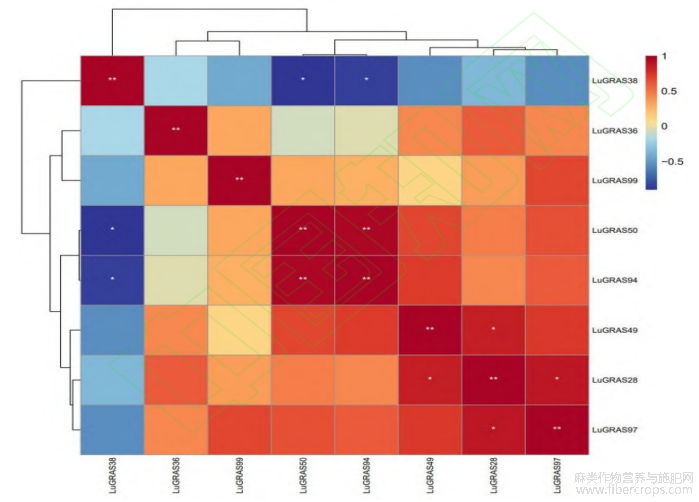
利用 RNA-seq 数据对选取的8个 LuGRAS 基因的表达水平进行了相关分析(图8)。结果表明,LuGRAS50 与 LuGRAS94 的表达模式显示出极高的正相关性,它们之间的相关系数高达0.96,表明这两个基因在表达调控上可能存在紧密的联系。LuGRAS28 与 LuGRAS97 相关系数为0.89。同样地,LuGRAS28 与 LuGRAS49相关系数达到0.85。进一步分析发现,LuGRAS38 与 LuGRAS50 之间的表达模式呈现出明显的负相关性,其相关系数低至—0.89;LuGRAS38 与 LuGRAS94 的负相关系数为—0.86。以上结果表明,LuGRAS38 可能与 LuGRAS50 及 LuGRAS94在功能或调控机制上存在某种程度的拮抗作用,为后续的功能验证及机制探索提供了重要线索。
图8 亚麻 GRAS 基因表达的相关性图
3 讨论
GRAS 基因家族在植物生长发育、光敏信号传导和胁迫反应过程中发挥着重要作用[13-14],目前,已经在多种植物中对该基因家族进行了研究,但在亚麻中还尚未见研究报道。本研究对亚麻GRAS基因家族进行了全基因组分析,共鉴定出114个家族成员,分为10个亚家族。番茄53个[15]、板栗48个[16],均少于本研究。大豆[17]、小麦[18]中分别鉴定到117个、188个GRAS成员。这些物种中GRAS成员个数均多于本研究亚麻GRAS基因个数。分布同一亚家族或分支的GRAS基因可能具有相似的功能。AtSHR 和 AtSCR 蛋白在根和茎的径向发育中扮演重要角色[19],而与其划分在同一组内同源性较高的 LuGRAS 基因也可能存在类似功能。
外显子-内含子组织分析显示,78个(68.42%) LuGRAS 基因不含内含子(图2),而在荔枝[20]、番茄[15]和谷子[21]中GRAS基因的比例分别为81.3%、77.4%和64.91%。植物中无内含子基因在GRAS基因家族中所占的比例较高,说明GRAS蛋白的进化关系密切。节段和串联重复都是GRAS基因家族扩增的重要贡献者。除了串联重复外,相当多的LuGRAS家族成员来自节段重复事件。与单子叶植物相比,双子叶植物中的GRAS成员与已鉴定的LuGRAS成员表现出更好的同源性(图5)。因此推测GRAS成员之间的同基因相关性可能与物种的进化分化有关。总之,不同物种同基因GRAS成员的交叉可能有助于对GRAS进化进行相关探索。
基因的表达模式在一定程度上决定着基因功能。GRAS基因家族功能十分广泛,水稻中 DLT 亚家族的同源基因 OsGRAS29 通过调节油菜素内酯信号来影响水稻的高度和分蘖[22]。SCL3 能够减弱根内皮层中的 DELLA 抑制因子,并整合和维持功能性赤霉素通路以调节根的发育过程[23],并且 SCL3 和 DELLA 在控制下游赤霉素反应和上游赤霉素生物合成基因方面相互拮抗[24]。结合 RNA-seq 数据,114个 LuGRAS 基因根据表达变化可以分为7个组,其中B组和G组中的 LuGRAS 基因主要在茎的发育阶段表达。根据系统发育分析,挑选出8个具有潜在研究价值的 LuGRAS 基因通过 qRT-PCR进一步验证,结果发现,这些基因能够不同程度地响应赤霉素反应,有4个基因(LuGRAS49、LuGRAS50、LuGRAS94和LuGRAS99)表达量变化明显,说明 LuGRAS 基因可能参与亚麻对赤霉素的响应。结合互作网络预测和基因表达相关性分析的结果,观察到LuGRAS38可能与LuGRAS50及LuGRAS94 存在一定互作关系。
4 结论
本研究基于亚麻全基因组数据共鉴定出114个亚麻GRAS基因成员,分为10个亚家族。不同亚家族间具有不同的蛋白结构及表达模式。LuGRAS 基因在花、茎、种子中的表达量差异较大,表明 LuGRAS 基因的表达具有组织特异性。有4个LuGRAS 基因经赤霉素处理后不同时间段的表达量变化明显。特别值得关注的是,LuGRAS38、LuGRAS50 和 LuGRAS94 均表现出对赤霉素的积极响应,并且它们之间的相关系数较高,表明这三个基因可能参与亚麻对赤霉素的响应从而调控亚麻的生长发育,但具体的功能还需进一步验证。本研究为解析亚麻 LuGRAS 基因响应赤霉素的分子机制提供了科学依据,并为通过基因工程进一步研究 LuGRAS 基因在亚麻生长发育中的功能提供了有价值的基础。
参考文献
[1] BOLLE C. The role of GRAS proteins in plant signal transduction and development[J]. Planta, 2004, 218(5): 683-692.
[2] 殷龙飞,张中保,于荣,等.植物GRAS家族蛋白结构和功能的研究进展[J].分子植物育种,2019,17(19):6323-6331.
[3] Hakoshima T. Structural basis of the specific interactions of GRAS family proteins[J]. FEBS Letters, 2018, 592(4): 489-501.
[4] ZHANG Q Q, WANG J G, WANG L Y, et al. Gibberellin repression of axillary bud formation in Arabidopsis by modulation of DELLA-SPL9 complex activity[J]. Journal of Integrative Plant Biology, 2020, 62(4): 421-432.
[5] DUTTA M, SAHA A, MOIN M, et al. Genome-wide identification, transcript profiling and bioinformatic analyses of GRAS transcription factor genes in rice[J]. Frontiers in Plant Science, 2021, 12: 777285.
[6]GUO Y Y, WU H Y, LI X, et al. Identification and expression of GRAS family genes in maize (Zea mays L.)[J]. PLoS One, 2017, 12(9): e0185418.
[7] SUN X L, XUE B, JONES W T, et al. A functionally required unfoldome from the plant Kingdom: intrinsically disordered Nterminal domains of GRAS proteins are involved in molecular recognition during plant development[J]. Plant Molecular Biology, 2011, 77(3): 205-223.
[8] FENG J, CHENG L C, ZHU Z Y, et al.GRAS transcription factor LOSSOF AXILLARY MERISTEMS is essential for stamen and runner formationin wild strawberry[J]. Plant Physiology, 2021, 186(4): 1970-1984.
[9] MUNTHA S T, ZHANG L L, ZHOU Y F, et al. Phytochrome A signal transduction 1 and CONSTANS-LIKE 13 coordinately orchestrate shoot branching and flowering in leafy Brassica juncea[J]. Plant Biotechnology Journal, 2019, 17(7): 1333-1343.
[10] 伍叶娜,潘根,姜慧,等.亚麻全基因组关联分析研究进展[J].中国麻业科学,2022,44(2):109-118.
[11] 李彩凤,赵东升.赤霉素对亚麻过氧化物酶及主要农艺性状的影响[J].作物杂志,2009(4):38-41.
[12] Gorshkova T A, Ageeva M, Chemikosova S, et al. Tissue-specific processes during cell wall formation in flax fiber[J]. Plant Biosystems-an International Journal Dealing with All Aspects of Plant Biology,2005, 139(1): 88-92.
[13] WASEEM M, NKURIKIYIMFURA O, NIYITANGA S, et al. GRAS transcription factors emerging regulator in plants growth, development, and multiple stresses[J]. Molecular Biology Reports, 2022, 49(10): 9673-9685.
[14] Neves C, Ribeiro B, Amaro R, et al. Network of GRAS transcription factors in plant development, fruit ripening and stress responses[J]. Horticulture Research, 2023, 10(12): uhad220.
[15] HUANG W, XIAN Z Q, KANG X, et al. Genome-wide identification, phylogeny and expression analysis of GRAS gene family in tomato[J]. BMC Plant Biology, 2015, 15(1): 209.
[16] YU L Y, HUI C, HUANG R M, et al. Genome-wide identification, evolution and transcriptome analysis of GRAS gene family in Chinese chestnut (Castanea mollissima)[J]. Frontiers in Genetics, 2023, 13: 1080759.
[17] WANG L, DING X L, GAO Y Q, et al. Genome-wide identification andcharacterization of GRAS genes in soybean (Glycine max)[J]. BMC Plant Biology, 2020, 20(1): 415.
[18] LIU Y F, WANG W. Characterization of the GRAS gene family reveals their contribution to the high adaptability of wheat[J]. PeerJ, 2021, 9: e10811.
[19] CUI H C, Levesque M P, Vernoux T, et al. An evolutionarily conserved mechanism delimiting SHR movement defines a single layer of endodermis in plants[J]. Science, 2007, 316(5823): 421-425.
[20] CHEN J W, YAN Q, LI J W, et al. The GRAS gene family and its roles in seed development in Litchi (Litchi chinensis Sonn)[J]. BMC Plant Biology, 2021, 21(1): 423.
[21] FAN Y, WEI X B, LAI D L, et al. Genome-wide investigation of theGRAS transcription factor family in foxtail millet (Setaria italica L.)[J]. BMC Plant Biology, 2021, 21(1): 508.
[22] TONG H N, JIN Y, LIU W B, et al. DWARF AND LOW-TILLERING, a new member of the GRAS family, plays positive roles in brassinosteroid signaling in rice[J]. The Plant Journal, 2009, 58(5): 803-816.
[23] Heo J O, Chang K S, Kim I A, et al. Funneling of gibberellin signaling by the GRAS transcription regulator SCARECROW-LIKE 3 in the Arabidopsisroot[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(5): 2166- 2171.
[24] ZHANG Z L, Ogawa M, Fleet C M, et al. SCARECROW-LIKE 3 promotes gibberellin signaling by antagonizing master growth repressor DELLA in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(5): 2160-2165.
文章摘自:李静,华鑫涛,杨芸,鲁若妍,刘畅,孙健,戴志刚,谢冬微.亚麻GRAS基因家族的全基因组分析及其对外源赤霉素的响应研究[J/OL].中国麻业科学.https://link.cnki.net/urlid/43.1467.S.20250123.1749.002。