摘 要:目的TCP基因家族在植物叶片发育、侧枝形成、花器官形成、植物激素合成与信号转导等生理过程中发挥着关键的作用。对工业大麻Cannabis sativa TCP基因进行全基因组鉴定及表达分析为工业大麻TCP基因家族的功能分析提供科学参考。方法 基于已发布的工业大麻基因组,利用生物信息学方法对工业大麻TCP基因家族成员进行鉴定并对其理化性质、基因结构、以及表达模式进行分析。结果 共鉴定出17个工业大麻TCP基因(Cs TCP1~Cs TCP17)分为2类3个亚科(PCF、CIN和CYC/TB1),编码的氨基酸长度在163~585 aa,蛋白质相对分子质量为18 310~64 070,蛋白等电点介于4.99~9.36,亚细胞定位大部分为细胞核。共线性进化分析发现,工业大麻与拟南芥、番茄、水稻分别存在9、20和6对同源基因。顺式元件预测结果表示光响应元件和脱落酸元件在TCP基因启动子区域分布广泛。基因表达分析结果表明,工业大麻TCP基因的表达存在组织特异性,大部分在叶和花中高表达。结论 为深入研究工业大麻TCP基因家族的功能奠定了基础,进一步阐明了TCP基因可能参与工业大麻生长发育的各个阶段,并对工业大麻的药用资源发展提供理论基础。
关键词:生物信息学;工业大麻;TCP基因家族;鉴定;功能分析
大麻Cannabis sativa L.是二倍体(2n=20)一年生草本植物隶属于大麻科大麻属,分布于世界各地[1,2]。大麻在历史上被广泛应用于药用、食品和纤维等领域,具有悠久的历史。根据中医传统,大麻果实被称为“火麻仁”,最早见于《神农本草经》,它被认为有润肠通便和滋养作用,并对消化系统、心血管系统和中枢神经系统具有一定的药理作用[3]。因此,它可以用于治疗肠燥便秘、风湿性疼痛等疾病。此外,大麻还富含90多种不饱和脂肪酸,具备药食两用的功能[4,5]。大麻的根部常被用于治疗炎症和疼痛[6]。而雌性大麻花序表面的分泌型腺毛则富含大麻素[7],这些物质具有药用或娱乐特性。值得一提的是,大麻还富含植物纤维,其茎材可用于纤维生产,是制造高品质纺织品和造纸的优质原料[8]。在数千年的人工驯化和野生栽培过程中,种植者培育出多个品种的大麻,主要用于药物和纤维制品[9]。总的来说,大麻在不同部位的应用广泛且多样化,为人类提供了重要的药用价值和经济效益。
TCP转录因子是植物特有的蛋白家族,最早于20世纪90年代末被描述[10,11]。TCP是最早发现的3个物种中的基因名称的首字母缩写:来自玉米Zea mays的TB1(大刍草BRANCHED 1)[12],来自金鱼草(金鱼草)的环形棘皮纲(CYC)[13],以及来自水稻Oryza sativa L.的Proliferating cell factor1和2(PCF1和2)[11]。TCP基因在植物界中高度保守,含有一个保守的非典型碱性-螺旋-环-螺旋(b HLH)保守结构域,由58~62个氨基酸残基组成,参与DNA结合、蛋白质与蛋白质的相互作用和蛋白质的核定位[11]。根据TCP结构域的序列相似度不同,可以将其分为2类:I类(也称为PCF类[14]或TCP-P类[15])和II类(也称为TCP-C类)。与II类蛋白质相比,I类蛋白质的基本结构域中有4个氨基酸的缺失,而II类也可以根据TCP结构域内的差异进一步细分为2个分支[11]:CIN和CYC/TB1。有研究表明,I类TCP基因主要参与促进细胞增殖和植物生长,如水稻中的PCF1/PCF2和拟南芥中的TCP20[16,17],而CYC/TB1进化枝(或ECE进化枝)包括主要参与腋生分生组织发育(产生花和侧枝)的基因,如斑叶草中的Cp CYC1/2[18]可以和玉米中的BAD1[19]可以调控叶夹角。
TCP基因家族作为植物特有的转录因子,广泛参与植物分支生长发育调控,已在拟南芥[20]、烟草[21]等模式植物以及杜仲[22]、薏苡[23]、人参[24]和藜麦[25]等药用植物中被鉴定。以生产火麻仁为目标的大麻品种需要更多分枝及花序形成,以达到高产需求。TCP是植物分枝发育和花发育重要调控因子,因此,研究大麻TCP具有重要意义。然而关于大麻TCP基因家族的鉴定及分析尚未报道,本研究在大麻基因组及转录组数据基础上,通过生物信息学技术对大麻TCP基因家族进行鉴定和分析,为后续研究大麻TCP基因功能提供科学基础及参考依据,为工业大麻分子育种提供理论支持。
1 材料
本研究使用的雌性大麻CBDRx的全基因组和注释文件(GCA_900626175.1)以及水稻、金鱼草、玉米TCP基因序列信息,参考序列来源于NCBI(https://www.ncbi.nlm.nih.gov)网站,拟南芥基因组数据来源于TAIR在线数据库(https://www.arabidopsis.org)。番茄和水稻基因组数据分别来源于https://solgenomics.net/和http://rapdb.dna.affrc.go.jp/download/irgsp1.html。工业大麻C.sativa L.的叶、花、茎和种子转录组数据由本课题组测序获得。
2 方法
2.1 工业大麻Cs TCP基因家族成员及理化性质鉴定
采用2种方法在工业大麻中鉴定TCP基因家族成员,首先,利用TBtools进行工业大麻和拟南芥的双向blast去预测大麻中的TCP成员,然后使用Pfam数据库(http://pfam-legacy.xfamorg/family/browse)中的TCP结构域(PF03634)进一步筛选TCP家族成员,最后去除2种方法中的重复转录本后获取Cs TCP蛋白序列。采用Ex PASy在线网站(https://web.expasy.org/protparam/)对Cs TCP蛋白理化性质进行预测。在Wo LF PSORT(https://wolfpsort.hgc.jp)网站对Cs TCP蛋白进行亚细胞定位。
2.2 工业大麻Cs TCP基因家族系统进化分析
利用MEGA 6.0软件中的MUSCLE对氨基酸序列进行比对。以工业大麻和拟南芥TCP家族成员为研究对象,采用1000个bootstrap的邻接法(neighbor-joining,NJ)构建系统发树。并利用i TOL在线工具进一步对系统发育树进行美化。
2.3 工业大麻Cs TCP基因保守基序及基因结构分析
通过MEME数据库(https://meme-suite.org/meme/)对Cs TCP的蛋白保守基序进行预测,使用CDD在线网站(https://www.ncbi.nlm.nih.gov/cdd)对Cs TCP的蛋白结构域进行预测。使用TBtools软件将蛋白保守基序、保守结构域和其系统进化树整合及可视化作图。
2.4 工业大麻Cs TCP基因顺式作用元件预测
通过TBtools使用基因组序列和结构注释文件分别提取17个Cs TCP基因上游各2000 bp序列作为启动子区域。然后对启动子区的顺式作用元件的预测通过Plant Care(https://www.plantcare.co.uk/)进行,将预测结果进行可视化作图。
2.5 工业大麻Cs TCP基因染色体及共线性分析
使用TBtools软件分析工业大麻的基因组注释文件,提取每个TCP基因的在染色体上的位置信息并进行可视化绘制。从NCBI网站下载拟南芥、工业大麻的全基因组数据,从https://solgenomics.net/和http://rapdb.dna.affrc.go.jp/download/irgsp1.html网站分别下载水稻和番茄的全基因组数据,使用TBtools软件对其共线性关系进行可视化处理。
2.6 工业大麻Cs TCP基因差异表达分析
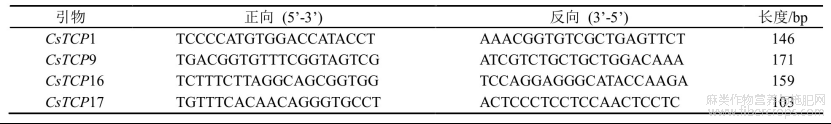
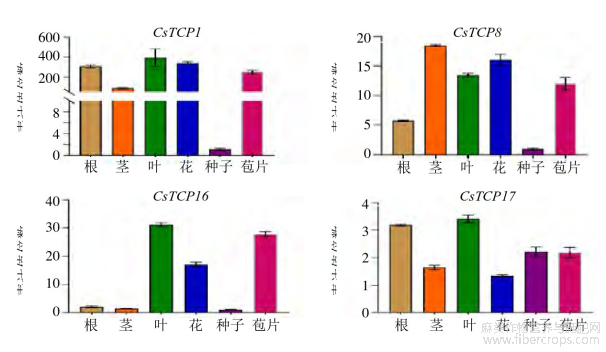
使用Dinamed kush CBD autoflowering(Diku)的花、叶、苞片、茎、种子和根的转录组数据计算Cs TCP基因的表达量(FPKM),并运用TBtools软件绘制热图,进行基因聚类和差异表达模式分析。根据RNAseq数据,选取Cs TCP1、Cs TCP9、Cs TCP16、Cs TCP17进行Real-time PCR验证分析。使用Quick RNA isolation Kit(0416-50,华越洋有限公司)试剂盒对工业大麻的根、茎、叶、花、苞片、种子进行RNA提取,然后用Hi Script II Q RT Super Mix for q PCR(R223-01,Vazyme)试剂盒反转录得到对应组织的c DNA。利用NCBI设计引物(表1),用Ace Q q PCR SYBR Green Master Mix(Q111-02,Vazyme)试剂盒进行Real-time PCR分析,反应程序为95℃预变性3min,95℃变性10s,60℃退火15s,72℃延伸15s,循环40次。
3 结果与分析
3.1 Cs TCP基因家族成员鉴定
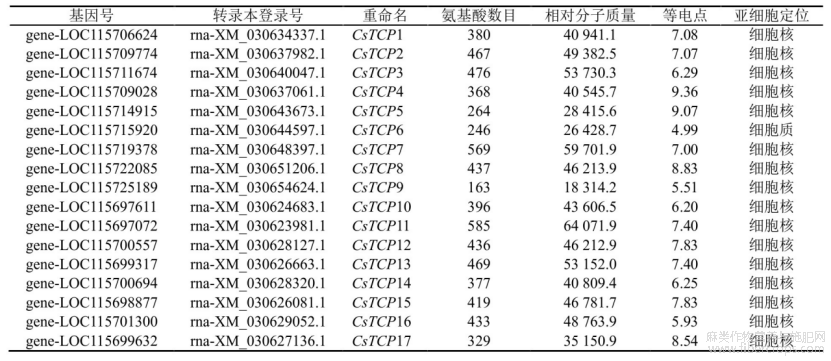
通过与拟南芥TCP基因家族双向比对以及Pfma网站结构域筛选,去除重复及冗余后共鉴定到17个大麻Cs TCP基因,按照染色体位置将其命名为Cs TCP1~Cs TCP17。利用Ex PASy在线网站对鉴定到的Cs TCP蛋白的基本理化性质进行分析(表2)。研究发现,所有的Cs TCP蛋白质氨基酸大小范围为163~585,蛋白质相对分子质量为18310~64070,此外这些Cs TCP蛋白的等电点介于4.99~9.36。亚细胞定位预测显示,全部的Cs TCP基因家族成员大部分在细胞核中,与其他物种中TCP基因家族成员理化性质相近(表2)。
表1 q RT-PCR引物
表2 Cs TCP基因家族成员基本信息
3.2 Cs TCP基因家族成员的系统进化分析及分类
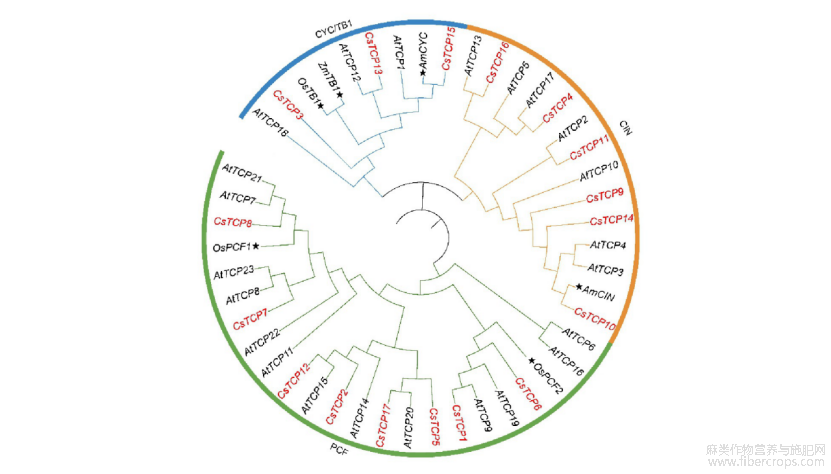
为了研究Cs TCP与其他植物物种TCP之间的进化关系,基于Cs TCP基因家族成员的多样性,将工业大麻、拟南芥、水稻、玉米、金鱼草的TCP基因共同构建系统发育树,以拟南芥TCP基因家族的分类方法为参考将Cs TCP分为3个亚家族(图1)。大麻中3个亚家族的分布情况为PCF亚家族有8个成员,其中Cs TCP8和Cs TCP7聚在一个分支,Cs TCP12、Cs TCP2、Cs TCP17和Cs TCP5聚在一个分支,Cs TCP1和Cs TCP6聚为一支。CIN亚家族有6个成员,其中Cs TCP10、Cs TCP14、Cs TCP9和Cs TCP11在一个分支上,Cs TCP4和Cs TCP16在同一分支。CYC亚家族有3个成员,Cs TCP3、Cs TCP13和Cs TCP15与拟南芥、水稻和金鱼草的CYC亚家族的成员聚为一支。
图1 Cs TCP基因家族系统发育分析
红色字体代表Cs TCP基因,AT-拟南芥Os-水稻Am-金鱼草Zm-玉米
3.3 Cs TCP基因的染色体定位及共线性分析
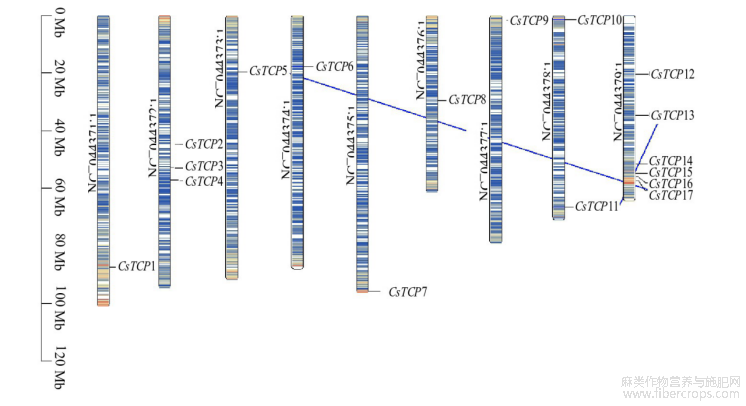
根据工业大麻基因组数据对Cs TCP基因家族成员进行染色体定位分析(图2),发现17个Cs TCP基因不均匀的分布在9条染色体上,而NC_044370.1染色体上没有Cs TCP基因,其余染色体上都有不同数量的Cs TCP基因。NC_044379.1染色体上分布的Cs TCP基因最多为6个,其次为NC_044372.1染色体,有3个Cs TCP基因分布在该染色体上,有2个Cs TCP基因分布在NC_044378.1染色体,其余染色体上均含有1个Cs TCP基因。
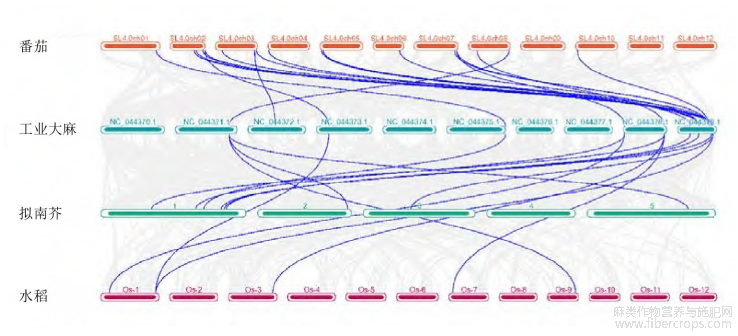
为了更深入的探究Cs TCP基因的结构和功能之间的相关性,以及阐明不同物种之间的进化关系,对工业大麻、拟南芥、水稻、番茄进行共线性分析(图3),彩色线代表其他物种中基因与Cs TCP基因具有共线性,其中拟南芥有9个、番茄有20个、水稻有6个与大麻TCP存在共线性基因对。其中有4个Cs TCP基因(Cs TCP1、Cs TCP10、Cs TCP11、Cs TCP17)在这4个物种中共同发生了基因组共线性。此外,Cs TCP基因组内部发生了2次共线性事件,其中有一对Cs TCP11/Cs TCP13发生在相邻的染色体上,另外一对为Cs TCP5/Cs TCP17(图2)。以上结果表明TCP基因家族在不同物种中的具有一定进化保守性。
图2 Cs TCP基因染色体分布情况
黑色字体为染色体名称及Cs TCP名称,蓝色的线代表Cs TCP种内共线性基因
图3 Cs TCP共线性分析
3.4 Cs TCP基因家族保守结构域和基因结构分析
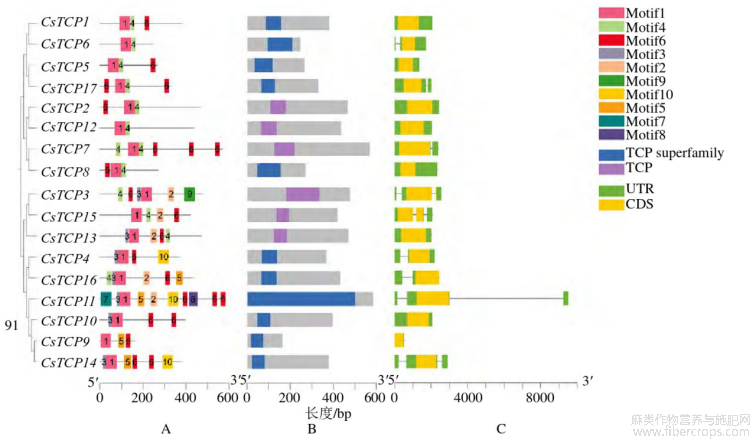
基因保守结构域通常与特定功能相关,例如转录激活/抑制、核定位和蛋白质相互作用。因此,利用MEME网站鉴定了Cs TCP蛋白的保守结构域,并选择了10个保守基序进行进一步分析(图4)。结果表明Motif1和Motif 6 2个保守基序在所有的Cs TCP均分布,但是没有Cs TCP包含所有的10个保守基序,表明这2个基序在Cs TCP蛋白序列中分布最广、保守程度最高(图4-A)。所有的Cs TCP蛋白均包含TCP保守结构域(图4-B)。另外,在所有的Cs TCP中均存在不同程度的保守基序变异现象,但每个Cs TCP含有3~10个保守基序。
为了进一步解析Cs TCP基因的结构多样性,对Cs TCP中的内含子、外显子进行了研究(图4-C),结果显示Cs TCP中的内含子数量为0~2不等,其中Cs TCP1、Cs TCP2、Cs TCP12、Cs TCP8、Cs TCP13、Cs TCP10和Cs TCP9没有内含子,其余均含有内含子。这些Cs TCP中都至少有2个外显子,Cs TCP11有5个外显子。Cs TCP基因的内含子和外显子分布结构紧密相连,高度保守,为基因家族复制研究奠定了基础。
3.5 Cs TCP基因顺式作用元件预测
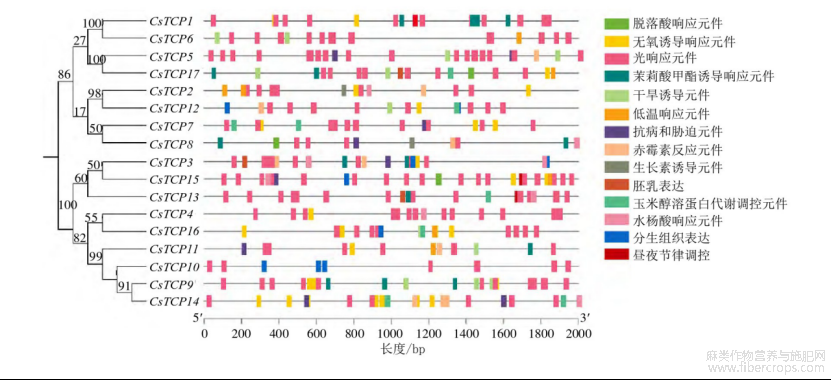
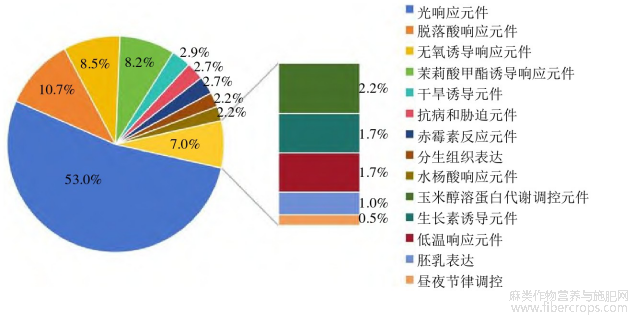
转录因子在控制植物如何响应生物和非生物胁迫方面很重要,其启动子序列中的各种顺式调节元件调节基因表达。为了探索17个Cs TCP基因在大麻中的潜在功能,预测了其启动子中的顺式调节元件(起始密码子前2000 bp)(图5)。所有Cs TCP总共包括419个顺式元件。它们参与了14个非生物胁迫,包括光响应性、水杨酸响应性、防御和胁迫响应性和低温响应性等。每个启动子中都有大量的光响应性元素占所有顺式元件的53%,其数量远远超过其他元素,其次是脱落酸响应元件,占所有顺式元件的10.7%,在所有顺式元件中昼夜节律调控元件占比最少,仅为0.5%(图6)。此外,17个Cs TCP中有9个对Me JA有反应,有14个参与脱落酸反应。此外,由于参与光响应性,脱落酸响应性,Me JA响应性和厌氧诱导调节的TCP基因构成了这些基因的很大一部分,推测这些基因可能在响应这些胁迫方面发挥重要作用。这些结果可以作为未来预测基因功能的理论基础。
图4 Cs TCP家族功能结构域
A-保守基序B-保守结构域C-基因结构
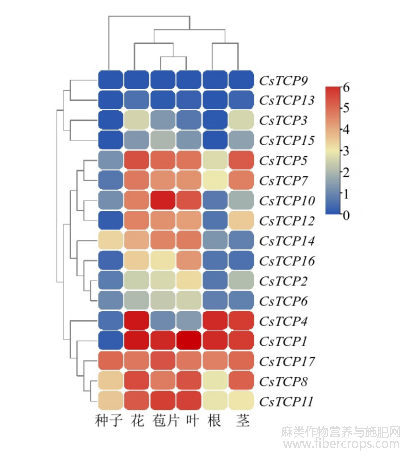
3.6 Cs TCP基因表达模式分析
为了探究Cs TCP基因在工业大麻不同组织的表达模式,对工业大麻品种的根、茎、叶、花、苞片、种子的转录组测序数据进行分析。结果表明Cs TCP基因在6个组织中表达模式不同,通过聚类分析发现,大部分基因在花、叶、苞片中高表达,小部分在根、茎、种子中高表达(图7)。Cs TCP17、Cs TCP8、Cs TCP11在6个组织中均有较好的表达,Cs TCP1在种子中几乎不表达,在其余5个组织中均高表达,在所有Cs TCP基因中花和叶中Cs TCP1表达量最高。此外Cs TCP9、Cs TCP13、Cs TCP3、Cs TCP15在6个组织中的表达均偏低,而这几个基因均属于CYC/TB1和CIN亚家族。筛选在组织间有显著差异且表达量较高的Cs TCP1、Cs TCP9、Cs TCP16、Cs TCP17 4个基因进行q RT-PCR验证,结果显示(图8),结果显示4个基因不同组织间的表达趋势和转录组结果一致。
图5 Cs TCP顺式作用元件分析
图6 Cs TCP顺式作用元件数量分析
4 讨论
TCP基因是植物中一类独特的小分子转录因子,在激素反应、次生代谢产物积累和生长发育调控中起着重要的作用。TCP蛋白参与调控植物生命周期中的许多生长发育过程,如萌发、叶片发育、开花、花器官发育、枝条生长、花粉发育等,通过其他因子的互作和不同激素途径的调节发挥作用[26,27,28,29]。然而工业大麻中TCP基因的机制尚未得到研究。
图7 Cs TCP基因表达模式分析
图8 Cs TCP Real-time PCR检测
在过去的几十年中,TCP转录因子的特性和表达研究取得了很大的进展,提高了人们对TCP基因家族的认识。TCP基因家族成员已在多个物种中被鉴定,目前被鉴定到最多的TCP基因的是烟草,多达63个[21],在其他植物中TCP基因的数量在24~58个(拟南芥24个[30]、小麦58个[24]、水稻27个[31])。本研究在工业大麻基因组中共鉴定到在工业大麻基因组中共鉴定出17个TCP转录因子,基因数量和拟南芥相当,但明显少于烟草和玉米的TCP基因。在不同植物基因组中TCP的数量差异可能与物种是否存在古老的全基因组复制或者近期的串联重复复制有关。此外,我们还研究了基因家族的物理化学特征;结果表明,17个Cs TCP在分子量、等电点和氨基酸数量方面表现出较大的差异,这表明工业大麻在进化过程中发生了变化。
先前的研究已经证明了内含子在调节基因表达方面的重要性,因此研究Cs TCP基因的结构可以揭示其功能。结果表明,所有CSTCP中的内含子范围为0~2,几种Cs TCP具有1或2个内含子,如Cs TCP6、Cs TCP7和Cs TCP3(图4-C),表明这些特殊的内含子可能与特定的基因功能有关。TCP结构域由大约60个氨基酸组成,并且在许多植物中高度保守[11,16,21],来自工业大麻的TCP包含相同的保守结构域(图4-B)。
植物中已知的TCP主要参与叶和花的形态以及茎分支的相关调控,例如有研究表明,拟南芥中的部分TCP基因表达下调之后,与野生型相比叶片形状发生了较大的变化[32],在玉米中也已经证明TCP相关基因BAD1可以调控叶的夹角[19],这说明TCP蛋白在叶发育过程中发挥了极其重要的作用。TCP也与花瓣的发育相关,例如在拟南芥中发现tcp5突变体花瓣较野生型更宽,而将TCP5沉默之后的花瓣比野生型窄[33]。有研究也表明,在斑叶草中CYC类基因具有不对称性的自调控和相互调控功能,在花对称性、花朝向和蜜导模式关联的进化中扮演了重要的角色[18]。在工业大麻基因组中共鉴定出的17个TCP转录因子,可分为2类3个亚科(PCF、CIN和CYC/TB1)。其中Cs TCP3、Cs TCP13、Cs TCP15、Cs TCP16、Cs TCP4、Cs TCP11、Cs TCP9、Cs TCP14和Cs TCP10属于CIN和CYC/TB1亚科,而转录组数据分析显示Cs TCP4、Cs TCP11和Cs TCP10在花中均有较高的表达,根据上述已知TCP功能研究证明CIN和CYC/TB1亚科的TCP基因通常参与调控花的形态,故推测Cs TCP4、Cs TCP11和Cs TCP10在工业大麻中可能也有调控花发育的功能,可能对火麻仁生产具有指导意义。在侧枝发育过程中,TCP基因主要起到抑制侧枝形成的作用。一个典型例子是在玉米中最早发现的TB1基因。这个基因通过抑制植物的侧枝生长来发挥功能,在玉米中过度表达TB1会显著减少分支数量[12]。类似于TB1在水稻中的同源基因Os TB1,过表达该基因也会导致水稻的分蘖数下降[34]。此外,在拟南芥中与TB1有功能相似的同源基因TCP18和TCP12,过表达它们可以使拟南芥的分枝数显著增多[35,36]。此外,TB1基因还被报道在番茄[37]、豌豆[38]和马铃薯[39]中具有抑制分枝的能力,这表明TCP转录因子对组织生长的抑制活性在植物演化过程中是高度保守的。将玉米、拟南芥的TB1基因和大麻的TCP构建了系统发育树,发现大麻中的Cs TCP3、Cs TCP13和Cs TCP15与上述的TB1基因聚集在一起。这提示在大麻中,Cs TCP3、Cs TCP13和Cs TCP15可能具有类似于TB1的抑制分枝的功能,其中转录组分析发现Cs TCP3在茎中具有较高表达量,在研究工业大麻侧枝发育时可优先将其作为候选基因进行分析。这一发现可能对以大麻茎材为原料的纤维类品种选育具有指导意义,但需要更多地后续研究来确定其功能。由于工业大麻作为经济和药用价值都很高的植物,在近年来得到了广泛关注和研究。随着技术的进步,我们可以期待未来对工业大麻中TCP基因的机制进行更详尽的探索,以揭示其在工业大麻生长发育和代谢调控中的作用机制。
利益冲突 所有作者均声明不存在利益冲突
参考文献
[1] McPartland J M, Small E. A classification of endangered high-THC Cannabis(Cannabis sativa subsp. indica)domesticates and their wild relatives[J]. PhytoKeys, 2020,144:81-112.
[2] 李俊,米要磊,王思嘉,等.基于UPLC-QQQ-MS/MS的工业大麻中6种大麻素类成分定量研究[J].中草药,2022, 53(4):1163-1172.
[3] 张汉文,张文君,张国锋,等.基于中药配伍的火麻仁药理作用研究进展[J].中国医院药学杂志, 2022,42(6):659-664.
[4] Schluttenhofer C, Yuan L. Challenges towards revitalizing hemp:A multifaceted crop[J]. Trends Plant Sci, 2017,22(11):917-929.
[5] Callaway J C. Hempseed as a nutritional resource:An overview[J]. Euphytica, 2004, 140(1):65-72.
[6] Ryz N R, Remillard D J, Russo E B. Cannabis roots:A traditional therapy with future potential for treating inflammation and pain[J]. Cannabis Cannabinoid Res,2017, 2(1):210-216.
[7] Livingston S J, Quilichini T D, Booth J K, et al. Cannabis glandular trichomes alter morphology and metabolite content during flower maturation[J]. Plant J, 2020,101(1):37-56.
[8] 周永凯,张建春,张华.大麻纤维的抗菌性及抗菌机制[J].纺织学报, 2007, 28(6):12-15.
[9] Xie Z Y, Mi Y L, Kong L Z, et al. Cannabis sativa:Origin and history, glandular trichome development, and cannabinoid biosynthesis[J]. Hortic Res, 2023, 10(9):150.
[10] Cubas P, Lauter N, Doebley J, et al. The TCP domain:A motif found in proteins regulating plant growth and development[J]. Plant J, 1999, 18(2):215-222.
[11] Kosugi S, Ohashi Y. PCF1 and PCF2 specifically bind to cis elements in the rice proliferating cell nuclear antigen gene[J]. Plant Cell, 1997, 9(9):1607-1619.
[12] Doebley J, Stec A, Hubbard L. The evolution of apical dominance in maize[J]. Nature, 1997, 386(6624):485-488.
[13] Luo D, Carpenter R, Vincent C, et al. Origin of floral asymmetry in Antirrhinum[J]. Nature, 1996, 383(6603):794-799.
[14] Viola I L, Gonzalez D H. TCP transcription factors in plant reproductive development:Juggling multiple roles[J].Biomolecules, 2023, 13(5):750.
[15] Martín-Trillo M, Cubas P. TCP genes:A family snapshot ten years later[J]. Trends Plant Sci, 2010, 15(1):31-39.
[16] Li C X, Potuschak T, Colón-Carmona A, et al. Arabidopsis TCP20 links regulation of growth and cell division control pathways[J]. Proc Natl Acad Sci U S A, 2005, 102(36):12978-12983.
[17] Howarth D G, Donoghue M J. Phylogenetic analysis of the ECE(CYC/TB1)clade reveals duplications predating the core eudicots[J]. Proc Natl Acad Sci USA, 2006, 103(24):9101-9106.
[18] Yang X, Wang Y, Liu T X, et al. CYCLOIDEA-like genes control floral symmetry, floral orientation, and nectar guide patterning[J]. Plant Cell, 2023, 35(8):2799-2820.
[19] Bai F, Reinheimer R, Durantini D, et al. Tcp transcription factor, branch angle defective 1(bad1), is required for normal tassel branch angle formation in maize[J]. Proc Natl Acad Sci U S A, 2012, 109(30):12225-12230.
[20] Aggarwal P, Das Gupta M, Joseph A P, et al. Identification of specific DNA binding residues in the TCP family of transcription factors in Arabidopsis[J]. Plant Cell, 2010,22(4):1174-1189.
[21] 张路阳,韩文龙,徐晓雯,等.烟草TCP基因家族的鉴定及表达分析[J].生物技术通报, 2023, 39(6):248-258.
[22] 刘俊,李龙,吴耀松,等.杜仲TCP转录因子鉴定及生物信息学分析[J].中草药, 2022, 53(18):5813-5824.
[23] 雷静,班秀文,刘荣,等.薏苡(Coix lacryma-jobi L.)TCP家族全基因组鉴定及响应非生物胁迫的表达规律[J/OL].分子植物育种,[2023-05-11]. https://kns.cnki.net/kcms/detail/46.1068.S.20230510.1438.004.html.
[24] 吕齐,焦红红,余祺瑞,等.人参TCP转录因子家族生物信息学分析与功能预测[J].中国中药杂志, 2021,46(15):3838-3845.
[25] 肖玉林.藜麦TCP基因家族鉴定分析与功能验证[D].济南:山东师范大学, 2021.
[26] Viola I L, Alem A L, Jure R M, et al. Physiological roles and mechanisms of action of class I TCP transcription factors[J]. Int J Mol Sci, 2023, 24(6):5437.
[27] Nicolas M, Cubas P. TCP factors:New kids on the signaling block[J]. Curr Opin Plant Biol, 2016, 33:33-41.
[28] Danisman S. TCP transcription factors at the interface between environmental challenges and the plant’s growth responses[J]. Front Plant Sci, 2016, 7:1930.
[29] Manassero N G, Viola I L, Welchen E, et al. TCP transcription factors:Architectures of plant form[J].Biomol Concepts, 2013, 4(2):111-127.
[30] Danisman S, van Dijk A D J, Bimbo A, et al. Analysis of functional redundancies within the Arabidopsis TCP transcription factor family[J]. J Exp Bot, 2013, 64(18):5673-5685.
[31] 关紫微,曹希雅,张先文,等.水稻TCP家族的全基因组鉴定及表达分析[J].分子植物育种, 2022, 20(10):3145-3156.
[32] Nath U, Crawford B C W, Carpenter R, et al. Genetic control of surface curvature[J]. Science, 2003, 299(5611):1404-1407.
[33] Huang T B, Irish V F. Temporal control of plant organ growth by TCP transcription factors[J]. Curr Biol, 2015,25(13):1765-1770.
[34] Takeda T, Suwa Y, Suzuki M, et al. The Os TB1 gene negatively regulates lateral branching in rice[J]. Plant J,2003, 33(3):513-520.
[35] Aguilar-Martínez J A, Poza-Carrión C, Cubas P.Arabidopsis BRANCHED1 acts as an integrator of branching signals within axillary buds[J]. Plant Cell,2007, 19(2):458-472.
[36] Poza-Carrión C, Aguilar-Martínez J A, Cubas P. Role of TCP gene BRANCHED1 in the control of shoot branching in Arabidopsis[J]. Plant Signal Behav, 2007, 2(6):551-552.
[37] Martín-Trillo M, Grandío E G, Serra F, et al. Role of tomato BRANCHED1-like genes in the control of shoot branching[J]. Plant J, 2011, 67(4):701-714.
[38] Braun N, de Saint Germain A, Pillot J P, et al. The pea TCP transcription factor PsBRC1 acts downstream of Strigolactones to control shoot branching[J]. Plant Physiol, 2012, 158(1):225-238.
[39] Nicolas M, Rodríguez-Buey M L, Franco-Zorrilla J M, et al. A recently evolved alternative splice site in the BRANCHED1a gene controls potato plant architecture[J].Current Biol, 2015, 25(14):1799-1809.
文章摘自:秦振芬,孟祥霄,陈伟强等. 工业大麻TCP基因家族的鉴定及表达分析 [J]. 中草药, 2023, 54 (23): 7806-7814.