摘 要:不饱和脂肪酸为人体提供基本代谢所必需的能量,须从膳食中补充。脂肪酸去饱和酶FAD(fatty acid desaturase)是植物不饱和脂肪酸合成途径中的关键酶,植物体内脂肪酸的各组分比例和不饱和度与FAD的去饱和作用息息相关。为探究亚麻FAD基因家族的表达与进化,为其在亚麻高品质育种中的应用提供理论依据。运用生物信息学方法对亚麻全基因组FAD基因家族的43个LuFADs基因进行分析。结果显示,该家族成员编码的蛋白质大小为152~453个氨基酸,大部分为碱性不稳定亲水蛋白。与拟南芥FADs蛋白序列构建系统发育树,可分为4个主要亚家族:Δ12/ω-3去饱和酶、“前端”去饱和酶、Δ7/Δ9去饱和酶和SAD去饱和酶。保守结构域和外显子-内含子结构分析得出,同一亚组中的家族成员具有较为相似的基因结构。染色体定位分析呈随机性分布。亚细胞定位预测得出,叶绿体上的家族成员最多。启动子顺式作用元件分析发现,该家族成员中抗氧化反应元件(ARE)数量最多。
关键词:亚麻;FAD基因家族;生物信息学分析
亚麻(Linum usitatissimum L.)是一年生自交草本植物,在我国甘肃、内蒙古、山西、宁夏和河北等地广泛种植[1]。亚麻可分为油用、纤维用和油纤兼用3种类型,其中油用亚麻俗称胡麻,胡麻籽含油率为40%左右,具有较高的食用营养价值[2]。胡麻籽油的脂肪酸组成包括2种脂肪酸(棕榈酸及硬脂酸)和3种不饱和脂肪酸(油酸、亚油酸及α-亚麻酸),这5种脂肪酸占到胡麻油粗脂肪含量的99%,其中α-亚麻酸达到54%左右[3]。α-亚麻酸是人体必需脂肪酸,属于ω-3系不饱和脂肪酸,具有降血脂、抗血栓肿瘤、健脑明目和改善肌肤等多种保健功能[4]。
不饱和脂肪酸的合成以饱和脂肪酸硬脂酸为底物,在脂肪酸延长酶(fatty acid elongase,FAE)和脂肪酸去饱和酶FAD(fatty acid desaturase)的作用下,通过一系列的脱氢、延长、去饱和作用形成[5]。其中FAD是植物不饱和脂肪酸合成的关键酶,植物体内脂肪酸各组分的比例及其不饱和度与FAD的去饱和作用息息相关[6]。FAD家族主要在质体和内质网中催化油酸生成亚油酸、亚油酸生成亚麻酸[7]。依据辅因子和亚细胞定位的不同,FAD可划分为膜结合和可溶性2种类型。膜结合FAD包括ω-6FAD(催化油酸形成亚油酸)和ω-3FAD(催化亚油酸形成a-亚麻酸),可溶性FAD包括Δ4FAD(催化二十碳五烯酸EPA生成十二碳六烯酸DHA)、Δ6FAD(催化亚油酸生成γ-亚麻酸)及Δ9SAD(催化硬脂酸生成油酸)[8]。学者们对芝麻、油菜、向日葵、油茶等油料作物的FAD基因家族进行了生物信息学分析[9-12],在其他作物中,例如藜麦、荞麦、茄子等植物中也有该基因家族分析相关报道[13-15]。相关研究发现,FAD与细胞膜的稳定性和流动性也相关,进而对植株抗逆性产生影响[16]。亚麻FAD基因家族研究基于转录组数据的分析[17],但随着高通量测序技术的发展以及亚麻全基因组数据的更新,对于脂肪酸合成途径相关基因家族的分析有待进一步完善。我们运用生物信息学方法对亚麻全基因组的FAD基因家族进行分析,包括蛋白理化性质分析、系统进化分析、保守结构域及基因结构分析、染色体定位分析、亚细胞定位预测和启动子顺式作用元件分析,以期为其在亚麻高品质育种中的应用提供理论依据。
1材料与方法
1.1序列来源
从NCBI上获得亚麻(version2.0)15条染色体的基因序列(登录号为CP027619-CP027633)[18],从phytozome数据库(https://phytozome.jgi.doe.gov/pz/portal.html#)中下载蛋白质序列。
1.2研究方法
1.2.1LuFADs基因家族成员筛选与鉴定
使用以下2种方法鉴定亚麻FAD蛋白基因。第1种是基于拟南芥基因组(Version10.0,https://www.arabidopsis.org/)中的27个参考序列,通过BLASTP来鉴定亚麻基因组中的FAD蛋白,E值设置为1.0E-10。第2种是从Pfam蛋白家族数据库(http://pfam.xfam.org/)下载与FA_Desaturase(PF00487)、FA_Desaturase2(PF03405)和TMEM189(PF10520)结构域对应的隐马尔可夫模型(HMM)文件作为query,使用HMMERv3.3(http://eddylab.org/software/hmmer/hmmer-3.3.tar.gz)中的hmmsearch工具来查找亚麻蛋白数据库中所有可能的FAD蛋白序列,E值<1e-5。
1.2.2亚麻LuFADs基因的生物信息学分析
利用ClustalW对亚麻、拟南芥FAD蛋白序列进行多重序列比对,接着在MEGA11软件中使用邻接法进行系统进化树构建。使用Bioperl工具(https://bioperl.org/)计算亚麻FAD家族成员的氨基酸数量、蛋白理论等电点(PI)和分子量大小(Mw)等指标[19]。利用本地MEME工具(版本4.11.2,http://alternate.meme-suite.org/tools/meme)搜索亚麻FAD家族成员序列中的保守基序。通过在线软件(MG2C,http://mg2c.iask.in/mg2c_v2.1/)和(GSDS,http://gsds.cbi.pku.edu.cn/)分别构建亚麻FAD家族成员的染色体位置图和外显子-内含子结构图。使用CELLO(http://cello.life.nc-tu.edu.tw/)进行LuFAD家族成员亚细胞定位预测。采用Perl脚本提取亚麻FAD家族成员基因编码区上游1500bp的序列,并利用在线数据库PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对该基因家族成员中的顺式作用元件进行鉴定[20]。
2结果与分析
2.1LuFADs基因家族成员鉴定及蛋白理化性质分析
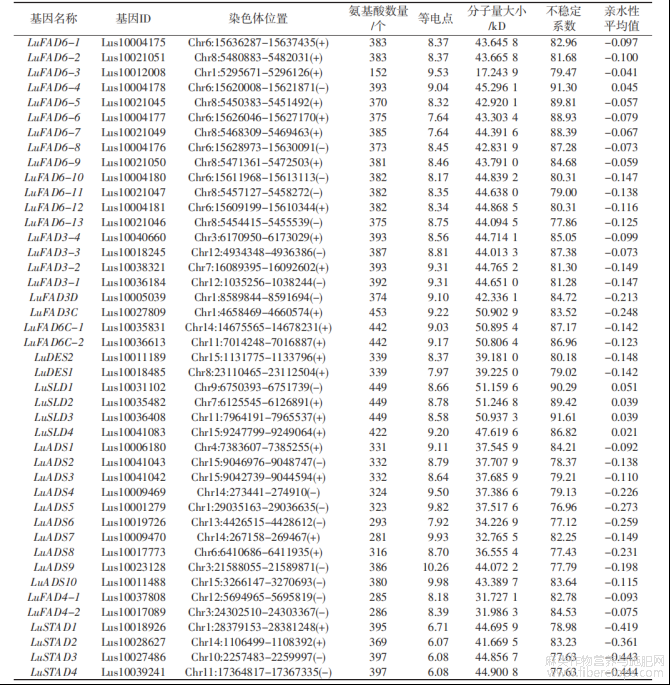
综合2种方法的筛选结果,运用SMART(http://smart.embl.de/)和NCBI-CDD(https://www.ncbi.nlm.nih.gov/cdd/)数据库进一步验证筛选结果中是否具有FAD结构域组成,去除有错误、短小(<100aa)和没有FAD结构域的蛋白质序列,最终鉴定了43个LuFADs基因,并依据它们在15条染色体位置信息和系统发育分析结果进行命名(表1、图1)。编码的蛋白质范围为152~453个氨基酸。LuFADs的理论分子量为17.2439(LuFAD6-3)~51.2468kD(LuSLD2),等电点(pI)为6.07~10.26,LuSTAD(1~4)系列的pI<7.00,呈酸性,其余39个成员pI均大于7,呈碱性。LuFADs基因蛋白的不稳定性指数范围为76.96~91.61,均为不稳定蛋白。亲水性平均值(GRAVY)为正数的有5个,分别为LuSLD(1~4)系列和LuFAD6-4,表现为不亲水性,其余38个家族成员均为亲水蛋白(表1)。
表1 LuFADs基因家族成员鉴定
2.2LuFADs家族成员系统进化分析
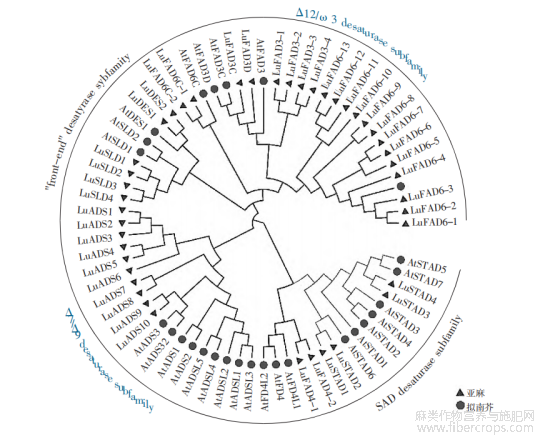
运用邻接法将LuFADs蛋白序列(43)与拟南芥FADs蛋白序列(27)构建系统发育树(图1),可分为4个主要亚族:Δ12/ω-3去饱和酶亚族、“前端”去饱和酶亚族、Δ7/Δ9去饱和酶亚族和SAD去饱和酶亚族。
Δ12/ω-3去饱和酶亚家族包括3个分支:FAD6、FAD3和FAD6C。其中FAD6分支包括拟南芥的AtFAD6和亚麻的LuFAD6(1-13);FAD3分支为膜结合ω-3FAD系列基因,包括拟南芥的AtFAD3、AtFAD3C、AtFAD3D和亚麻的LuFAD3C、LuFAD3D、LuFAD3-1、LuFAD3-2、LuFAD3-3、LuFAD3-4;FAD6C分支包括拟南芥的AtFAD6C和亚麻的LuFAD6C-1、LuFAD6C-2。其余3个亚族中,亚麻基因成员和拟南芥基因成员均表现较高的聚集性,特别是在Δ7/Δ9去饱和酶亚族中,亚麻基因LuADS(1-10)和拟南芥AtADS系列基因分别各自聚为1个分支。
图1 亚麻LuFADs和拟南芥AtFADs系统发育树
2.3亚麻FAD家族保守结构域和基因结构分析
对亚麻LuFADs基因家族进行保守结构域分析(表2,图2)可知,同一亚族中保守结构域的分布较为相似,LuADS亚组中均包含motif-1、motif-3和motif-7;LuSTAD亚组中均只含motif-10;LuDES和LuSLD亚组中含有motif-2;LuFAD6亚组中,除LuFAD6-3外其他基因均含有motif-1、motif-2、motif-4、motif-5、motif-6、motif-8和motif-9;LuFAD4-1和LuFAD4-2不含有motif。
对LuFADs基因家族进行外显子-内含子结构分析(图2)显示,在LuFAD6亚组中,只有Lu-FAD6-4含有2个内含子,其余12个基因均只有外显子;LuFAD4-1、LuFAD4-2和LuSLD系列没有内含子,剩余基因有不同数量(1~7,9)内含子。LuSLD(1~3)的CDS区最长,大于1000bp;Lu-ADS10的基因长度最长,约为4500bp,LuFAD6-3基因最短,不足500bp。
综合家族系统进化可以看出,同一亚组中的基因具有较为相似的结构。LuSTAD亚组中均含有2个内含子。LuADS亚组中,除LuADS8的显子数量为4外,其余均为5。LuFAD6C-1和Lu-FAD6C-2内含子最多,具有9个。LuADS10的内含子序列最长。
表2 保守基序motif序列分布情况
图2 亚麻FAD家族成员的系统进化、保守motif和基因结构分析
2.4LuFADs基因家族成员染色体定位和亚细胞预测
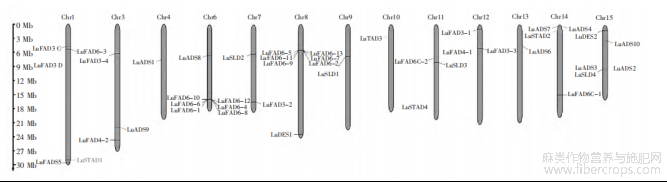
由图3可知,LuFADs基因家族随机分布于除第2号和5号染色体(Chr2、Chr5)之外的所有亚麻染色体上,分布广泛。Chr4、Chr9、Chr10和Chr13上各自有1个基因成员,Chr7上有2个基因,Chr3、Chr11和Chr12上各分布着3个基因,Chr14上有4个基因,Chr1和Chr15各为5个基因。Chr6和Chr8的LuFADs家族成员最多,均为7个,其中在Chr6上LuFAD6-1、LuFAD6-4、LuFAD6-6、LuFAD6-8、LuFAD6-10和LuFAD6-12距离较近,在Chr8染色体上LuFAD6-2、Lu-FAD6-5、LuFAD6-7、LuFAD6-9、LuFAD6-11和LuFAD6-13的距离较为接近。
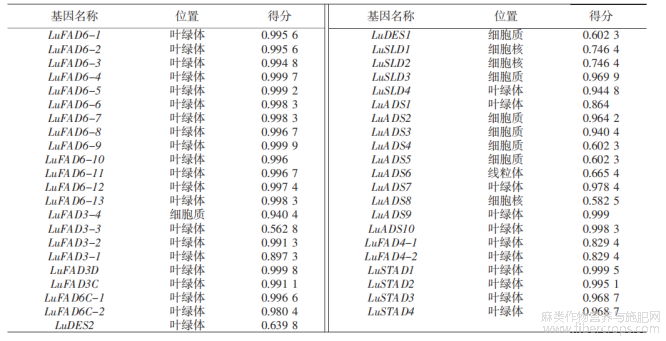
对LuFADs家族成员进行亚细胞定位预测(表3)可知,定位结果分为4类,包括叶绿体、细胞质、细胞核和线粒体。定位在叶绿体上的家族成员最多,共32个基因。细胞质上7个基因成员,细胞核上3个基因,线粒体上1个基因。其中Lu-FAD6、LuFAD4和LuSTAD成员的基因均定位在叶绿体上。
图3 LuFADs基因家族成员染色体分布情况
表3 LuFADs的亚细胞定位
2.5LuFAD启动子顺式作用元件分析
对LuFADs进行启动子顺式作用元件分析(图4)可知,该基因家族含有的元件可分为4类,光响应元件(G-box、Box4、GATA-motif、I-box、TCT-motif、Sp1、TCCC-motif、GT1-motif)、激素响应元件(TGACG-motif、CGTCA-motif、ABRE、TGA-element、AuxRR-core、TCA-element、P-box、GARE-motif、TATC-box)、胁迫响应元件(MBS、TC-richrepeats、LTR、ARE、GC-motif)和生长发育相关元件(A-box、O2-site、CCAAT-box)。其中抗氧化反应元件ARE数量最多(89),其次是G-box(77),Box4(74)排第3,TGACG-motif和CGTCA-motif在基因家族中的含量分布数量相同(72)。基因LuFAD3-3含元件数最多,为35个;LuADS3和LuADS7的元件数最少,均为6个。
图4 LuFAD基因家族成员启动子顺式作用元件分布情况
3讨论与结论
近年来,FAD基因家族在多种植物物种中得到鉴定验证,其家族成员数量不尽相同,如甜荞麦中10个[14],大豆中29个[21],陆地棉中41个[22]。本研究分析鉴定了亚麻43个LuFADs基因家族成员,gDNA长度为456~4547bp,编码的氨基酸为152~453个。等电点(pI)6.07~10.26。除LuSLD(1~4)系列和LuFAD6-4,其余38个家族成员均为亲水蛋白。有研究者对茄子的FAD家族序列进行理化性质分析,表明分子量与成员所含氨基酸数量成正比,大部分家族成员为亲水性蛋白,这与本研究结果相似[15]。系统发育树分析聚为4类,同一亚族中保守结构域的分布较为相似,在Δ7/Δ9去饱和酶亚族中均含motif-1、motif-3和motif-7,在SAD去饱和酶亚族中均只含motif-10;在“前端”去饱和酶亚族中只含motif-2,在Δ12/ω-3去饱和酶亚族中LuFAD6、LuFAD6C、LuFAD3C和LuFAD3D各分支的保守结构域相似。Liu等[23]用最大似然法将核桃FADs蛋白序列与拟南芥FADs蛋白序列一起构建系统发育树,分析JrFADs基因家族,也分为4个亚家族,与本研究相一致。
对基因外显子-内含子结构分析表明,同一亚组中的基因具有较为相似的基因结构。Xue等[10]对油菜FAD基因家族进行基因结构分析,各个FAD亚家族的外显子-内含子结构高度保守。Liu等[23]分析JrFADs基因家族,结果表明同一亚家族中的基因具有相似的基因结构。Cheng等[24]对香蕉同一亚科中的MaFAD家族成员进行分析,显示出相似的内含子/外显子结构和内含子相,并且它们编码的蛋白质由相似的基序组成。
染色体定位和亚细胞预测发现,LuFADs基因家族随机地分布在除Chr2和Chr5之外的所有亚麻染色体上。亚细胞定位在叶绿体上的家族成员最多,共有32个基因。Xue等[10]的研究表明,油菜3个芸薹属物种中FAB2、FAD4、FAD6和FAD8蛋白质的所有成员均位于叶绿体中。启动子顺式作用元件分析得出,抗氧化反应元件ARE的数量最多,为89个,基因LuFAD3-3所含元件数最多。Yasemin等[25]对向日葵的FAD基因家族的研究表明,主要定位在叶绿体和内质网膜中,其中FAB2家族成员位于叶绿体中,可能与FAD在细胞不同部位脂肪酸去饱和中的作用有关。综合分析向日葵、芝麻、油菜、可可树和亚麻的FAD基因家族,在氧化还原过程、脂肪酸生物合成过程或脂质代谢过程中发挥作用,这与本研究结果相似。
参考文献
[1]伍叶娜,潘根,姜慧,等.亚麻全基因组关联分析研究进展[J].中国麻业科学,2022,44(2):109-118.
[2]张辉,贾霄云,高凤云,等.胡麻[M].北京:中国农业科学技术出版社,2021.
[3]廖振林,李倩滢,陈俊杰,等.亚麻籽油组分的功能活性研究进展[J].现代食品科技,2021,37(11):379-389;337.
[4]唐佳芮,魏冰,石珊珊.α-亚麻酸分离纯化技术研究进展[J].中国油脂,2020,45(8):28-31;38.
[5]LEE K R, CHEN G Q, KIM H U. Current progress to- wards the metabolic engineering of plant seed oil for hy- droxy fatty acids production[J]. Plant Cell Reports, 2015, 34(4):603-15.
[6]POVKHOVA L V, MELNIKOVA N V, ROZHMINA T A, et al. Genes associated with the flax plant type (oil or fiber) identified based on genome and transcriptome se- quencing data[J]. Plants, 2021, 10(12): 2616.
[7]CHELLAMUTHU M, KUMARESAN K, SUBRAMANIAN S. Increase in alpha-linolenic acid content by simultane- ous expression of fatty acid metabolism genes in Sesame (Sesamum indicum L.)[J]. Physiology and Molecular Biology of Plants, 2022, 28(3): 559-572.
[8]吴端,王力军,杨仕梅,等.植物种子a-亚麻酸形成及调控机理研究进展[J].植物遗传资源学报,2020,21(1):49-62.
[9]WANG L H, YU S, TONG C B, et al. Genome sequenc- ing of the high oil crop sesame provides insight into oil biosynthesis[J]. Genome Biology,2014,15(2):39- 46.
[10]XUE Y, CHEN B, WANG R, et al. Genome-wide sur- vey and characterization of fatty acid desaturase gene family in brassica napus and its parental species[J]. Applied Biochemistry and Biotechnology, 2018, 184 (2): 582-598.
[11]于海峰,韩平安,李美娜,等.基于RNA-Seq技术的向日葵油酸形成的转录组学分析[J].中国油料作物学报,2018,40(6):769-776.
[12]江南,谭晓风,张琳,等.基于RNA-Seq的油茶种子α-亚麻酸代谢途径及相关基因分析[J].林业科学,2014,50(8):68-75.
[13]刘江,王育川,董艳辉,等.藜麦FAD2基因鉴定及生物信息学分析[J].分子植物育种,2022,20(3):715-721.
[14]赵训超,魏玉磊,丁冬,等.甜荞麦脂肪酸脱氢酶基因(FeFAD)家族的鉴定与分析[J].东北农业科学,2021,46(1):36-41.
[15]朱宗文,张爱冬,吴雪霞,等.生物信息学鉴定分析茄子脂肪酸去饱和酶(FAD)基因家族[J/OL].分子植物育种:1-15(2021-05-12)[2022-05-18].http://kns.cnki.net/kcms/detail/46.1068.S.20210512.0826.002.html
[16]DOMINGUEZ T, HERNANDEZ M L, PENNYCOOKE J C, et al. Increasing ω-3 desaturase expression in toma- to results in altered aroma profile and enhanced resis- tance to cold stress[J]. Plant Physiology, 2010, 153(2): 655-665.
[17]YOU F M, LI P C, KUMAR S, et al. Genome-wide i- dentification and characterization of the gene families controlling fatty acid biosynthesis in flax (Linum usi- tatissimum L.)[J]. Journal of Proteomics & Bioinfor- matics, 2014, 7(10):310.
[18]YOU F M, XIAO J, Li P, et al. Chromosome-scale pseu- domolecules refined by optical, physical and genetic maps in flax[J]. Plant Journal, 2018, 95(2): 371-384.
[19]刘畅畅,黄敏.玉米LEC1基因家族的鉴定与生物信息学分析[J].甘肃农业科技,2022,53(1):31-37.
[20]张国琴,葛玉彬,张正英.高粱耐冷基因全基因组鉴定及进化分析[J].甘肃农业科技,2020(12):37-41.
[21]CHI X Y, YANG Q L, LU Y D, et al. Genome -wide analysis of fatty acid desaturases in soybean(Glycine max)[J]. Plant Molecular Biology Reporter, 2011, 29 (4): 769-783.
[22]FENG J, DONG Y, LIU W, et al. Genome-wide identi- fication of membrane-bound fatty acid desaturase genes in Gossypium hirsutum and their expressions during abi- otic stress[J]. Sci Rep, 2017, 1(7):1-12.
[23]LIU K, ZHAO S, WANG S, et al. Identification and analy- sis of the FAD gene family in walnuts(Juglans regia L.) based on transcriptome data[J]. BMC Genomics, 2020, 21(1):299-303.
[24]CHENG C, LIU F, SUN X, et al. Genome-wide identifi- cation of FAD gene family and their contributions to the temperature stresses and mutualistic and parasitic fungi colonization responses in banana[J]. International Jour- nal of Biological Macromolecules, 2022, 204: 661-676.
[25]YASEMIN C A, NECDET M U, MEHMET C B, et al. Comparative identification and evolutionary relationship of fatty acid desaturase (FAD) genes in some oil crops: the sunflower model for evaluation of gene ex- pression pattern under drought stress[J]. Biotechnology & Biotechnological Equipment, 2018, 32(4): 846-857.
文章摘自:侯静静,赵利,王斌.亚麻FAD基因家族的生物信息学鉴定分析[J].寒旱农业科学,2023,2(03):246-253.