摘 要:磷脂酶A1(PhospholipaseA1,PLA1)在植物生长发育及胁迫反应中发挥着重要作用,但目前关于PLA1在亚麻中的鉴定与表达特征尚未见报道。本研究利用生物信息学方法对包括亚麻在内的7个物种PLA1基因家族进行鉴定,分析了亚麻PLA1(LuPLA1)的序列特征、系统进化关系、顺式作用元件、共线性关系及其复制事件,并利用转录组数据分析了其在不同遗传背景、不同器官中的表达模式,利用qRT-PCR分析了其在不同组织、不同发育时期及胁迫处理下的表达模式。结果表明,在亚麻、拟南芥、玉米、水稻、大豆、蓖麻及木薯基因组中分别鉴定到14、21、15、20、41、18和35个PLA1成员;LuPLA1基因分布于8条染色体上。序列特征分析发现,除LuPLA1-7和LuPLA1-10外,其他家族成员都具有内含子且多数含有1个内含子;LuPLA1蛋白序列长度为388~1759aa,等电点为5.99~9.19,分子量为41.55~192.61kD,均为亲水性蛋白,大多数定位于液泡,含有4~25个Motif。系统进化分析将PLA1蛋白分为4个分支,其中第IV分支PLA1成员数量最多。共线性分析表明,LuPLA1在拟南芥、玉米、水稻、木薯及大豆中均具有同源基因且与大豆、木薯的同源基因最多。LuPLA1家族成员中共有2对基因发生串联复制,7对基因发生片段复制,且都经历了纯化选择。转录组分析发现大多LuPLA1成员表现为器官特异性表达。LuPLA1启动子区含大量激素与逆境响应相关元件,qRT-PCR分析进一步证实LuPLA1基因受激素、干旱、高盐、低温及高温诱导表达。其中LuPLA1-1受IAA诱导表达;LuPLA1-1和LuPLA1-6受NAA诱导表达;LuPLA1-1、LuPLA1-5和LuPLA1-6受GA3诱导表达;LuPLA1-12/14受NaCl和PEG诱导表达;除LuPLA1-2/4外,其余基因均受高温诱导表达;所有LuPLA1受低温诱导表达,对低温的响应最为明显。本研究为进一步解析LuPLA1基因家族的功能奠定了基础。
关键词:亚麻;LuPLA1;基因家族;表达分析
亚麻(LinumusitatissimumL.)属于亚麻科亚麻属,自花授粉,是一种一年生的二倍体草本双子叶植物(2n=2x=30)。亚麻是我国重要的油料作物及经济作物,具有耐旱、耐寒、耐瘠薄等特点,广泛应用于油脂、纺织、印刷、制革、医药、食品等工业,具有良好的经济价值[1-3]。近年来,随着人们健康意识的不断增强,开发亚麻籽中的有益成分(α-亚麻酸、木酚素、膳食纤维等)已成为亚麻加工业的重点目标之一[4]。
磷脂是细胞膜的主要成分,磷脂酶(Phospholipase,PLs)是磷脂切割酶,参与植物生长发育及胁迫应答等多种生命过程[5]。磷脂酶因磷脂分子内的水解位点不同而被分为5类,分别为磷脂酶A1(PhospholipaseA1,PLA1)、磷脂酶A2(PhospholipaseA2,PLA2)、溶血磷脂酶(PhospholipaseB,PLBs)、磷脂酶C(PhospholipaseC,PLCs)和磷脂酶D(PhospholipaseD,PLDs),其中PLBs不存在于植物中。甘油主链上的疏水脂肪酸尾部由PLAs和PLBs去除,而亲水磷酸头部附着在其上的侧链由PLCs和PLDs去除[6-7]。植物几乎所有的生命活动和应答机制都有磷脂酶的参与,如种子萌发[8]、细胞伸长[9]、植株衰老[10]、植物抗逆性的提高[11]等。与其他磷脂酶相比,PLA1相关研究相对较少。
PLA1广泛存在于自然界中,许多微生物和动植物组织细胞都具有PLA1活性[12]。磷脂分子sn-1位酯键可以被PLA1特异性水解进而生成游离脂肪酸和溶血磷脂,包括亚麻酸等不饱和脂肪酸[13-14]。在哺乳动物中鉴定到9个PLA1蛋白,其中6个是细胞外酶,3个是细胞内酶,其氨基酸序列具有较大差异,且表现出不同的功能[15]。相关研究表明,PLA1在微生物中也广泛存在,其中液化沙雷氏菌、假单孢菌及曲霉菌等均具有PLA1蛋白[16]。PLA1在植物中的发现较迟于动物和微生物,1995年,首次在植物中发现的PLA1蛋白为花药不开裂蛋白1(DAD1)[17]。拟南芥中DAD1蛋白参与植物生长调控因子茉莉酸的合成[18]。此外,拟南芥中AtLCAT3蛋白也具有PLA1活性[19]。Seo等[20]在辣椒中克隆到了PLA1的cDNA序列,并在拟南芥中对其进行了过表达,与野生型相比,转基因株系的根、叶和叶柄明显较长,生长速度更快,引起过早抽苔并且具有较长的花序,这表明PLA1可能参与细胞及组织生长的一系列正向调控机制。姜惠娜等[21]在西藏野生垂穗披碱草中克隆到PLA1基因并对其进行了表达分析,发现低温胁迫下PLA1基因表达量明显上调。有研究表明,PLA1在植物响应机械损伤的过程中也起着十分重要的作用[22]。近年来在玉米单倍体诱导的研究中[23]发现,在ZmPLA1的C端编码区插入一个四碱基对,导致其编码磷脂酶基因功能的减弱或丧失,最终诱导单倍体的产生,且在水稻[24]、小麦[25]等作物中对PLA1/MATL基因进行编辑,也成功获得了单倍体诱导系,推测ZmPLA1同源基因在禾本科植物中单倍体诱导具有保守的功能。到目前为止,亚麻中尚未有PLA1相关的研究。
本研究利用生物信息学手段,对亚麻PLA1基因家族进行全基因组鉴定,并对其理化性质、结构特征、进化关系、染色体定位、复制事件及启动子顺式元件等进行了系统分析,同时采用qRT-PCR分析该家族成员在亚麻不同时期、不同器官及非生物胁迫下的表达模式,旨在为深入研究亚麻PLA1家族各成员的功能奠定基础,为亚麻分子育种提供基因资源。
1材料与方法
1.1 试验材料
亚麻品种陇亚15号、陇亚10号和黑亚14号由甘肃省农业科学院作物研究所胡麻课题组提供。供试材料陇亚15号于2021年3月种植于甘肃省农业科学院兰州试验基地。开花后采集不同时期花药以及开花后10d、20d、30d、40d、50d的种子。在培养皿中萌发陇亚15号种子,10d后选取生长一致的幼苗移至1/2MS液体培养基中,3d后进行胁迫处理同时更换新的培养液。分别对亚麻幼苗进行盐胁迫(150mmol·L-1氯化钠)、聚乙二醇处理(20%PEG)、吲哚乙酸处理(15µmol·L-1IAA)、萘乙酸处理(5µmol·L-1NAA)、赤霉素处理(5µmol·L-1GA3)、低温胁迫(4℃处理3h)及高温胁迫(45℃处理3h),分别在处理后0h、3h、6h、9h、12h、24h、48h、72h及恢复后48h取其叶片。所有样品用液氮速冻后保存于-80℃超低温冰箱备用。
1.2 PLA1基因家族的鉴定
从NCBI下载亚麻基因组序列(登陆号为QMEI02000000),从网址https://doi.org/10.6084/m9.figshare.13614311.v3下载CDS序列,蛋白质序列及gff文件。从网址http://www.phytozome.net/下载拟南芥、玉米、水稻、大豆、蓖麻及木薯全基因组蛋白序列,水稻及玉米等已知MATL/PLA1蛋白序列从NCBI获取(登录号分别为XP_015630145.1和XP_008656522.1)。利用Blastp,将已知PLA1蛋白序列与亚麻、拟南芥、玉米、水稻、大豆、蓖麻及木薯全基因组蛋白序列进行比对,之后用Pfam(http://pfam.xfam.org/search#searchBatchBlock)进行结构域确认,剔除不含PLA1蛋白典型结构域的序列。
1.3 LuPLA1基因家族染色体定位、复制分析及共线性分析
根据LuPLA1基因在染色体上的位置对其进行命名,并利用MapGenetoChromosome(http://mg2c.iask.in/mg2c_v2.0/)绘制LuPLA1家族成员的染色体定位图谱。利用MCScanX分析其复制事件,之后采用DnaSP6计算基因对之间的非同义替换率(Ka)、同义替换率(Ks)及Ka/Ks比值,利用Ka/Ks比值分析选择压力。基因对之间的分歧时间(millionyearsago,MYA)用Mya=Ks/(2×6.1×10-9)´10-6计算[26]。利用TBtools分析亚麻基因组内及亚麻与拟南芥、大豆、木薯、水稻及玉米之间的共线性并进行可视化。
1.4 LuPLA1蛋白理化性质及结构分析
利用ExPASyProtParam(http://web.wxoasy.org/protparam/)[27]对LuPLA1蛋白的分子量、等电点、氨基酸组成等理化性质进行预测;利用Plant-mPloc[28](http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)对LuPLA1蛋白的亚细胞定位进行预测;同时使用ExPASyProtScale(https://web.expasy.org/protscale/)预测LuPLA1蛋白的亲疏水性;利用SOPMA[29](https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)预测LuPLA1蛋白的二级结构。
1.5 LuPLA1蛋白系统进化分析
利用MEGA7.0软件对亚麻、拟南芥、玉米、水稻、大豆、蓖麻及木薯的PLA1蛋白序列进行多序列比对,随后采用邻接法(Neighbor-Joiningmethod)构建系统进化树,Bootstrap校验参数为1000次。
1.6 LuPLA1家族基因结构及蛋白质保守基序分析
利用GSDS(http://gsds.cbi.pku.edu.cn/)进行LuPLA1基因结构分析及可视化;利用MEME在线软件(http://alternate.meme-suite.org/tools/meme)分析LuPLA1蛋白的保守基序,预测值设置为25个,最后采用TBtools对保守基序进行可视化绘图。
1.7 LuPLA1基因家族成员启动子分析
提取LuPLA1基因转录起始位置上游2000bp作为启动子序列,通过PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析顺式作用元件。
1.8 LuPLA1基因表达分析
利用Primer5.0软件设计荧光定量引物(附表1),由于LuPLA1-2与LuPLA1-4、LuPLA1-3与LuPLA1-11、LuPLA1-7与LuPLA1-10、LuPLA1-8与LuPLA1-9、LuPLA1-12与LuPLA1-14序列相似性分别达到了94.07%、97.19%、89.74%、96.20%和95.58%,无法设计特异性引物,故分别共用一对定量引物。采用RNA提取试剂盒(BIOMGA)提取所采样品总RNA,并通过琼脂糖凝胶电泳和NanoDrop-2000对RNA的浓度和质量进行检测,随后用Prime-ScriptII试剂盒(TaKaRa)反转录合成cDNA第一条链,保存于-20℃冰箱用于qRT-PCR。以GAPDH作为内参基因,使用2×SYBRMixture(Biomiga)荧光定量试剂盒进行qRT-PCR。每个样品3次重复,采用2-ΔΔCt方法计算目标基因的相对表达量[30],最后利用GraphPadPrism8.0.1软件作图。本研究所用转录组数据从NCBI下载(登录号见附表2),利用TBtools绘制热图。
2 结果与分析
2.1 LuPLA1基因家族的鉴定、染色体定位及共线性分析
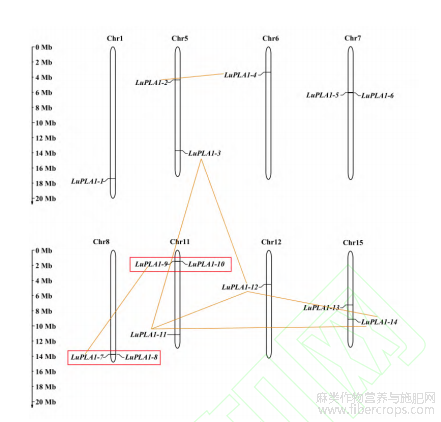
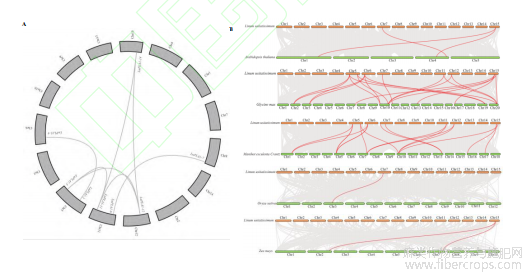
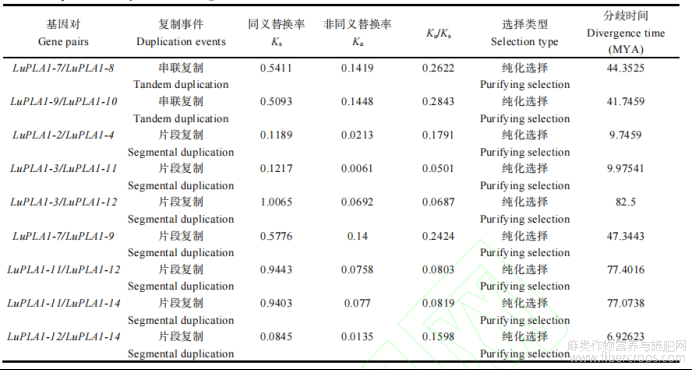
通过Blastp比对、Pfam数据库进行结构域确认,在亚麻、拟南芥、玉米、水稻、大豆、蓖麻及木薯中分别鉴定到14、21、15、20、41、18及35个PLA1成员。根据亚麻PLA1基因在染色体上的位置,将其依次命名为LuPLA1-1~LuPLA1-14。LuPLA1基因染色体定位结果显示,14个LuPLA1基因分布于8条染色体上,呈不均匀分布(图1)。对LuPLA1基因进行复制分析发现,有2对基因发生串联复制,分别为LuPLA1-7和LuPLA1-8、LuPLA1-9和LuPLA1-10;7对基因发生片段复制,分别为LuPLA1-2和LuPLA1-4、LuPLA1-3和LuPLA1-11、LuPLA1-3和LuPLA1-12、LuPLA1-7和LuPLA1-9、LuPLA1-11和LuPLA1-12、LuPLA1-11和LuPLA1-14、LuPLA1-12和LuPLA1-14(表1)。计算上述基因对的Ka/Ks比值进行选择压力分析发现,发生复制的基因对Ka/Ks比值在0.05~0.3之间,均小于1,表明这些LuPLA1基因在进化过程中均受到纯化选择。对复制事件发生的分歧时间进行计算发现,LuPLA1发生串联复制的分歧时间分别为4174.59和4435.25万年前,发生片段复制的分歧时间最短和最长分别在692.623和8250万年前。分析LuPLA1基因在亚麻基因组内部的共线性关系,显示发生片段复制的7对LuPLA1基因存在共线性关系(图2-A)。为进一步解析LuPLA1家族的进化机制,对单子叶植物玉米、水稻,双子叶植物拟南芥、大豆、木薯、蓖麻与亚麻分别进行共线性分析。结果显示分别有8个、9个、2个、1个和1个LuPLA1基因与GmPLA1、MePLA1、AtPLA1、ZmPLA1和OsMATL具有共线性,与RcPLA1无共线性,与上述作物形成的直系同源基因对分别为20对、17对、3对、2对和1对(图2-B)。此外,LuPLA1-1、LuPLA1-7、LuPLA1-LuPLA1-9及LuPLA1-10与以上作物均无共线性,其中大部分为串联重复基因。
图1 LuPLA1基因染色体定位
图2 亚麻基因组内LuPLA1基因共线性及其与其他物种间的共线性
表1 亚麻LuPLA1基因复制分析
2.2 LuPLA1蛋白理化性质分析
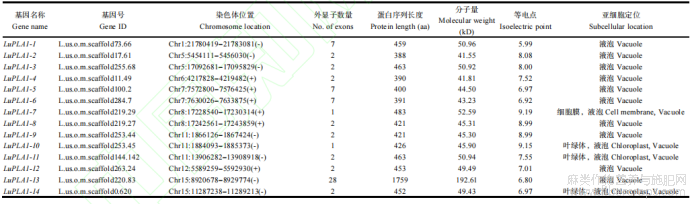
亚麻LuPLA1蛋白理化性质分析如表2所示。结果表明,LuPLA1蛋白序列长度在388~1759aa之间;LuPLA1-13分子量为192.61kD,其他LuPLA1蛋白质均在40~60kD之间;蛋白质等电点(PI)介于5.99(LuPLA1-1)与9.19(LuPLA1-7)之间,所有LuPLA1蛋白均为亲水性蛋白(附图1)。亚细胞定位结果表明,10个LuPLA1蛋白定位于液泡,3个定位于叶绿体和液泡,还有1个定位于细胞膜和液泡(表2)。对LuPLA1蛋白二级结构进行预测,结果显示所有LuPLA1蛋白主要由α-螺旋、β-转角、无规则卷曲和延伸链组成(附图2)。
表2 LuPLA1家族成员基本特征
2.3 LuPLA1家族成员系统进化分析
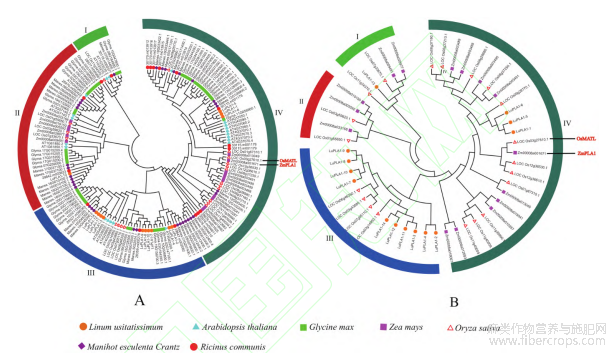
将亚麻、拟南芥、水稻、玉米、蓖麻、大豆及木薯PLA1家族成员的蛋白序列进行多序列比对并进行系统进化分析(图3-A)。结果表明,PLA1家族成员可以分为四大支,各分支中均含有单子叶和双子叶植物PLA1,且各分支成员数量具有明显差异,其中第II分支和第III分支成员数分别为40和42个,分别含有1个和10个LuPLA1;第I分支成员最少(8个),且无LuPLA1的存在;第IV分支成员最多(74个),含有全部7个物种PLA1成员,且具有3个LuPLA1。LuPLA1-1、LuPLA1-5及LuPLA1-6与单倍体诱导基因ZmPLA1和OsMATL都在第IV分支[23-24],又对亚麻、玉米及水稻PLA1家族成员单独进行系统进化分析(图3-B),发现LuPLA1-1、LuPLA1-5及LuPLA1-6与单倍体诱导基因ZmPLA1和OsMATL也分布于同一分支,LuPLA1-1、LuPLA1-5及LuPLA1-6为ZmPLA1和OsMATL的同源基因,所以初步推测它们可能与单倍体诱导相关。
图3 不同物种PLA1家族系统进化树
2.4 LuPLA1家族基因结构与蛋白质Motif分析
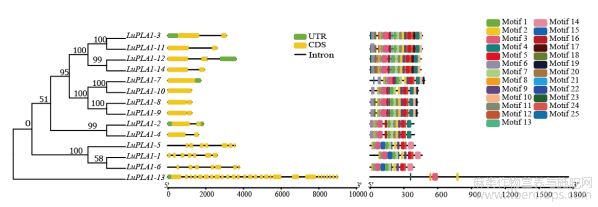
基因结构分析结果显示,除LuPLA1-7和LuPLA1-10外,其他成员都具有内含子且多数含有1个内含子,其中LuPLA1-13所含内含子最多(27个)(图4)。结合LuPLA1系统进化分析,发现亲缘关系较近的各成员之间具有相似的外显子和内含子结构,亲缘关系较远的各成员间内含子和外显子结构有明显差异。LuPLA1蛋白保守基序分析结果表明,14个LuPLA1蛋白共鉴定到25个Motif,其中LuPLA1-13含有的Motif最少,只具有4个Motif。Motif2、Motif3和Motif8存在于所有LuPLA1成员,除LuPLA1-13外,Motif5、Motif7、Motif9和Motif12存在于其他13个LuPLA1蛋白中。整体来看,除LuPLA1-13外,其他LuPLA1成员均含有10个以上Motif(图4)。
图4 LuPLA1基因家族的系统进化树、保守基序及基因结构分析
2.5 LuPLA1基因家族启动子顺式作用元件分析
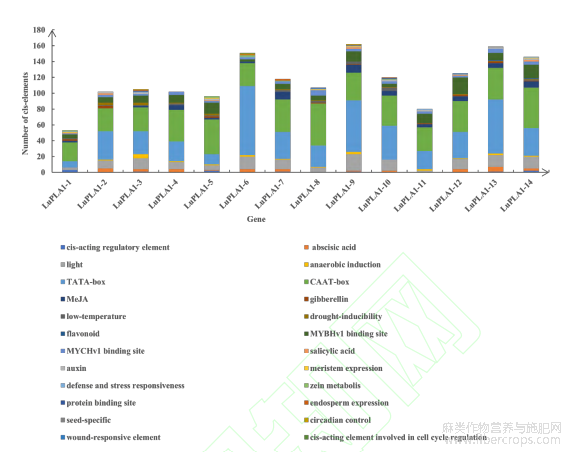
对LuPLA1基因家族各成员启动子顺式元件进行分析(图5)发现,所有LuPLA1基因都含有核心元件CAAT-box、TATA-box、光响应元件(如TCT-motif、Sp1、ATCT-motif、ACA-motif、AE-box、chsCMA1a和ACE等)、MYB结合位点(如CCAAT-box)及MYC结合位点;LuPLA1启动子区还含有四种激素响应元件,分别为脱落酸(ABA)响应相关元件(如ABRE)、茉莉酸甲酯(MEJA)响应相关元件(如CGTCAmotif,TGACG-motif)、赤霉素(GA3)响应相关元件(如P-box、TATC-box、GARE-motif)和生长素(Auxin)响应相关元件(如TGA-element)。此外,LuPLA1启动子区还含有2种与逆境胁迫相关的响应元件,分别为低温响应元件(如LTR)和干旱响应元件(如MBS),这2种元件均存在于10个LuPLA1中。大多数LuPLA1启动子区含有厌氧诱导响应元件(如ARE),少数LuPLA1启动子区还含有独特的作用元件,如LuPLA1-13含有种子特异性元件(如RY-element),LuPLA1-12含有损伤响应元件(如WUN-motif),LuPLA1-6含有细胞周期调节相关元件(如MSA-like)。
图5 LuPLA1基因家族各成员启动子区顺式作用元件
2.6 LuPLA1基因表达模式分析
2.6.1 转录组数据分析LuPLA1基因在不同品种、不同器官中的表达模式
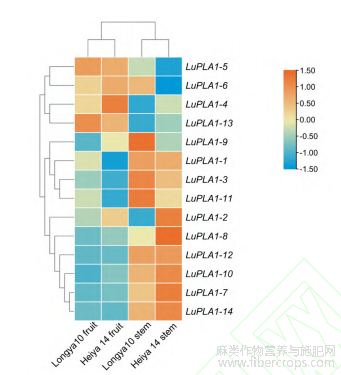
转录组数据分析结果表明,除LuPLA1-1在黑亚14号果实,LuPLA1-5在黑亚14号茎中无表达外,其他基因在不同品种、不同器官中均有表达(图6)。LuPLA1-1、LuPLA1-3、LuPLA1-7、LuPLA1-8、LuPLA1-10、LuPLA1-11、LuPLA1-12和LuPLA1-14在2个品种茎中的表达量均高于在果实中的表达量,其中LuPLA1-7、LuPLA1-8和LuPLA1-10在纤维用品种黑亚14号中的表达量高于油用品种陇亚10号;而LuPLA1-4、LuPLA1-5和LuPLA1-13在2个品种的果实中表达量均高于茎中的表达量,其中LuPLA1-5在陇亚10号中的表达量高于黑亚14号。
图6 LuPLA1基因在不同品种,不同器官中的表达量热图
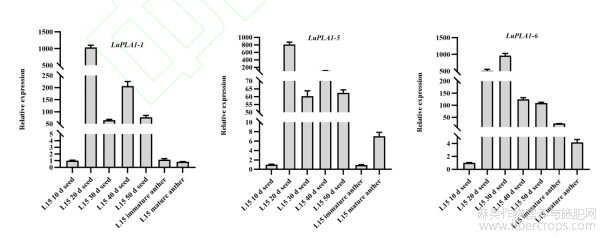
2.6.2 LuPLA1在不同发育阶段种子及花药中的表达模式
qRT-PCR分析结果(附图3)显示,LuPLA1-1在开花后30d种子中表达量最高,在开花后10d种子中表达量最低;LuPLA1-5和LuPLA1-6在开花后20d种子中的表达量最高,LuPLA1-5在成熟花药中表达量最低,LuPLA1-6在未成熟花药中表达量最低。考虑到系统进化分析中LuPLA1-1、LuPLA1-5、LuPLA1-6与单倍体诱导基因ZmPLA1和OsMATL为同源基因,而LuPLA1-1、LuPLA1-5、LuPLA1-6的表达模式与其同源基因ZmPLA1和OsMATL不同,初步推测PLA1在双子叶作物与单子叶作物中具有不同的功能。
2.6.3 LuPLA1在胁迫处理下的表达模式
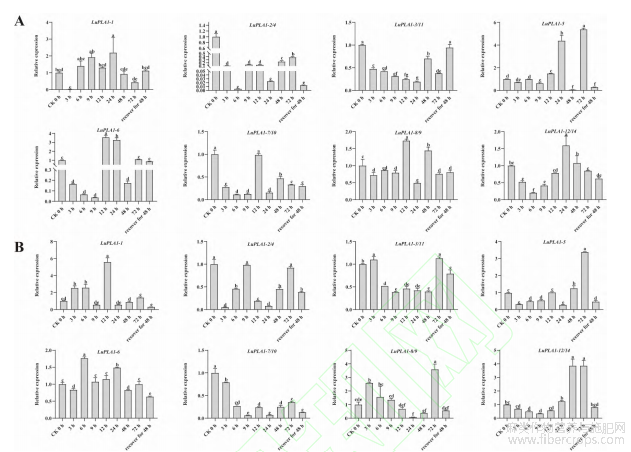
为进一步解析LuPLA1基因家族各成员的功能,利用qRT-PCR分析14个LuPLA1成员在非生物胁迫(IAA、NAA、GA3、NaCl、PEG、低温、高温)下的表达模式发现,除LuPLA1-13外,其余成员在逆境胁迫下均有不同程度的表达。在IAA处理下(图7-A),LuPLA1-1在不同时期的表达量整体高于对照,且在24h时达到最高,相较对照上调了2倍以上;LuPLA1-2/4、LuPLA1-3/11和LuPLA1-7/10的表达量较对照呈下调趋势,且表达量整体较低;其他LuPLA1的表达量在某些特定时间高于对照,如LuPLA1-5、LuPLA1-6、LuPLA1-8/9、LuPLA1-12/14的表达量分别在处理后72h、12h、12h、24h时达到峰值,各自为对照的5.38、3.54、1.73、1.31倍,但整体较对照呈下降趋势。
在NAA处理下(图7-B),LuPLA1-1的表达量在12h时差异最大,较对照上调了将近6倍,在恢复48h时表达量最低;LuPLA1-2/4、LuPLA1-7/10表达量整体低于对照;LuPLA1-3/11在3h和72h时,其表达量高于对照,其他时间段均低于对照;LuPLA1-5、LuPLA1-6和LuPLA1-8/9分别在处理后72h、6h和72h时表达量达到最高,分别为对照的3、1.7和3.5倍以上;LuPLA1-12/14在处理后48h和72h时表达量最高,为对照的3.85倍。
图7 LuPLA1基因在IAA(A)和NAA(B)胁迫下的表达模式
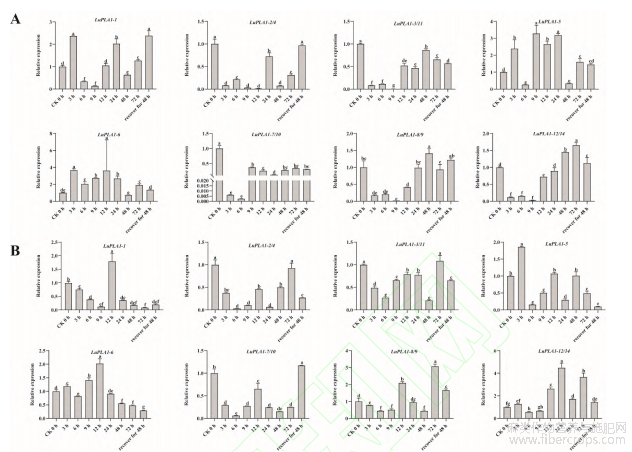
在GA3处理下(图8-A),LuPLA1-1的表达量整体表现为上调,且在3h表达量最高,是对照的2.4倍;LuPLA1-5和LuPLA1-6表达量较对照整体也呈增长趋势,最高可达到对照的3倍以上;LuPLA1-2/4、LuPLA1-3/11和LuPLA1-7/10的表达量在各时间段均低于对照,且最低时基本无表达;LuPLA1-8/9和LuPLA1-12/14的表达量较对照整体降低,但在有些时间段高于对照,分别在处理后48h和72h时达到峰值。
在NaCl处理下(图8-B),LuPLA1-1表达量在处理后12h达到峰值,为对照的1.8倍;LuPLA1-2/4的表达量在各时间段均低于对照;LuPLA1-3/11和LuPLA1-7/10的表达量呈不规律变化,分别在处理后72h时和恢复48h时达到最高,但较对照无明显增长;LuPLA1-5、LuPLA1-6、LuPLA1-8/9和LuPLA1-12/14表达量较对照既有增长也有下降,但整体呈现上调趋势,最高分别为对照的1.85、2.02、3.07及4.48倍。
图8 LuPLA1基因在GA3(A)和NaCl(B)胁迫下的表达模式
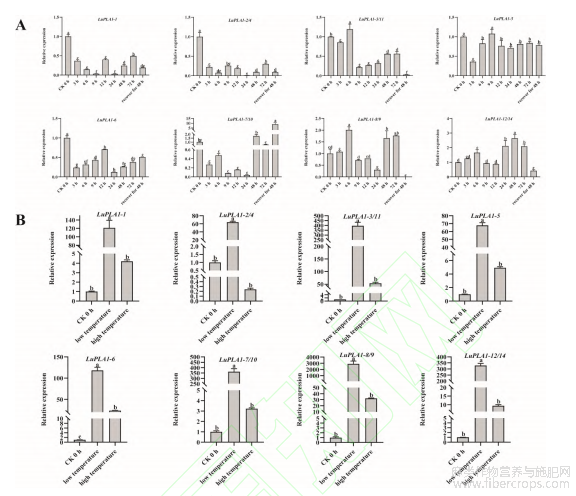
在PEG处理下(图9-A),LuPLA1-1、LuPLA1-2/4和LuPLA1-6的表达量与对照相比整体呈下调趋势,LuPLA1-3/11和LuPLA1-5的表达量仅在个别时间点较对照上调,大部分时间点均低于对照;LuPLA1-7/10的表达量较对照虽整体下调但在恢复48h时差异倍数最大,为对照的8.9倍;LuPLA1-8/9及LuPLA1-12/14表达量较对照整体呈增长趋势,且分别在处理后6h和48h时达到峰值。
在高温和低温胁迫下(图9-B),除LuPLA1-2/4在高温胁迫下表达量低于对照外,其余LuPLA1基因表达量均明显高于对照。在低温胁迫下,LuPLA1-8/9的表达量较对照差异最大,达到了对照的2906倍;在高温胁迫下,各基因表达量较低温胁迫差异程度较小,差异倍数均未超过百倍,其中LuPLA1-3/11的表达量较对照差异最大,差异倍数为53.6倍。
图9 LuPLA1基因在PEG(A)和不同温度(B)胁迫下的表达模式
3 讨论
3.1 LuPLA1基因家族进化分析
磷脂酶在植物中普遍存在,在植物生长、发育过程中扮演着重要角色,同时也是生长素信号通路的重要调控因子,参与调控植物器官发育、形态建成和抗逆应答等生理过程[31]。不同的磷脂酶存在于不同的组织器官,发挥不同的功能。其中,PLA1在植物非生物胁迫应答中具有重要作用,此外,PLA1还与单倍体诱导相关[23,32],通过对PLA1/MATL进行编辑,成功在玉米[23]、水稻[24]、小麦[25]等作物中获得了单倍体诱导系。然而目前关于PLA1的研究主要集中在单子叶植物,在双子叶植物中尚未有相关报道。
本研究共鉴定到14个LuPLA1基因,分布于8条染色体上。LuPLA1蛋白大多定位于液泡,表明PLA1家族成员主要在液泡中起作用。另外,保守基序分析结果显示,Motif2、Motif3和Motif8存在于所有LuPLA1成员,说明这3个Motif是LuPLA1家族的保守基序。基因结构分析显示,14个LuPLA1成员中有5个含有1个内含子,说明1个内含子是LuPLA1基因的主要结构形式。基因复制事件是造成基因功能多样化及特异性的重要原因之一。LuPLA1在进化中片段复制基因占全部基因的57.1%,说明片段复制在LuPLA1基因家族扩张中起主要作用,且LuPLA1的Ka/Ks比值均小于1,说明LuPLA1家族经历了纯化选择,证实了其在进化过程中的保守性。共线性分析结果显示,亚麻与双子叶作物拟南芥、大豆、木薯间分别有3、20、17对直系同源基因,与单子叶作物玉米、水稻间分别存在2对和1对同源基因,这符合14LuPLA1与其他物种PLA1的系统进化关系。LuPLA1-1、LuPLA1-7、LuPLA1-8、LuPLA1-9及LuPLA1-10
在6种植物中均未鉴定到,其中80%为串联重复基因,这些基因可能是单子叶与双子叶植物PLA1功能差异相关基因。
3.2 LuPLA1基因具有器官特异性表达模式
系统进化分析发现,3个LuPLA1成员(LuPLA1-1/5/6)与玉米、水稻单倍体诱导基因PLA1/MATL同在第IV支,亲缘关系较近,故将LuPLA1-1/5/6作为单倍体诱导候选基因进行qRT-PCR分析。结果显示LuPLA1-1/5/6在亚麻不同时期的种子、未成熟花药和成熟花药中均有表达且在未成熟花药和成熟花药中的表达量低于其在种子中的表达量,这说明PLA1在亚麻中的表达模式与其在玉米、水稻、小麦中的表达模式不同,初步推测PLA1在双子叶作物与单子叶作物中具有不同的功能,但其在双子叶作物中的具体功能还有待进一步研究。此外,转录组数据分析结果表明,有8个LuPLA1基因在2个品种茎中的表达量均大于果实中的表达量,其中LuPLA1-7、LuPLA1-8和LuPLA1-10在纤维品种中的表达量高于油用品种,初步推测其可能与茎的生长发育相关;而有3个基因在果实中的表达量较高,特别是LuPLA1-5在油用品种陇亚10号中的表达量高于黑亚14号,初步推测它与果实发育或脂肪酸含量相关。
3.3 LuPLA1基因参与调控亚麻对非生物胁迫的响应
基因表达通常受其上游启动子序列中顺式元件的调控,这些顺式元件调节不同环境条件下基因的表达行为[33]。因此,本研究对LuPLA1启动子顺式元件进行分析发现,LuPLA1家族成员均具有与光响应相关的顺式元件,大多成员还具有干旱响应、低温响应、赤霉素响应等相关元件。此外,部分家族成员具有独特的顺式作用元件。为判断启动子顺式元件分析的准确性,利用qRT-PCR分析LuPLA1家族成员在非生物胁迫下的表达模式。结果表明,在IAA处理下,LuPLA1-1的表达量较对照整体呈增长趋势,且最大差异倍数为2.19倍,推测其与IAA响应相关。相同的,在NAA处理下,LuPLA1-1和LuPLA1-6表达量较对照上调,最高分别为对照的2.56和1.76倍,推测这2个基因参与亚麻生长发育中NAA的调控;GA3处理下,LuPLA1-1、LuPLA1-5和LuPLA1-6较对照明显上调,推测其可能参与GA3调控;LuPLA1-12/14的表达量在NaCl和PEG处理下整体呈上调趋势,最大差异倍数分别为4.48和2.64倍,推测LuPLA1-12/14可能受NaCl和PEG诱导表达;低温和高温胁迫下,除LuPLA1-2/4在高温胁迫下表达量低于对照外,所有LuPLA1基因在高温、低温胁迫下表达量均高于对照且差异倍数较大,最大达到了2000倍以上,表明LuPLA1基因与低温和高温响应相关。总的来说,在不同胁迫处理下各成员具有不同的表达模式,表明这些基因参与调控亚麻对激素和非生物逆境胁迫的响应,与顺式作用元件分析结果一致,但具体功能还需进一步研究。
4 结论
本研究在亚麻基因组中共鉴定到14个LuPLA1家族成员,对其理化性质、基因结构、顺式作用元件、种内和种间共线性关系、基因复制事件及表达模式等进行分析发现,LuPLA1基因含有激素及逆境胁迫相关作用元件,且在不同处理下LuPLA1基因家族各成员具有不同的表达模式,LuPLA1-1、LuPLA1-5、LuPLA1-6与单倍体诱导基因ZmPLA1/OsMATL表达模式不同。转录组数据分析发现LuPLA1-7、LuPLA1-8和LuPLA1-10与LuPLA15可能分别参与茎和果实生长发育的调控。
参考文献
[1] Huis R, Hawkins S, Neutelings G. Selection of reference genes for quantitative gene expression normalization in flax (Linum usitatissimum L.). Bmc Plant Biol, 2010, 10: 71.
[2] 党照, 张建平, 王利民, 李闻娟, 齐燕妮, 谢亚萍, 赵玮. 胡麻新品种陇亚 15 号选育技术报告. 中国麻业科学, 2020, 42(4): 145–149. Dang Z, Zhang J P, Wang L M, Li W J, Qi Y N, Xie Y P, Zhao W. Technical report on breeding of a new flax variety Longya 15. Plant Fiber Sci China, 2020, 42(4): 145–149 (in Chinese with English abstract).
[3] 米智, 刘荔贞, 李慧, 张弘驰. 响应面法优化胡麻籽饼粕黄酮提取工艺及抗氧化活性的研究. 中国粮油学报, 2022, 37(3): 1–11. 15 Mi Z, Liu L Z, Li H, Zhang H C. Optimization of flavonoids extraction and antioxidant activity of flax seed cake by response surface method. J Chin Cereals Oils Assoc, 2022, 37(3): 1–11 (in Chinese with English abstract).
[4] 赵利, 王斌, 苗红梅, 马琴. 胡麻种质资源籽粒表型与品质性状评价及其相关性研究. 植物遗传资源学报, 2020, 21: 243–251. Zhao L, Wang B, Miao H M, Ma Q. Evaluation and correlation study on grain phenotype and quality traits of flax germplasm resources. J Plant Genet Resour, 2020, 21: 243–251 (in Chinese with English abstract).
[5] 杨阳, 王晶懋. 百合响应非生物胁迫的分子机制研究. 分子植物育种, 2018, 16: 5046–5054.Yang Y, Wang J M. Molecular mechanism of Lily response to abiotic stress. Mol Plant Breed, 2018, 16: 5046–5054 (in Chinese with English abstract).
[6] Gilles L M, Khaled A, Laffaire J B, Chaignon S, Gendrot G, Laplaige J, Bergès H, Beydon G, Bayle V, Barret P, Comadran J, Martinant J P, Rogowsky P M, Widiez T. Loss of pollen-specific phospholipase NOT LIKE DAD triggers gynogenesis in maize. EMBO J, 2017, 36: 707–717.
[7] 葛金涛, 王江英, 汤雪燕, 腾年军, 朱朋波, 孙明伟, 赵统利, 邵小斌. 百合叶片磷脂酶基因家族转录组学分析. 江苏农业科学, 2022, 50(3): 36–42. Ge J T, Wang J Y, Tang X Y, Teng N J, Zhu P B, Sun M W, Zhao T L, Shao X B. Transcriptomic analysis of the phospholipase gene family in Lily leaves. Jiangsu Agric Sci, 2022, 50(3): 36–42 (in Chinese with English abstract).
[8] Chapman K D. Phospholipase activity during plant growth and development and in response to environmental stress. Trends Plant Sci, 1998, 3: 419–426.
[9] Günther F E. Scherer. Activation of phospholipase A2, by auxin and mastoparan in hypocotyl segments from zucchini and sunflower. J Plant Physiol, 1995, 145: 483–490.
[10] Günther F, Scherer E, Arnold B. Inhibitors of animal phospholipase A2 enzymes are selective inhibitors of auxin-dependent growth. Implications for auxin-induced signal transduction. Planta, 1997, 202: 462–469.
[11] Fan L, Wang Z X. Antisense suppression of phospholipase Dα retards abscisic acid- and ethylene-promoted senescence of postharvest Arabidopsisleaves. Plant Cell, 1997, 9: 2183–2196.
[12] 苏燕南. 磷脂酶 A1 基因的克隆和原核表达. 安徽工程大学硕士学位论文, 安徽芜湖, 2013. Su Y N. Cloning and Prokaryotic Expression of Phospholipase A1 Gene. MS Thesis of Anhui Polytechnic University, Wuhu, Anhui, China, 2013 (in Chinese with English abstract).
[13] 程实. 非钙离子依赖型磷脂酶 A1 的异源表达及制备. 江南大学博士学位论文, 江苏无锡, 2021. Chen S. Heterologous Expression and Preparation of Non Calcium Ion Dependent Phospholipase A1. PhD Dissertation of Jiangnan University, Wuxi, Jiangsu, China, 2021 (in Chinese with English abstract).
[14] 安炎黄. 磷脂酶参与冬凌草甲素对拟南芥的化感潜能作用. 西北师范大学硕士学位论文, 甘肃兰州, 2019. An Y H. Phospholipase Involved in the Allelopathic Effect of Rubescens on Arabidopsis thaliana. MS Thesis of Northwest Normal University, Lanzhou, Gansu, China, 2019 (in Chinese with English abstract).
[15] Kenji M, Sachiko F, Miho I, Tadahiko K. A tomato lipase homologous to DAD1 (LeLID1) is induced in post-germinative growing stage and encodes a triacylglycerol lipase. FEBS Lett, 2004, 569: 195–200.
[16] Nishihara M, Kamata M, Koyama T, Yazawa K. New Phospholipase A 1-producing bacteria from a marine fish. Mar Biotechnol, 2008, 10: 382–387.
[17] Tavernier E, Pugin A. Phospholipase activities associated with the tonoplast from Acer pseudoplatanus cells: identification of a phospholipase A1 activity. Biochim Biophys Acta, 1995, 1233: 118–122.
[18] Ishiguro S, Kawai-Oda A, Ueda J, Nishida I, Okada K. The DEFECTIVE IN ANTHER DEHISCIENCE gene encodes a novel phospholipase A1 catalyzing the initial step of jasmonic acid biosynthesis, which synchronizes pollen maturation, anther dehiscence, and flower opening in Arabidopsis. Plant Cell, 2001, 13: 2191–2209.
[19] Noiriel A, Benveniste P, Banas A, Stymne S, Bouvier-Navé P. Expression in yeast of a novel phospholipase A1 cDNA from Arabidopsis thaliana. Eur J Biochem, 2004, 271: 3752–3764. 1617
[20] Seo Young Sam. Heterologous expression, and biochemical and cellular characterization of CaPLA1 encoding a hot pepper phospholipase A1 homolog. Plant J Cell Mol Biol, 2008, 53: 895–908.
[21] 姜惠娜, 敬松, 李晗玉, 佘木子, 张新飞, 呼天明, 付娟娟, 苗彦军. 西藏野生垂穗披碱草 EnPLA1 基因克隆与表达分析. 草地学报, 2021, 29:2141–2148. Jiang H N, Jing S, Li H Y, She M Z, Zhang X F, Hu T M, Fu J J, Miao Y J. Cloning and expression analysis of EnPLA1 gene in wild Flagellae from Xizang. Acta Agrest Sin, 2021, 29: 2141–2148 (in Chinese with English abstract).
[22] Hyun Y, Choi S, Hwang H J, Yu J, Nam S J, Ko J, Park J Y, Seo Y S, Kim E Y, Ryu S B, Kim W O, Lee Y H, Kang H, Lee I. Cooperation and functional diversification of two closely related galactolipase genes for jasmonate biosynthesis. Dev Cell, 2008, 14:183–192.
[23] Liu C X, Li X, Meng D X, Zhong Y, Chen C, Dong X, Xu X W, Chen B J, Li W, Li L, Tian X L, Zhao H M, Song W B, Luo H S, Zhang Q H, LaiJ S, Jin W W, Yan J B, Chen S J. A 4-bp insertion at ZmPLA1 encoding a putative phospholipase a generates haploid induction in maize. Mol Plant, 2017, 10: 520–522.
[24] 文钦, 贾思思, 王加峰, 黄翠红, 王慧, 陈志强, 郭涛. 水稻单倍体诱导基因 OsMATL 突变体的创制与分析. 作物学报, 2021, 47: 816–825. Wen Q, Jia S S, Wang J F, Huang C H, Wang H, Chen Z Q, Guo T. Creation and analysis of OsMATL mutant of rice haploid inducible gene. Acta Agron Sin, 2021, 47: 816–825 (in Chinese with English abstract).
[25] Liu H Y, Wang K, Jia Z M, Gong Q, Lin Z S, Du L P, Pei X W, Ye X G. Efficient induction of haploid plants in wheat by editing of TaMTL using an optimized Agrobacterium-mediated CRISPR system. J Exp Bot, 2020, 71: 1337–1349.
[26] Lynch M, Conery J S. The evolutionary fate and consequences of duplicate genes. Science, 2000, 290: 1151–1155.
[27] 张丹丹, 杨瑶君, 江纳, 付春. 黄藤 Dof 家族的全基因组鉴定及系统进化分析. 西南林业大学学报(自然科学), 2021, 41(6): 126–138. Zhang D D, Yang Y J, Jiang N, Fu C. Genome-wide identification and phylogenetic analysis of the Dof family of Rattan japonica. J Southwest For Univ (Nat Sci), 2021, 41(6): 126–138 (in Chinese with English abstract).
[28] 刘艳丽, 周媛, 曹丹, 马林龙, 龚自明, 金孝芳. 基于茶蛋白质组学数据分析植物亚细胞定位预测软件的应用. 植物科学学报, 2020, 38: 671–677. Liu Y L, Zhou Y, Cao D, Ma L L, Gong Z M, Jin X F. Application of plant subcellular localization prediction software based on tea proteomic data analysis. Plant Sci J, 2020, 38: 671–677 (in Chinese with English abstract).
[29] 冷非凡, 罗文, 李渊利, 孙尚琛, 王永刚. 嗜铁钩端螺旋菌中铁硫簇相关蛋白的生物信息学分析. 基因组学与应用生物学, 2018, 37: 5296–5303. Leng F F, Luo W, Li Y L, Sun S C, Wang Y G. Bioinformatics analysis of iron-sulfur cluster related proteins in Helicobacter ferrophila. Genomics Appl Biol, 2018, 37: 5296–5303 (in Chinese with English abstract).
[30] 唐永凯, 贾永义. 荧光定量 PCR 数据处理方法的探讨. 生物技术, 2008, (3): 89–91. Tang Y K, Jia Y Y. Discussion on data processing methods of fluorescence quantitative PCR. Biotechnology, 2008, (3): 89–91 (in Chinese with English abstract).
[31] 张群, 贾倩茹, 章文华. 磷脂介导的生长素信号转导研究进展. 南京农业大学学报, 2020, 43(2): 195–203. Zhang Q, Jia Q R, Zhang W H. Research progress in phospholipid-mediated auxin signal transduction. J Nanjing Agric Univ, 2020, 43(2): 195–203 (in Chinese with English abstract).
[32] Kelliher T, Starr D, Richbourg L, Chintamanani S, Delzer B, Nuccio M L, Green J, Chen Z Y, McCuiston J, Wang W L, Liebler T, Bullock P, Martin B. MATRILINEAL, a sperm-specific phospholipase, triggers maize haploid induction. Nature, 2017, 7639: 105–109.
[33] Zhu S H, Wang X Y, Chen W, Yao J B, Li Y, Fang S T, Lyu Y J, Li X X, Pan J W, Liu C Y, Li Q L, Zhang Y S. Cotton DMP gene family: Characterization, evolution, and expression profiles during development and stress. Int J Biol Macromol, 2021, 183: 1257–1269.
附表 1qRT-PCR引物
附表2 转录组数据登录号
附图1 亚麻PLA1家族成员的亲水性与疏水性预测
附图2 LuPLA1基因编码的蛋白质的二级结构
附图3 LuPLA1基因在陇亚15号不同时期种子及花药中的表达模式
文章摘自:赵丽蓉,李雯,王利民等.亚麻PLA1基因家族的鉴定及表达分析[J/OL].作物学报:1-20[2023-05-31].http://kns.cnki.net/kcms/detail/11.1809.S.20230519.1811.002.html.