摘 要:扩展蛋白(Expansin)作为植物细胞壁的重要组成部分,通过调节细胞壁组分间松驰度和增强细胞胞壁组分间松驰度和增强细胞壁的柔韧性,对植物的生长发育和环境抗性等方面有着至关重要的作用。为了揭示和探讨大麻扩展蛋白基因家族的组成和特征,本研究利用扩展蛋白共有的保守结构域从大麻的全基因组中筛选出32个扩展蛋白基因组成大麻扩展蛋白基因家族,分布在大麻除第4条染色体以外剩下的9条染色体上。分析其结构特征及系统发育关系,表明大麻扩展蛋白基因家族分为4个亚家族:包含19个基因的EXPA亚家族,含有7个基因的EXPB亚家族,包含5个基因的EXLB亚家族和只含有1个基因的EXLA亚家族。大麻扩展蛋白基因的氨基酸长度范围为471-212,其等电点平均值为7.48,有信号肽带领的两个结构域,大多为疏水蛋白,具有比较好的稳定性。本研究通过分析大麻扩展蛋白基因的基本信息,为以后深入研究各植物扩展蛋白基因的生物学功能和进化特性奠定了基础。
关键词:大麻;扩展蛋白基因家族;生物信息学分析;系统进化
扩展蛋白基因是位于植物细胞壁上与细胞膨大密切相关的非酶类蛋白,其功能在于使纤维素微纤维和半纤维素之间连接的氢键断裂来调节细胞壁组分间松驰度以及增加细胞壁的柔韧性(Feng et al.,2019)。扩展蛋白广泛存在于在植物细胞的生长,果实的成熟,果皮破裂,根毛的出现,花粉管进入柱头和子房,分生组织的生长等植物的生长发育过程中。因此扩展蛋白在种子萌发(Yan et al.,2014)、耐盐(Lu et al.,2013)、抗旱(Chen et al.,2016)、果实成熟(Jiang et al.,2019)和根伸长(Noh et al.,2013)等过程中都发挥了重要作用。
扩展蛋白家族是一个较原始且保守的大基因家族,据基因结构和氨基酸序列分析,表明扩展蛋白基因均来源于一个共同的祖先,即目前的扩展蛋白家族一共可以分为4个亚家族:EXPA、EXPB、EXLA和EXLB亚家族。首次在黄瓜中被发现的命名为EXPA,EXPA亚家族多作用于植物的生长发育(Choi et al.,2003;Yu et al.,2011)。而在植物花粉中被发现的EXPB在首次发现是曾经被认为是禾本科花粉过敏原,之后才被证明属于扩展蛋白家族,这种扩展蛋白亚家族经研究表明和生殖系统有着紧密的联系(Russell et al.,2008;Tabuchi et al.,2011)。近几年来,随着基因组测序的深入和基因组分析的进步,有越来越多物种的扩展蛋白基因得以鉴定分析,如拟南芥(Sampedro et al.,2006),红皮柳(杨锐霞等,2021),银杏(王瑞雪等,2021)和水稻(施杨等,2014)等。因此扩展蛋白基因在不同物种基因家族中的序列组成、基因数量和结构功能等方面都有很大的不同(李昊阳等,2014)。
大麻(Cannabis sativa L.)别名火麻,是一年生的大麻科(Cannabinaceae)大麻属(Cannabis L.)的植物(Kovalchuk et al.,2021),因其同时具有很高的药用价值和丰富的商业价值而闻名于世。大麻不仅有消炎镇痛、抗失眠、缓解精神类症状和治疗类风湿性关节病等一系列生理功能,还兼具有利尿、抗癌细胞、抗细菌真菌等药用价值。但同时大麻也可能会引起焦虑、产生认知功能障碍和降低自身的免疫力,所以这类物质在日常生活中被严格管制(杨柳秀等,2020)。而关于其扩展蛋白的研究仍然处于空白。因此,本研究以大麻的全基因组为基础,筛选大麻的扩展蛋白基因,蛋白理化性质、染色体分布、家族成员组成、基因结构、保守结构域和不同物种间共线性关系等生物信息学分析,获得得了大麻扩展蛋白基因家族的基本信息,为深入研究大麻的扩展蛋白基因的分子进化与生物学功能奠定基础。
1结果与分析
1.1大麻扩展蛋白基因家族的筛选鉴定及其理化分析
用拟南芥作为基因探针,在大麻基因组中得到32个相匹配的基因序列。利用TBtools v1.098689(https://github.com/CJ-Chen/TBtools)软件从Supercann网站(https://gdb.supercann.net/index.php/download)的数据库中对获取的PF03330和PF01357结构域对所有的大麻蛋白序列进行筛选,获得32个大麻的扩展蛋白基因(表1)。其中21个基因都与利用拟南芥作为探针获取的基因重复,因此以筛选后的基因作为目的基因进行下一步的分析。通过文献可知扩展蛋白基因家族的保守结构域在pfam中分别为DPBB_1(PF03330)和Pollen_allerg_1,且都和植物细胞的扩展有关,通过结合Pfam和smart分析其保守结构域,找到其中同时含有PF03330和PF01357两个保守结构域的基因序列,所筛选出的32个基因就是大麻中的扩展蛋白基因。
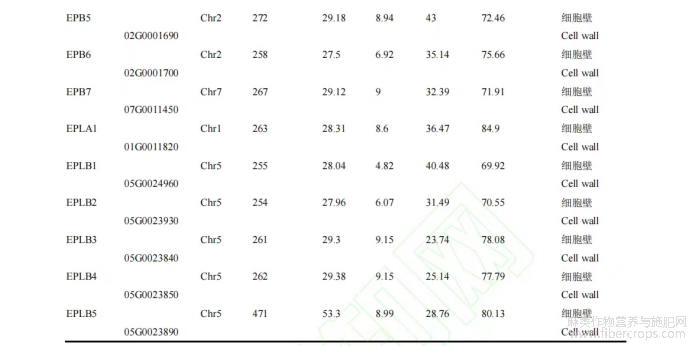
由大麻扩展蛋白基因组序列的分析可得,大麻扩展蛋白基因共可分为EXPA、EXPB、EXLA和EXLB四个亚家族。19个大麻扩展蛋白组成A亚家族,B亚家族有7个成员,LA亚家族有1个大麻扩展蛋白,LB亚家族则由5个家族成员组成。大麻的扩展蛋白基因数量与拟南芥(36)、黄瓜(35)和西瓜(32)相近(Hao et al.,2015)。大麻扩展蛋白基因是黄瓜扩展蛋白基因的0.91倍、是水稻扩展基因的0.55倍、是大白菜扩展基因的0.6倍(表2)。
大麻的32个扩展蛋白基因的分子量从23.58 kD到53.3 kD不等,平均值为31.2 kD;等电点范围从4.82到10.12不等,平均值为7.48;而其中蛋白最长的有471个,短的则有212个氨基酸残基,平均值为285.49,跨度比较大。通过不稳定指数确定大麻扩展蛋白为稳定蛋白并预测其亚细胞定位信息,由结果可知这32个大麻扩展蛋白基因都定位于细胞外,这也与已知的扩展蛋白属于细胞壁蛋白这一结论吻合。
表1 大麻扩展蛋白基因家族基本信息及特征



表2 不同植物的扩展蛋白及其 4 类亚家族数目分析
1.2大麻扩展蛋白重复基因分析及其染色体定位
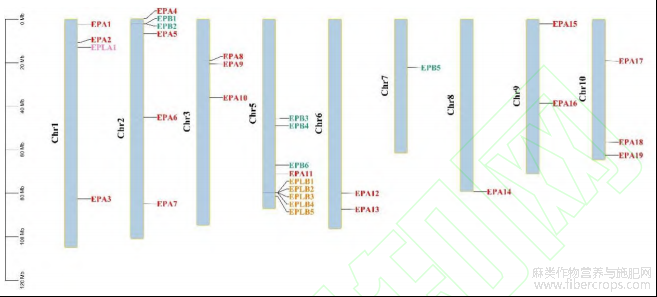
确定了大麻基因组的10条染色体后,通过Supercann网站(https://gdb.supercann.net/index.php/download)数据库获得大麻基因注释文件,利用大麻基因注释文件通过TBtools v1.098689(https://github.com/CJ-Chen/TBtools)软件中Gene Location Visualize工具进行染色体定位的绘图(图1)。结果表明,大麻的32个扩展蛋白基因分布在除了4号染色体以外的其余9条染色体上,同时可以看出大麻的扩展蛋白基因在染色体上的分布是非常分散且没有规律的(图1),其中7号和8号染色体只包含1个扩展蛋白基因,6号和9号染色体都含2个扩展蛋白基因,3号和10号染色体都含3个扩展蛋白基因,1、2、5号染色体分别含4、6、10个基因。其中EXLA亚家族的唯一的基因位于1号染色体上,2号染色体上扩展蛋白最多且EXLB亚家族全部的5个扩展蛋白基因均在2号染色体上并形成了基因簇,这是由于同源基因在同一条染色体上扩增形成的。
在同一个物种中形成许多基因的主要方式有多倍化,片段复制,反转录转座,串联重复四种方法。形成的这些基因之同的功能,但是都有相似的蛋白结构和功能,有相同的结构域,通过未知基因和已知的基因之间的比对就可以推测未知基因的功能。
通过Tbtools软件中One Step MCScanX-Super Fast对大麻扩展蛋白基因家族的片段重复和串联重复基因进行分析,并使用Tbtools软件中Advanced Circos可视化大麻扩展蛋白基因家族中的重复基因(图2)。在大麻的32条染色体中有重复的序列,分别为:第6条染色体上的基因簇中的EXPA12和第9条染色体上的EXPA16,第2条染色体上的EXPA4和第6条染色体上的EXPA13以及第9条染色体上的EXPA15形成了重复基因间的相互关系是旁系同源基因,在同一个物种中有相同的祖先基因进化而来或许会展现不同的功能,但是都有相似的蛋白结构和功能,有相同的结构域,通过未知基因和已知的基因之间的比对就可以推测未知基因的功能。
图1 大麻32个扩展蛋白基因在9条染色体的分布
图2 大麻32个扩展蛋白的共线性分析
1.3基因家族的motif分析及其结构分析
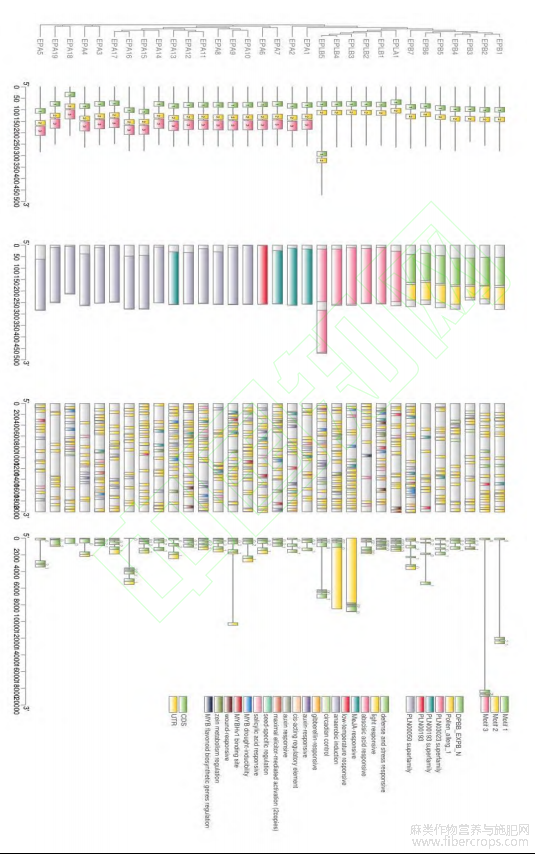
通过大麻扩展蛋白的基因结构(图3),可以看到大麻扩展基因家族中除了一个基因无内含子外,其余成员均含有外显子和内含子,但成员之间仍然有明显差异,这体现在外显子和内含子的数量和位置不同上。基因家族成员中所具有的内含子相对较少,其中最多的含有4个内含子,最少的为0。基因家族中只有1个成员不含内含子,约占整个家族的3%,有2个基因只含有1个内含子,约占整个家族的6%,有18个基因含有2个内含子,约占整个家族的56%,有8个基因含有3个内含子,占整个家族的25%,剩下的3个基因含有4个内含子,占整个家族的9%。同时也表明了大麻扩展蛋白基因家族成员具有的基因亚家族特异性显而易见。每个亚家族的平均内含子个数为EXPA亚族2.1个,EXPB亚族2.57个,EXPLA亚族4个和EXPLB亚族2.6个。
使用Tbtools软件中Simple MEME Wrapper对筛选的32个大麻扩展蛋白家族成员进行分析,并通过Tbtools软件中Gene Structure View生成motif保守分析图(图3),通过在线网站MEME分析得到大麻扩展蛋白基因家族得3个保守基序(图4)。
结果可得,在大麻的扩展蛋白基因中一共找到了3个预测的motif,基因家族中的每个成员中分别含有2-3个的motif。大麻扩展蛋白基因家族中的不同基因都有不同的motif,这也说明了扩展蛋白基因家族中基因的多样性。但在所有的大麻扩展蛋白基因都有几个相同的motif以及夹杂着特有的motif结构域。PF03330和结构域PF01357包含motif1和motif2。所以大麻扩展蛋白基因的四个亚家族都含有motif1和motif2,而属于EXPA亚家族的21个成员除了都含有motif1和motif2,还含有该家族所特有的motif3,这是其他三个家族所没有的。其中共有的motif1、2在不同的大麻扩展蛋白基因中都定位到基本相同的位置,因此可以推测得出共同的motif1、2就是大麻扩展蛋白基因的功能基础。
图3 大麻扩展蛋白家族进化树,保守基序,保守结构域,启动子顺式作用元件及基因结构
注:顺式元件分布从上到下依次为参与防御和应激反应的顺式作用元件,参与光响应的顺式作用元件,响应脱落酸的顺式作用元件,响应茉莉酸甲酯的顺式作用元件,参与低温反应的顺式作用元件,厌氧诱导的顺式作用元件,参与昼夜节律调控顺式作用元件,参与赤霉素反应的顺式作用元件,参与生长素反应顺式作用元件,最大激发子介导的激活的顺式作用元件,种子特异性调控中的顺式作用元件,参与水杨酸反应性顺式作用元件,MYB干旱诱导性顺式作用元件,MYB结合位点顺式元件,玉米醇溶蛋白代谢调控中的顺式作用元件
1.4基因家族的进化特性
通过软件MEGA v7.0对得到的大麻和拟南芥的扩展蛋白基因进行序列比对构建N-J进化树(图5),分析扩展蛋白家族成员间的进化关系,并通过拟南芥扩展蛋白基因的各亚家族和大麻扩展基因在染色体上的位置对其进行命名。由进化树分析,扩展蛋白基因家族的32个成员被分为四个亚家族:EXPA,EXPB,EXLA和EXLB。其中EXPA亚家族的占比最高,在32个扩展蛋白基因中共有19个,占比约60%。而EXLA亚家族基因占比最小,只有1个亚家族成员基因。大麻扩展蛋白中相应的亚家族基因占比率同其他作物的扩展蛋白中基因亚家族的占比率是相似的。
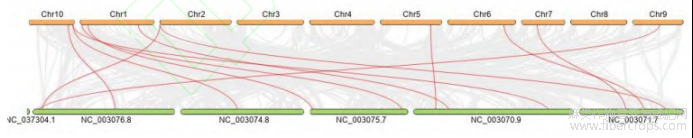
同时为了分析扩展蛋白基因家族在不同物种间的进化关系,利用拟南芥和大麻的同源关系,将得到的大麻基和拟南芥的扩展蛋白基因组之间的进行共线性关系(图6),共有12条亮线,代表有12个扩展蛋白基因的同源基因对,这一结果表明在单子叶与双子叶植物分化之前基因家族的基本特征就已经形成了。还可以看出成树之后相同的基因家族聚合到了一起而并非是同种作物之间的基因由于相似较高而聚合到一起,这一结果表明直系同源基因的相似程度要比旁系同源基因的相似性高。
图5 大麻和拟南芥扩展蛋白基因系统发育树
图6 拟南芥和大麻的共线性关系
1.5基因家族的顺式元件分析
取大麻扩展蛋白基因序列上游2kb的序列,预测大麻扩展蛋白基因家族成员的顺式作用元件(图3)。由光响应元件、厌氧诱导元件、脱落酸响应元件、水杨酸响应元件、响应茉莉酸甲酯的顺式作用元件等主要顺式元件组成。其中大麻扩展蛋白家族成员中都存在着光响应元件,它的元件数量也是所有家族成员中元件数量最多的,共有460个。其次是预测到的厌氧诱导的顺式作用元件,数量为72个,响应茉莉酸甲酯的顺式作用元件有48个,响应脱赤霉素的顺式作用元件12个。含量最少的是最大激发子介导的激活的顺式作用元件,只有2个。而种子特异性调控中的顺式作用元件也含有4个,分别存在于EXPA15、EXPA13、EXPA11和EXPB2的启动子区。
2讨论
本研究从大麻的全基因组中筛选出了32个基因组成大麻扩展蛋白基因家族,其基因家族成员分布于9条染色体上,通过亚细胞定位分析表明均定位于细胞壁上,与拟南芥等作物的扩展蛋白基因定位结果十分相符。大麻扩展蛋白作为植物细胞壁中十分重要组成部分且广泛存在于各细胞器官中,参与植物生长发育等多个生理过程(郝西等,2015)。扩展蛋白在水稻(Shin et al.,2005)、玉米(Zhang et al.,2014)、大白菜(Krishnamurthy et al.,2015)、陆地棉(张奇艳等,2019)、黄瓜(郝西等,2015)等作物中分别存在34、36、39、46、35个家族成员,可以看出不同物种中的扩展蛋白的数量有比较大的差别。由拟南芥扩展蛋白基因家族成员系统进化分成的EXPA、EXPB、EXLA和EXLB四个亚家族中,只有EXPB4、EXPB6、EXPLB1和EXPLB2等电点小于7,说明这四个基因可能带有编码碱性蛋白质。扩展蛋白家族中各亚家族的占比也被发现存在着明显不同。例如,EXPB亚家族的成员比例在被子植物和裸子植物中差距过大,在大白菜的玉米等被子植物中分别达到16.98%和54.55%,而属于裸子植物的银杏在EXPB亚家族的占比只有3.57%(王瑞雪等,2021)。
通过基因结构域分析大麻扩展蛋白基因家族的32个家族成员都包含PEBP结构域,只有一个家族成员不含内含子,其余的家族成员均含有一个或多个内含子。而在黄瓜、银杏和红皮柳的扩展蛋白中只有黄瓜扩展蛋白基因家族中有一个基因家族成员不含内含子。再次说明大麻扩展家族基因分布广泛,植物中有内含子的扩展蛋白基因占比非常高。同时也在基因家族中发现了许多会参与到光响应的顺式作用元件、种子特异性调控中的顺式作用元件、根特异性顺式作用调控元件和玉米醇溶蛋白代谢调控中的顺式作用元件等,表明该基因家族广泛的影响着大麻的生长发育过程,并在大麻的种子萌发和根和茎的发育等方面有着十分重要的调控作用(Zimmermann et al.,2010);而赤霉素、水杨酸、响应茉莉酸甲酯的诱导类顺式作用元件的发现,可推测出大麻扩展蛋白基因会受到各种激素信号分子的诱导(Wang et al.,2009)。
本次研究通过对筛选出的32个大麻扩展蛋白基因进行分类并命名,且对大麻扩展蛋白基因家族开展基因家族的理化性质、保守基序、基因结构、系统进化、物种内及物种间共线性分析和顺式作用元件分析等生物信息学分析。研究结果对大麻扩展基因家族成员的鉴定和深入解析具有深远的意义。
3材料与方法
3.1大麻扩展蛋白基因家族成员的筛选
从TAIR网站得到拟南芥扩展蛋白家族的基因序列和蛋白序列,将其作为基因探针(Goodstein et al.,2012)。从Supercann网站(https://gdb.supercann.net/index.php/download)获得大麻的扩展蛋白家族的全基因组和基因注释文件。利用TBtools(https://github.com/CJ-Chen/TBtools)软件(Chen et al.,2018),在已经得到的大麻蛋白数据库中利用结构域模型PF01357和PF03330进行搜索,得到同时包含两个结构域模型的蛋白序列,筛掉相似度比较小的和只含有一个结构域的序列,将两种方法进行比对重合得到候选序列(郝西等,2015)。
将得到的大麻扩展蛋白基因上传至网站Pfam隐马尔可夫模型进行保守的结构域的验证,排除不包含DPBB_1和Pollen_allerg_1的结构域以及重复序列,最终确定大麻扩展蛋白基因家族成员和每个家族成员的基因序列(施杨等,2014)。
3.2基因家族蛋白性质分析及亚细胞定位
通过网站Prot Paramtool分析大麻扩展蛋白基因家族每个成员的物理与化学参数;并用网站Euk-mPLoc2.0来预测每个大麻扩展蛋白基因家族成员的亚细胞定位信息。
3.3家族基因的进化分析
使用软件Clustal X2对大麻和拟南芥的所有扩展蛋白基因进行多序列对比分析,通过亚家族和染色体位置为标准对大麻扩展蛋白基因家族的32名成员进行命名,使用MEGA7.0程序中的邻接法构建Neighbor-joiningTree表征大麻与拟南芥的扩展蛋白基因家族系统进化树(Gascuel and Steel,2006;沙伟等,2011),通过进化树看到不同基因间的远近关系(Lozano et al.,2015)。
3.4大麻和拟南芥扩展蛋白基因家族共线性分析
从Supercann网站获得大麻的全基因组及注释文件,从NCBI网站获得拟南芥的全基因组序列及注释文件。TBtools对两者进行可视化的共线性关系分析,以此发现拟南芥和大麻扩展蛋白基因家族间的同源基因对。
3.5基因家族染色体定位及重复基因分析
大麻扩展蛋白基因染色体定位分析使用TBtools(https://github.com/CJ-Chen/TBtools)软件中Gene Location Visualize工具进行绘图,从而更直观地看到扩展蛋白基因在染色体上的分布(李昊阳等,2014)。
基因定位之后通过Tbtools软件中One Step MCScanX-Super Fast对大麻扩展蛋白基因家族的片段重复和串联重复基因进行分析。用Tbtools软件中Advanced Circos作图可视化大麻扩展蛋白基因家族中的重复基因,以此分析进化过程中扩展蛋白基因的复制,要求重复基因的相似度高于75%。
3.6基因家族蛋白结构域的motif分析和保守motif预测
大麻扩展蛋白的保守motif预测通过Tbtools软件中Simple MEME Wrapper分析完成,其中最大模体数设置为10,其他参数默认。通过在线网站MEME得到大麻扩展蛋白基因家族得3个保守基序。
3.7基因家族的结构分析
大麻扩展蛋白基因结构分析使用TBtools(https://github.com/CJ-Chen/TBtools)软件中的Gene Structure View工具绘图得到大麻扩展蛋白家族中族内进化树+Motifs+结构域+启动子顺势作用元件(上游2kb序列)+基因结构的多图合一大图。
参考文献
[1]Chen C.J., Xia R., and Chen H., 2018, TBtools, a Toolkit for Biologists integrating various HTS-data handling tools with a user-friendly interface, 23: 56-58.
[2]Chen Y.K., Zhang B., Li C.H., Lei C.X., Kong C.Y., Yang Y., and Gong M., 2019, A comprehensive expression analysis of the expansin gene family in potato (Solanum tuberosum) discloses stress-responsive expansin-like B genes for drought and heat tolerances, PLoS One, 14(7): e0219837.
[3]Choi D., Lee Y., Cho H.T., and Kende H., 2003, Regulation of expansin gene expression affects growth and development in transgenic rice plants, Plant Cell, 15(6): 1386-1398.
[4]Feng X., Xu Y.Q., Peng L.N., Yu X.Y., Zhao Q.Q., Feng S.S., Zhao Z.Y., Li F.L., and Hu B.Z., 2019, TaEXPB7-B, a β-expansin gene involved in low-temperature stress and ab-scisic acid responses, promotes growth and cold resistance in Arabidopsis thaliana, J. Plant Physiol., 9(240): 153004.
[5]Gascuel O., and Steel M., 2006, Neighbor-joining revealed, Mol. Biol. Evol., 23(11): 1997-2000.
[6]Goodstein D.M., Shu S., and Howson R., 2012, Phytozome: a comparative platform for green plant genomics, Nucleic Acids Research, 40: 1178-1186.
[7]Hao X., Li X.Y., La G.X., Dai D.D., and Yang T.G., 2015, Identification and bioinformatics analysis of cucumber expansin gene family, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 13(10): 2280-2289. (郝西, 理向阳, 腊贵晓, 代丹丹, 杨铁钢, 2015, 黄瓜扩展蛋白基因家族的鉴定与生物信息学分析, 分子植物育种, 13(10): 2280-2289.)
[8]Jiang F., Lopez A., Jeon S., De Freitas S.T., Yu Q., Wu Z., Labavitch J.M., Tian S., Powell A., and Mitcham E., 2019, Disassembly of the fruit cell wall by the ripening-associated polygalacturonase and expansin influences tomato cracking, Hortic. Res., 6: 17.
[9]Kovalchuk I., Pellino M., and Rigault P., 2020, The genomics of Cannabis and its close relatives, Annu. Rev. Plant Biol., 71: 713-739.
[10]Krishnamurthy P., Hong J.K., Kim J.A., Jeong M.J., Lee Y.H., and Lee S.I., 2015, Genome-wide analysis of the expansin gene superfamily reveals Brassica rapa-specific evolutionary dynamics upon whole genome triplication, Mol. Genet. Ge-nomics, 290(2): 521-530.
Li H.Y., Shi Y., Ding Y.N., and Xu J.C., 2014, Bioinformatics analysis of expansin gene family in poplar genome, Beijing Linye Daxue Xuebao (Journal of Beijing Forestry University), 36(2): 59-67. (李昊阳, 施杨, 丁亚娜, 徐吉臣, 2014, 杨树扩展蛋白基因家族的生物信息学分析, 北京林业大学学报, 36(2): 59-67.)
[11]Lozano R., Hamblin M T., and Prochnik S., 2015, Identification and distribution of the NBS-LRR gene family in the Cassava genome, BMC Genomics, 16(1): 360.
[12]Lu P.T., Kang M., Jiang X.Q., Dai F.W., Gao J.P., and Zhang C.Q., 2013, RhEXPA4, a rose expansin gene, modulates leaf growth and confers drought and salt tolerance to Arabidop-sis, Planta, 237(6): 1547-1559.
[13]Noh S.A., Lee H.S., Kim Y.S., Paek K.H., Shin J.S., and Bae J.M., 2013, Down-regulation of the IbEXP1 gene enhanced storage root development in sweetpotato, J. Exp. Bot., 64(1): 129-142.
[14]Russell S.D., Bhalla P.L., and Singh M.B., 2008, Transcrip-tome-based examination of putative pollen allergens of rice (Oryza sativassp. japonica), Mol. Plant, 1(5): 751-759.
[15]Sampedro J., Carey R.E., and Cosgrove D.J., 2006, Genome his-tories clarify evolution of the expansin superfamily: new in-sights from the poplar genome and pine ESTs, J. Plant Res., 119(8): 11-21.
[16]Sha W., Qin R.F., Meng L.J., Xin Y.M., Ma T.Y., and Zhang M.J., 2019, Cloning and expression analysis of drought resistance related gene RcAOS in sphagnum mongolicum, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 18(4): 1131-1137. (沙伟, 秦瑞峰, 孟灵军, 信雨萌, 马天意, 张梅娟, 2019, 砂藓(Racomitrium canescens)抗旱相关基因 RcAOS 的克隆及表达分析, 分子植物育种, 18(4): 1131-1137.)
[17]Shi Y., Xu X., Li H.Y., Xu Q., and Xu J.C., 2014, Bioinformatics analysis of the expansin gene family in rice, Yichuan (Hereditas), 36(8): 809-820. (施杨, 徐筱, 李昊阳, 徐倩, 徐吉臣, 2014, 水稻扩展蛋白家族的生物信息学分析, 遗传, 36(8): 809-820.)
[18]Shin J.H., Jeong D.H., Park M.C., and An G., 2005, Characteri-zationand transcriptional expression of the alphaexpansin gene family in rice, Mol. Cells, 20(2): 210-218.
[19]Tabuchi A., Li L.C., and Cosgrove D.J., 2011, Matrix solubilization and cell wall weakening by β-expansin (group-1 allergen) from maize pollen, Plant. J., 68(3): 546-559.
[20]Wang L., Wang Z., and Xu Y., 2009, OsGSR1 is involved in crosstalk between gibberellins and brassinosteroids in rice, Plant J., 57(3): 498-510.
[21]Wang R.X., Yang R.X., Yin P., Liu J.F., and Xu J.C., 2021, Identification and characterization of the expansin gene in Ginkgo biloba, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 19(6): 1741-1749. (王瑞雪, 杨锐霞, 尹鹏, 刘家福, 徐吉臣, 2021, 银杏扩展蛋白基因的鉴定与特征分析, 分子植物育种, 19(6): 1741-1749.)
[22]Yan A., Wu M.J., Yan L.M., Hu R., Ali I., and Gan Y.B., 2014, AtEXP2 is involved in seed germination and abiotic stress response in Arabidopsis, PLoS One, 9(1): e85208.
[23]Yang L.X., Li C.R., and Gao W., 2020, Research Progress on chemical constituents and species differences of cannabis, Zhongguo Zhongyao Zazhi (Chinese Journal of traditional Chinese Medicine), 45(15): 3556-3564. (杨柳秀, 李超然, 高雯, 2020, 大麻化学成分及其种属差异研究进展, 中国中药杂志, 45(15): 3556-3564.)
[24]Yang R.X., Liu X.R., Lan B.L., Wang H., Liu X., and Xu J.C., 2021, Genome identification and analysis of the expansin genes familyin Salix purpurea, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 19(8): 2538-2549. (杨锐霞, 刘新茹, 蓝宝良, 王浩, 刘晓, 徐吉臣, 2021, 红皮柳扩展蛋白基因家族的全基因组鉴定与分析, 分子植物育种, 19(8): 2538-2549.)
[25]Yu Z.M., Kang B., He X.W., Lv S.L., Bai Y.H., Ding W.N., Chen M., Cho H.T., and Wu P., 2011, Root hair-specific expansin modulate root hair elongation in rice, Plant J., 66(5): 725-734.
[26]Zhang Q.Y., Lei Z.P., Song Y., Jiang H.B., and He D.H., 2019, Identification and characterization of the expansin gene family in upland cotton (Gossypium hirsutum), Zhongguo Nongye Kexue (Scientia Agricultura Sinica), 52(21): 3713-3732. (张奇艳, 雷忠萍, 宋银, 海江波, 贺道华, 2019, 陆地棉扩展蛋白基因的鉴定与特征分析, 中国农业科学, 52(21): 3713-3732.)
[27]Zhang W., Yan H.W., Chen W.J., Liu J.Y., Jiang C.P., Jiang H.Y., Zhu S.W., and Cheng B.J., 2014, Genome-wide identifica-tion and characterization of maize expansin genes expressed in endosperm, Mol. Gene. Genomics, 289(6): 1061-1074.
[28]Zimmermann R., Sakai H., and Hochholdinger F., 2010, The gibberellic acid Stimulated-Like gene family in maize and its role in lateral root development, Plant Physiol., 152(1): 356-365.
文章摘自:梁子晓,祁宏英,徐洪国.大麻扩展蛋白基因家族的鉴定与生物信息学分析[J/OL].分子植物育种