摘 要:亚麻(Linum usitatissimum L.)是一种重要的经济作物,亚麻相关产品的研发涉及纺织、医药、建筑和材料等领域,具有广阔的应用前景。目前中国高品质的亚麻纤维原料严重不足,生产加工中需求的原麻大部分依赖进口。亚麻的纤维品质与其韧皮部纤维素和半纤维素的合成相关。文章在分子水平上总结了纤维素和半纤维素合成的关键酶促机理,并详细介绍了纤维素合酶(CesA)和类纤维素合酶(Csl)的结构和功能,旨在为改良纤维品质,培育高品质纤维亚麻品种提供理论参考。
关键词:亚麻;纤维素;纤维素合成酶基因;类纤维素合成酶基因
亚麻(Linum usitatissimum L.)为一年生草本植物,可分为纤维用、油用和油纤兼用3种类型,是在北温带地区分布很广的重要经济作物,具有5000多年的种植历史。早在3万年前,人类已经开始利用亚麻纤维。亚麻纤维的主要成分纤维素纤维是一类亲水性纤维,吸湿散湿能力比棉高18%,具有强度高、吸湿性强、散热快、耐摩擦、耐高温、不易燃、不易裂、导电性小、吸尘率低、抑菌保健等优点[1]。全球亚麻产业发展迅速,其中亚麻纺织品特性优良,深受消费者的喜爱。中国目前是世界上最大的亚麻纺织品制造基地[2]。然而,中国的高品质亚麻纤维原料严重短缺,每年进口亚麻原麻达15万~20万锭,占原料需求量的60%~70%[1]。因此改良亚麻纤维品质,培育高品质的纤维亚麻品种,对中国的亚麻产业经济具有重要意义。
亚麻属于韧皮纤维作物。成熟的亚麻韧皮纤维主要由70%~75%的纤维素、15%的半纤维素、10%~15%的果胶以及1.5%~4.2%的木质素组成[3,4,5,6]。亚麻纤维品质取决于纤维素和半纤维素的形成。因此,研究纤维素和半纤维素在分子水平上的合成机理可以为改良亚麻纤维品质提供理论支撑。
纤维素是植物细胞壁的重要组成成分,其是由β-1,4-糖苷键单链相连的吡哺式D-葡萄糖组成的具有一定立体结构的生物大分子多糖。半纤维素是由半乳糖、甘露糖等单糖构成的异质多聚体,化学结构复杂,是植物细胞壁中的一种重要组成成分。纤维素和半纤维素的合成是由许多酶控制的复杂生理过程,其中纤维素合成酶和类纤维素合成酶分别参与纤维素和半纤维的合成[7,8,9],是纤维素与半纤维素生物合成过程中的关键酶,并决定植物纤维素的品质。因此,研究纤维素合成酶基因和类纤维素合成酶基因在纤维发育过程中的作用机制,有利于在分子水平上改良纤维品质。
本文介绍了纤维素合成酶基因和类纤维素合成酶基因的发现与基因结构,汇总了纤维素合成酶基因和类纤维素合成酶基因在亚麻和模式作物拟南芥与水稻中的家族成员,并总结了其在亚麻和模式作物拟南芥与水稻中的研究进展,旨在为改良纤维品质、培育高品质纤维亚麻品种提供理论参考。
1纤维素合成酶(CesA)基因与类纤维素合成酶(Csl)基因
纤维素的合成由多种基因共同作用。纤维素合成酶(CesA)基因和类纤维素合成酶(Csl)基因共同参与纤维素的合成,是决定纤维素品质的关键基因。纤维素合成酶基因与类纤维素合成酶基因共同构成了纤维素合成酶超基因家族。
1.1纤维素合成酶(CesA)基因
纤维素合成酶基因最先在革兰氏阴性菌木醋杆菌(Acetobacter xylinum)中发现[10,11,12]。1982年Aioni等[13]利用细菌中克隆的纤维素合成酶基因在体外合成纤维素,紧接着纤维素合成酶相关基因相继从醋酸杆菌中被分离。细菌中的纤维素合成酶(CesA)是一种糖基转移酶(GT),其氨基酸序列存在一些保守序列,区别于细菌中的其他糖基转移酶。纤维素合成酶(CesA)典型的保守序列为DDDQxxRW,作为标志与其他糖基转移酶的氨基酸序列区分。1996年,通过氨基酸序列的同源性比较,Pear等[14]利用细菌纤维素合成酶基因的保守序列,在棉花(Gossypium hirsutum)中找到了纤维素合成酶基因。此后,一系列的植物纤维素合酶基因相继被鉴定出来,包括拟南芥(Arabidopsis thaliana)[15]、水稻(Oryzasativa L.)[15]、玉米(Zea mays L.)[16]和毛果杨(Populus trichocarpa)[17]等植物。拟南芥基因组全序列测序完成,使得纤维素合成酶的研究得以进一步地深入,随后水稻、玉米、杨树等植物纤维素合成酶基因相继成功被克隆。
植物纤维素合成酶基因长度约3.5~5.5kb,含有9~13个内含子,其外显子与内含子的排列保守,内含子的数量决定基因结构的差异[18]。植物纤维素合成酶基因的转录产物约为3.0~3.5kb,编码的肽链长为985~1088个氨基酸[19],其编码产物序列相似性为53%~98%[18]。不同植物的纤维素合成酶基因具体作用不同,多个纤维素合成酶基因共同参与植物纤维素的合成。植物的纤维素合成酶有共同的结构特征:(1)两端含有8个跨膜结构域,其中6个位于C末端,2个位于N末端,跨膜区是植物体β-1,4-葡萄糖苷链穿过质膜进入细胞壁的重要通道[20];(2)N末端含有锌指结构域或LIM功能结构域,该结构域中的保守重复序列CXXC(半胱氨酸-X-X-半胱氨酸)与各亚基互作维持纤维素合酶复合体的稳定性[20];(3)含有2个高变区,分别位于纤维素合成酶的N端和A区与B区之间,植物纤维素合成酶基因家族的存在与这2个高变区相关[21]。A区与B区之间具有典型的DDDQxxRW保守序列,与底物结合和催化相关[10,22]。
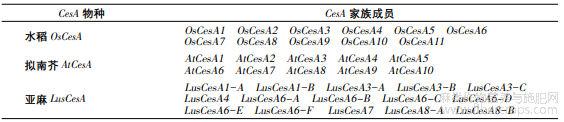
1.2纤维素合成酶(CesA)基因家族成员
不同植物种间的纤维素合成酶基因数目存在差异。目前,在单子叶模式植物水稻中共发现了11个纤维素合成酶基因,在双子叶模式植物拟南芥中共发现了10个纤维素合成酶基因[15]。利用拟南芥的纤维素合成酶基因检索,在亚麻中共找到15个纤维素合成酶基因[23](见表1)。
表1水稻、拟南芥和亚麻中的CesA成员
1.3类纤维素合成酶(Csl)基因
Richmond等[24]在拟南芥基因组中检索到41个与AtCesA1基因和GhCesA基因高度相关的基因。这41个基因中共鉴定出10个AtCesA基因,其余31个基因的功能尚不明确。因其在结构上与纤维素合成酶基因相似,这31个基因被命名为类纤维素合成酶(Csl)基因。类纤维素合成酶基因的长度为2.1~6.8kb,外显子数量为2~14。外显子的数量在不同组间有较大变化。Kimura等[25]通过免疫定位发现类纤维素合成酶基因位于高尔基体体腔,类纤维素合成酶作为一种重要的膜蛋白介导细胞内半纤维素的生物合成。类纤维素合成酶基因与纤维素合成酶基因结构相近,两者编码的氨基酸序列同源性在7%~35%。除不含植物纤维素合成酶所特有的锌指结构域,纤维素合成酶和类纤维素合成酶结构之间具有以下共同特征:(1)含有DDDQxxRW等保守特征,具有完整膜蛋白的结构特征;(2)在N端含有1~2个跨膜结构域;(3)在C端含有3~6个跨膜结构域。这些共同特征用于指示纤维素合成过程中的糖基转移酶[26]。
1.4类纤维素合成酶(Csl)基因家族成员
类纤维素合成酶(Csl)基因家族共含有9个不同家族,单子叶植物和双子叶植物的类纤维素合成酶基因家族间存在差异。水稻中含有CslA,CslB,CslC,CslD,CslE和CslG共6个类纤维素合成酶家族。拟南芥和亚麻中也存在6个类纤维素合成酶家族,分别为CslA,CslB,CslC,CslD,CslE和CslG。CslF和CslH是单子叶特有的类纤维素合成酶基因家族,CslB和CslG是双子叶植物特有的类纤维素合成酶基因家族(见表2)。
表2水稻、拟南芥和亚麻中的Csl家族成员
|
|
Csl家族 |
Csl成员 |
|
|
CslA,CslC,CslD,CslE,CslF,CslH |
9,6,5,3,8,3 |
|
|
CslA,CslB,CslC,CslD,CslE,CslG |
9,6,5,6,1,3 |
|
|
CslA,CslB,CslC,CslD,CslE,CslG |
2,1,8,15,1,4 |
2纤维素合成酶基因与类纤维素合成酶基因的功能
多糖是植物细胞壁的重要组成成分,不同植物细胞壁的多糖成分相似,但单子叶植物细胞壁与双子叶植物细胞壁中含有的半纤维素含量差异显著。纤维素合成酶和类纤维素合成酶是控制细胞壁合成的关键酶,因此纤维素合成酶基因与类纤维素合成酶基因的作用机制有待进一步挖掘。目前,在单子叶模式植物水稻和双子叶模式植物拟南芥中纤维素合成酶和类纤维素合成酶的作用机制研究比较成熟。亚麻具有独特的韧皮发育机制,是研究细胞生长和分化以及细胞壁建成的韧皮纤维模式作物。同时,纤维亚麻作为一种重要经济作物,对亚麻纤维素合成酶和类纤维素合成酶功能的研究,同样具有较大的生产价值和经济价值。
2.1纤维素合成酶CesA基因的功能
纤维素合成酶参与植物细胞壁的合成,是调控纤维素合成的关键酶。Doblin等[21]通过免疫标记发现,有不少于3种纤维素合成酶(CesA)亚型(α1,α2,β)参与细胞壁纤维素的合成。前人研究[27,28]表明,单个纤维素合成酶(CesA)亚基负责合成一条葡聚糖链,不同的纤维素合成酶(CesA)亚基彼此相互作用以形成更高级的复合物,分别在细胞初生壁和次生壁合成时期起不同的作用[29,30],共同参与纤维素的合成。这表明纤维素合成酶是通过聚合形成复合物,从而在植物组织内行使功能。
在水稻的11个纤维素合成酶基因中(表1),初生壁纤维素的合成主要由OsCesA1、OsCesA3和OsCesA8负责,OsCesA5和OsCesA6可能替代OsCesA3的功能。此外,OsCesA3、OsCesA5和OsCesA6的序列高度相似,但关于他们的功能仍需进一步证明。水稻的次生壁生物合成期,OsCesA4、OsCesA7和OsCesA9在纤维素的生物合成过程中起主要作用,是调控水稻次生细胞壁合成的必需酶。Tanaka等[29]在筛选水稻脆秆突变体的过程中发现,OsCesA4、OsCesA7和OsCesA9基因各自突变会导致纤维素含量降低,且茎秆强度显著下降,其纯合突变株系的纤维素含量降低8.9%~25.5%。这表明OsCesA4、OsCesA7和OsCesA9这3个基因之间不存在相互冗余的现象。目前,关于OsCesA2、OsCesA10和OsCesA11基因的功能尚不明确,有待于进一步研究。纤维素是影响植物机械强度的重要因子,水稻CesA基因的深入研究对提高水稻抗性、选育高品质水稻品种具有重大意义。
在双子叶模式植物拟南芥中鉴定的共10个纤维素合成酶基因中(表1),AtCesA1、AtCesA3、AtCesA6、AtCesA2、AtCesA5和AtCesA9基因与拟南芥初生壁合成相关[31,32,33],其中AtCesA1、AtCesA3和AtCesA6对于拟南芥初生细胞壁合成是必需的。Arioli等[34]发现AtCesA1基因突变会影响其纤维素的合成,且突变体植株对温度十分敏感,气温升高会导致其根系肿大并阻碍纤维丝结晶结构的形成。Desprez等[35]发现拟南芥AtCesA6基因的突变体与AtCesA1基因突变体存在同样的表型。由此可见,AtCesA1和AtCesA6两个基因之间存在联系。研究[33]发现,AtCesA1和AtCesA3基因的纯和突变会导致胚胎死亡,且其突变体植株的初生壁严重缺陷。这些研究表明,AtCesA1、AtCesA3和AtCesA63个亚基相互作用,聚合形成复合体,共同调控拟南芥初生壁合成。应用免疫共沉淀的方法,Taylor等[27,36]鉴定出AtCesA4、AtCesA7和AtCesA8共3个拟南芥次生壁合成相关的纤维素合成酶基因。AtCesA4、AtCesA7和AtCesA8基因突变阻碍次生壁纤维素合成,从而引起木质部塌陷,这表明该3个基因在木质部次生壁合成时期起重要作用。在进一步的研究中发现,这3个基因在纤维素合成过程中起协同作用,形成CSC复合体合成纤维素,从而控制木质部纤维的形成[19,27]。关于AtCesA基因作用机制和调控机理的研究,对双子叶植物CesA基因的研究具有重要指导和借鉴作用。
亚麻是韧皮纤维作物,其韧皮纤维细胞的发育主要经历2个阶段:细胞伸长和次生细胞壁加厚,这两个发育时期不重叠,分别决定了韧皮纤维的长度和强度。韧皮纤维发育受很多基因表达调控影响,其中纤维素合成酶是控制纤维合成的关键酶,因此纤维素合成酶基因对于提高亚麻纤维品质具有重要意义[37]。Natalia等[38,39]通过对比拟南芥和杨树中的纤维素合成酶基因,在亚麻中鉴定出的16个纤维素合成酶基因被划分为6个枝,分别命名为LusCesA1-A、LusCesA1-B、LusCesA3-A、LusCesA3-B、LusCesA3-C、LusCesA4、LusCesA6-A、LusCesA6-B、LusCesA6-C、LusCesA6-D、LusCesA6-E、LusCesA6-F、LusCesA7-A、LusCesA7-B、LusCesA8-A和LusCesA8-B。Chantreau等[40]发现LusCesA7B基因合成的蛋白缺乏锌指结构域,应属于类纤维素合成酶基因。Pydiura等[23]也指出亚麻基因组中只含有一个CesA7基因。这些研究表明,亚麻中只存在15个纤维素合成酶基因。这15个LusCesA被分为6大类:LusCesA1、LusCesA3、LusCesA4、LusCesA6、LusCesA7和LusCesA8[23]。这6类纤维素合成酶是控制亚麻初生壁和次生壁合成的关键酶,参与调控亚麻的生长发育,决定亚麻纤维的品质。LusCesA1、LusCesA3和LusCesA6与亚麻的初生细胞壁合成相关[41],且在花、叶和根中LusCesA1和LusCesA6的表达水平相同。这表明LusCesA1、LusCesA3和LusCesA6这3个基因互相作用形成复合体,从而控制亚麻初生壁的合成。Guo等[41]在开花后的亚麻韧皮部检测到富集的LusCesA4和LusCesA8基因,与拟南芥中的AtCesA44和AtCesA8基因的表达模式相一致。AtCesA44和AtCesA8基因是参与拟南芥次生壁合成的关键基因,这表明LusCesA4和LusCesA8基因与次生细胞壁中的纤维素合成相关。江海霞等[42]通过qRT-PCR方法研究LusCesAs基因在亚麻快速生长期的表达模式,发现LusCesA4、LusCesA7和LusCesA8在亚麻韧皮纤维细胞的表达模式与这一结论相一致。Galinovski等[43]研究也表明,LusCesA4、LusCesA7和LusCesA8在次生细胞壁中大量表达,控制次生壁的合成,其中LusCesA7在茎中的特异性表达会影响亚麻纤维素的品质。此外,Hansol等[44]研究发现乙烯可以介导发育早期亚麻的LusCesAs基因的上调或下调表达,这为早期控制亚麻植株的生长发育提供了有力的理论依据。目前,关于亚麻CesA基因的具体功能和调控机制尚有待进一步的阐明。亚麻CesA基因的研究空白,对培育高品质纤维亚麻品种造成不利影响。
2.2类纤维素合成酶Csl基因的功能
类纤维素合成酶(Csl)蛋白存在于高尔基体腔内,介导半纤维素的合成,是影响纤维素品质的关键酶之一。依据序列结构特征的不同,Csl蛋白被分为9个亚族,分别为CslA—CslH及CslJ,其中CslF、CslH和CslJ只存在于单子叶植物中[45]。CslA催化(1,4)-b-D-甘露聚糖的合成,CslC参与催化木葡聚糖骨架的形成,CslD则在多糖(木聚糖和半乳醛聚糖)的合成中起作用。但目前,CslB、CslE和CslG的生物学功能尚不清楚。
Wang等[46]在水稻中共鉴定出了34个类纤维素合成酶基因。水稻的类纤维素合成酶基因被分为6个家族,分别为CslA、CslC、CslD、CslE、CslF和CslH。其中CslA有9个成员,CslC有6个成员,CslD有5个成员,CslE有3个成员,CslF有8个成员,CslH有3个成员。水稻的类纤维素合成酶基因家族成员之间通常在同一组织中表现出高表达水平,存在功能冗余现象。水稻类纤维素合成酶Csl(A1,C9,D2,E1,F6和H1)基因的表达贯穿水稻的整个生命周期[46]。部分水稻类纤维素合成酶表达具有组织特异性,OsCslD3和OsCslD5,OsCslH2和OsCslC9表现出较高的雄激素特异性表达,而OsCslA5、OsCslD1和OsCslD4分别在胚乳、胚根和胚芽中具有特异性。OsCslF6基因突变体植株矮小,分蘖数减少,且突变体植株的纤维素含量降低20%[47]。由此可见,OsCslF6基因与OsCesA基因之间存在功能冗余现象。
拟南芥中共鉴定出了30个类纤维素合成酶基因。Pydiura等[23]通过与棉花、杨树、玉米和水稻中的类纤维素合成酶构建系统发育树,将拟南芥中的类纤维素合成酶基因分为6个家族,分别为CslA、CslB、CslC、CslD、CslE和CslG,其中CslA有9个成员,CslB6个成员,CslC5个成员,CslD6个成员,CslE1个成员,CslG3个成员。前人研究[25]表明,类纤维素合成酶与细胞壁的合成相关。AtCslB5、AtCslD和AtCslD3在根中表达最强,其中AtCslD3的突变会导致根毛细胞壁形成缺陷。拟南芥AtCslA9突变体增强了对根癌农杆菌的抗性,这证明AtCslA9调控细胞壁的合成,从而降低了农杆菌对植株的侵染。拟南芥类纤维素合成酶基因AtCslD1和AtCslD4在成熟的花粉粒和花粉管中特异表达,他们的缺失突变体csld1和csld4都会出现雄性不育的特征[48]。AtCslA10和AtCslG2在花中表达水平升高,可能在开花过程中具有与烟草花粉中特异性表达的NaCslD1和NaGsl1类似的特异功能[49]。AtCsl基因与拟南芥的生长发育密切相关,深入研究AtCsl基因的作用机理,对双子叶植物的抗性研究和品种改良具有重要指导意义。
袁红梅等[50]通过与拟南芥中的类纤维素合成酶蛋白进行氨基酸序列对比,鉴定出30个亚麻的类纤维素合成酶基因,并将这30个亚麻类纤维素合成酶分为6个家族,分别为CslA、CslB、CslC、CslD、CslE和CslG,其中CslA有2个成员,CslB有1个成员,CslC有8个成员,CslD有15个成员,CslE有1个成员,CslG有4个成员。LusCslG3被检测到在叶片中大量表达。LusCslB、LusCslC及LusCslD中的部分成员在不同发育时期表达丰度都极低,表达水平与纤维素、半纤维素在次生木质部中所占组分相符合。目前,关于亚麻类纤维素合成酶(Csl)蛋白的生物学功能尚不清楚,有待于深入研究。
3展望
亚麻作为一种重要的经济作物,在纺织、医疗、建筑和材料等行业具有广泛的应用前景。我国亚麻高品质纤维原料严重缺乏,改良亚麻纤维品质,培育出优良纤维亚麻品种一直是研究的重点。分子克隆和转基因方法改良纤维品质是培育高品质纤维亚麻品种的重要手段。亚麻的分子生物学研究起步较晚,但是发展迅速。目前,分子标记、基因克隆与转化、遗传多样性分析等技术已经在亚麻上取得了进展,且在一定程度上促进了亚麻相关产业的发展。但是相对于棉花等经济作物在转基因方面的研究,亚麻的研究水平还较为落后。CRISPR/Cas基因编辑等技术的问世,使我们可以更便捷地在转基因水平上改良亚麻纤维品质,培育出高品质纤维亚麻品种,进而改善我国高品质纤维亚麻不足的现状,对促进我国亚麻产业经济的发展具有重要意义。
参考文献
[1]王玉富,邱财生,龙松华,等.中国纤维亚麻生产现状与研究进展及建议[J].中国麻业科学,2013,35(4):214-218.
[2]何伟坚,吴霭弟.亚麻纤维的特性及其应用[J].化纤与纺织技术,2019,48(4):40-42+47+52.
[3]Gorshkova T A,Ageeva M V,Chemikosova S B,et al.Tissue-specific processes during cell-wall formation in flax fiber[J].Plant Biosystems,2005,139(1):88-92.
[4]Mooney C,Stone-Smits T,Schols H,et al.Analysis of retted and non retted flax fibres by chemical and enzymatic means[J].Journal of Biotechnology,2001,89(2-3):205-216.
[5]Day A,Addi M,Kim W,et al.ESTs from the fibre-bearing stem tissues of flax(Linum usitatissimum L.):expression analyses of sequences related to cell wall development[J].Plant Biology,2005,7(1):23-32.
[6]John S,Nilmini P,Amandeep S.Virk,et al.A review of bast fibres and their composites: Part1-Fibres as reinforcements[J].Composites: Part A,2010,41(10):1329-1335.
[7]Joshi C P,Mansfield S D.The cellulose paradox--simple molecule,complex biosynthesis[J].Current Opinion in Plant Biology,2007,10(3):220-226.
[8]魏建华,宋艳茹.植物纤维素合酶基因研究进展[J].植物学报,2002,19(6):641-649.
[9]刘长斌,薛永常,聂会忠.高等植物纤维素合酶超家族[J].生命的化学,2007,027(6):533-535.
[10]Saxena I.Cloning and sequencing of the cellulose synthase catalytic subunit gene of Acetobacter xylinum[J].Plant Molecular Biology, 1990,15(5):673-683.
[11]Wong H C,Fear A L,Calhoon R D,et al.Genetic organization of the cellulose synthase operon in Acetobacter xylinum[J].Proceedings of the National Academy of Sciences of the United States of America,1990,87(20):8130-8134.
[12]Saxena I M,Lin F C,Brown R M,et al.Identification of a new gene in an operon for cellulose biosynthesis in Acetobaeter xylinum[J].Plant molecular biology,1991,16(6):947-954.
[13]Aioni K,Delmer D P,Betmman M.Achievement of high rates of in vitro synthesis of 1,4-b glucan: activation by cooperative interaction of the Acetobacter xylinum enzyme system with GTP,polyethylene glycol,and a protein factor[J].Proceedings of the National Academy of Sciences,1982,79:6448-6452.
[14]Pear J R,Kaeagoe Y,Schreckengost W E,et al. Higher plants contain homologs of the bacterial celA genes encoding the catalytic subunit of cellulose synthase[J]. Proceedings of the National Academy of Sciences,1996,93( 22) : 12637-12642.
[15]Richmond T A,Somerville C R.The cellulose synthase superfamily[J].Plant physiology,2000,124(2):495-498.
[16]Appenzeller L,Doblin M,Barreiro R,et al.Cellulose synthesis in maize isolation and expression analysis of the cellulose synthase (CesA)gene family[J].Cellulose,2004,11(3-4):287-299.
[17]Joshi C P,Bhandari S,Ranjan P,et al.Genomics of cellulose biosynthesis in poplars[J].New Phytologist,2004,164(1):53-61.
[18]邰付菊,李学宝.植物纤维素生物合成及其相关酶类[J].中国细胞生物学学报,2004,026(5):490-494.
[19]Richmond T.Higher plant cellulose synthases[J].Genome Biology,2000,1(4):1-5.
[20]Kurek I,Kawagoe Y,Jacob-Wilk D.Dimerization of cotton fiber cellulose synthase catalytic subunits occurs via oxidation of the zinc-binding domains[J].Proceedings of the National Academy of Sciences,2002,99(17):11109-11114.
[21]Doblin M S,Kurek I,Jacobwilk D,et al.Cellulose Biosynthesis in Plants: from Genes to Rosettes[J].Plant and Cell Physiology,2002,43(12):1407-1420.
[22]Saxena I M.Brown R M.Identification of cellulose synthase( s) in higher plants sequence analysis of processive β-glycosyltransferases with the common motif‘D,D,D35Q(R,Q)XRW’[J].Cellulose,1997,4(1):33-49.
[23]Pydiura N A,Bayer G Y,Galinousky D V,et al.Bioinformatic search for cellulose synthase genes in flax(Linum usitatissimum)and their phylogenetic analysis[J].Cytology and Genetics,2015,49(5):279-287.
[24]Richmond T A,Somerville R C. The Cellulose Synthase Superfamily[J].PlantPhysiology,2000,124(2):495-498.
[25]Kimura S,Laosinchai W,Itoh T,et al.Immunogold labeling of rosette terminal cellulose synthesizing complexes in the vascular plant Vigna angularis[J].Plant Cell,1999,11(11):2075-2085.第3期肖青梅等:亚麻纤维合成关键酶CesA基因和Csl基因的研究进展153
[26]Saxena I M,Jr R M B,Fevre M,et al. Multidomain architecture of beta-glycosyl transferases: implications for mechanism of action [J]. Journal of Bacteriology,1995,177( 6) : 1419-1424.
[27]Taylor N G,Howells R M,Huttly A K,et al.Interactions among three distinct CesA proteins essential for cellulose synthesis[J].Proceedings of the National Academy of Sciences of the United States ofAmerica,2003,100(3):1450.
[28]Atanassov I I,Pittman J K,Turner S R.Elucidating the mechanisms of assembly and subunit interaction of the cellulose synthase complex of Arabidopsis secondary cell walls[J].Journal of Biological Chemistry,2009,284(6):3833-3841.
[29]Tanaka K,Murata K,Yamazaki M,et a1.Three Distinct Rice Cellulose Synthase Catalytic Subunit Genes Required for Cellulose Synthesis in the Secondary Wall[J].Plant Physiology,2003,133(1):73-83.
[30]Ranik M,Myburg A A.Six new cellulose synthase genes from Eucalyptus are associated with primary and secondary cell wall biosynthesis[J].Tree Physiology,2006,26(5):545.
[31]王振怡,王金朋,潘玉欣,等.拟南芥和水稻CesA基因家族的生物信息学分析[J].河南农业科学,2015(6):13-17.
[32]Persson S,Paredez A,Carroll A,et al.Genetic evidence for three unique components in primary cell-wall cellulose synthase complexes in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(39):15566-15571.
[33]Desprez T,Juraniec M,Crowell E F,et al. Organization of cellulose synthase complexes involved in primary cell wall synthesis in Arabidopsis thaliana[J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(39):15572-15577.
[34]Arioli T,Peng L,Betzner A S,et al.Molecular analysis of cellulose biosynthesis in Arabidopsis[J].Science,1998,279(5351):717-720.
[35]Desprez T,Vernhettes S,Fagard M,et al.Resistance against herbicide isoxaben and cellulose deficiency caused by distinct mutations in same cellulose synthase isoform CESA 6[J].Plant Physiology,2002,128(2):482-490.
[36]Taylor N G,Laurie S,Turner S R.Multiple cellulose synthase catalytic subunits are required for cellulose synthesis in Arabidopsis[J].The Plant cell,2000,12(12):2529-2540.
[37]刘岩.亚麻AtCesA1基因过表达载体的构建[J].中国麻业科学,2017,39(2):57-60.
[38]Natalia M,Tatyana G,Deyholos M K,et al.Chitinase-like(CTL)and cellulose synthase(CESA)gene expression in gelatinous-type cellulosic walls of flax (Linum usitatissimum L.)Bast Fibers[J].Plos One,2014,9(6):e97949.
[39]Wang Z W,Hobson N,Galindo L,et al.The genome of flax(Linum usitatissimum)assembled de novo from short shotgun sequence reads[J].Plant Journal,2012,72:461-473.
[40]Chantreau M,Chabbert B,Billiard S,et al. Functional analyses of cellulose synthase genes in flax(Linum usitatissimum)by virus- induced gene silencing[J].Plant Biotechnology Journal,2015,13(9):1312-1324.
[41]Guo Y,Qiu C,Long S,et al.Digital gene expression profiling of flax(Linum usitatissimum L.)stem peel identifies genes enriched in fiber-bearing phloem tissue[J].Gene,2017,626:32-40.
[42]江海霞,郭栋良,李玉环,等.亚麻快速生长期细胞壁形成相关基因的表达分析[J].中国农业科学,2017,050(13):2442- 2450.
[43]Galinovskiǐ D V,Anisimova N V,Raǐskirǐ A P,et al.Cellulose synthase genes that control the fiber formation of flax(Linum usitatissimum L.)[J].Genetika,2014,50(1):26-34.
[44]Hansol L,Seung-Ho P,Seung-Eun O.Effect of 1-aminocyclopropane-1-carboxylic acid(ACC)-induced ethylene on cellulose synthase A(CesA) genes in flax(Linum usitatissimum L.‘Nike’)seedlings[J].Genes&Genomics,2018,40:1237-1248.
[45]Hamann T,Osborne E,Youngs H L,et al.Global expression analysis of CESA and CSL genes in Arabidopsis[J].Cellulose,2004,11(3):279-286.
[46]Wang L,Guo K,Li Y,et al. Expression profiling and integrative analysis of the CESA/CSL superfamily in rice[J].BMC Plant Biology,2010,10(1):282.
[47]卓经迪.水稻细胞壁降解过程中的多糖变化及OsCslF6 基因的遗传转化研究[D].武汉:华中农业大学,2017.
[48]王伟,王丽,周奕华,等.拟南芥似纤维素合成酶CSLD1和CSLD4对花粉管生长的作用[A].中国植物学会细胞生物学专业委员会.中国植物学会植物细胞生物学2010年学术年会论文摘要汇编[C].中国植物学会细胞生物学专业委员会:中国植物学会,2010:1.
[49]DoblinM S,De-Melis L,Bacic A,et al.Pollen tubes of Nicotiana alata express two genes from different beta-glucan synthase families [J].Plant Physiology,2001,125:2040-2052.
[50]袁红梅,郭文栋,赵丽娟,等.亚麻纤维素合酶超基因家族的生物信息学及表达分析[J].中国农业科学,2016,49(23):4656-4668.
文章摘自:肖青梅,郭媛.亚麻纤维合成关键酶CesA基因和Csl基因的研究进展[J].中国麻业科学,2021,43(03):148-154.